Innledning
Sukkerrør (Saccharum spp., Poaceae) er en årlig eller flerårig avling dyrket i tropiske og subtropiske regioner over hele verden. Sukkerrør er en viktig avling for sukroseproduksjon, og står for 70% av verdens sukkerproduksjon. I tillegg, som et C4-anlegg, kan sukkerrør effektivt omdanne solenergi til kjemisk energi, og er derfor en ideell avling for biodrivstoff for etanolproduksjon (Lam et al., 2009; Santchurn et al., 2014). Slekten Saccharum har seks arter: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense og S. edule, hvorav S. spontaneum regnes som den ville arten med et grunnleggende kromosomsett av x = 8. S. spontaneum er også en av de to viktigste Saccharum-artene som bidrar til den moderne sukkerrørskulturen (Dhont et al., 1998; Ha et al., 1999). S. spontaneum har en sterk miljøtilpasningsevne og inneholder viktige genetiske egenskaper for sykdoms- og tørkebestandighet, og bidrar dermed til stresstoleransen til moderne kultivhybrider (Grivet et al., 2004). S. spontaneum har den bredeste geografiske fordelingen, og dens ploidnivåer varierer fra 2n = 5x = 40 til 2n = 16x = 128 (Panje og Babu, 1960).
Sukkerrankromosomer har lignende morfologier og har en liten størrelse på 1–6 μm på det kondenserte metafasetrinnet (Ha et al., 1999; Dhont, 2005); derfor er det veldig utfordrende å skille mellom de forskjellige kromosomene basert på tradisjonelle cytogenetiske metoder. Dessuten, som en autopolyploid, er de kromosomale strukturelle endringene forårsaket av polyploidisering, duplisering, sletting og rekombinasjon veldig vanlig (Piperidis et al., 2010). I S. spontaneum er det utviklet genetiske koblingskart (Silva et al., 1995), men utvikling av et cytologisk genetisk kart henger etter andre gressarter. Pålitelige cytologiske verktøy for å identifisere individuelle kromosomer kan brukes til effektiv genomforskning og utnyttelse av kimplasmeressurs, spesielt med den komplekse genombakgrunnen til moderne sukkerrørsorter. Kromosomspesifikke bakterielle kunstige kromosomkloner (BAC) er uvurderlig ressurs for forskning på sukkerrørgenomer, og vil ha mange anvendelser innen fysisk kartlegging, kromosomidentifikasjon og markørassistert avl av Saccharum spp., Og til å bistå sekvensering og montering av sukkerrørgenom .
Fluorescens in situ hybridisering (FISH) er et kraftig verktøy for molekylær cytologi (Jiang og Gill, 2006). I Saccharum spp. Ble 45S rDNA og 5S rDNA brukt som prober for å oppdage de grunnleggende kromosomene til S. spontaneum, S. robustum og S. officinarum, for å identifisere de forskjellige grunnleggende kromosomene, x = 8 i S. spontaneum, og x = 10 i S. robustum og S. officinarum (Dhont et al., 1998; Ha et al., 1999). Basert på FISH ble moderne sukkerrørsorter funnet å inneholde 70–80% av kromosomene avledet fra S. officinarum, og 10–23% av kromosomene fra S. spontaneum, mens 5–17% ser ut til å være produktet av rekombinasjonen mellom S. spontaneum og S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). Videre ble kromosomeliminering, rekombinasjon og translokasjonshendelser påvist i noen stamfedre avledet av hybridiseringen mellom Saccharum og Erianthus arundinaceus ved bruk av FISH (Dhont et al., 1995; Huang et al., 2015).
Sorghum har et lite diploid genom (730 Mbp) med lav frekvens av kromosomrekombineringshendelser. Sorghum deler høy synteny med sukkerrørgenomet, noe som gjør det til et ideelt referanseanlegg for komparative analyser med sukkerrørgenomet (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ). I denne studien, basert på tilgjengelige sekvenser for BAC-ressurser med høy dekning, ble pålitelige BAC-sonder utviklet for FISH-kromosomidentifikasjon og cytogenetisk kartkonstruksjon. Målet med denne studien var å: (1) utvikle et sett med kromosomspesifikke BAC-FISH-prober på S. spontaneum-kromosom og (2) utforske kromosomomdannelsen mellom S. spontaneum og sorghum.
Materialer og metoder
Materialer
Saccharum spontaneum SES208 (2n = 8x = 64) ble brukt til cytologiske analyser i denne studien. SES208-plantene ble dyrket i feltet på campus ved Fujian Agricultural and Forestry University (Fuzhou, Kina) i februar 2015 og opprettholdt under regelmessige vekstforhold for sukkerrør.
Et BAC-bibliotek ble konstruert fra det haploide genomet. av S. spontaneum AP85-441 (4x = 32), som ble avledet fra S. spontaneum SES208 (2n = 8x = 64) via anther in vitro-kultur. Genomstørrelsen på AP85-441 er omtrent 3,2 Gbp. Biblioteket besto av 38 400 kloner, med en gjennomsnittlig innsatsstørrelse på 100 kb, som dekker 6 × av hele genomet.Dette BAC-biblioteket har blitt brukt til å identifisere sukrosetransportører og fruktokinasegenfamilier i S. spontaneum (Zhang et al., 2016; Chen et al., 2017).
Screening av BAC-biblioteket
35 156 BAC-kloner fra AP85-441-bibliotekene ble samlet i 701 biblioteker. Hvert bibliotek inneholder i gjennomsnitt 50 BAC-kloner. DNA-bibliotekene ble utarbeidet med PhasePrepTM BAC DNA Kit (Sigma, USA) i henhold til produsentens protokoller. BAC DNA-biblioteker ble sekvensert ved hjelp av Illumina HiSeq 2500-plattformen med PE250-modellen. Totalt ble 686 biblioteker (18 biblioteker mislyktes for sekvensering) sekvensert, og 267,5 Gb renset data ble generert etter trimming ved bruk av Trimmomatic versjon 0.36 (Bankevich et al., 2012). Data fra hver BAC-pool ble samlet ved hjelp av SPAdes versjon 3.09 med standardparameter. Totalt 2,611,145 contigs ble samlet med contig N50 på 7,38 kbp (Zhang og Ming, upubliserte data).
AP85-441 BAC-biblioteksekvensene ble BLAST søkt mot sorghumgenomet med en E-verdi på 1e -4. Sekvensene som hadde enkelt BLAST-treff i sorghumgenomet ble valgt som kandidat FISH-prober. For å identifisere BAC-kloner med lavt antall kopier fra BAC-biblioteket, ble sekvensspesifikke primere designet med Primer 5.0-programvare for å muliggjøre screening av BAC-bassenger ved PCR. Primerlengder på 18-25 bp, Tm-verdier på 55-65 ° C og nukleotidsammensetninger på 40-60% cytosin og guanin ble valgt. En totrinns PCR-metode ble brukt til å skjerme 3D-dimensjonsbassengene i BAC-biblioteket (Asakawa et al., 1997; Crooijmans et al., 2000) med noen modifikasjoner. Hver klon fra en 384-brønns plate ble dyrket i 80 ul Lysogeny buljong + 34 mg / ml kloramfenikol over natten ved 37 ° C i en 384-brønns kulturplate. Totalt ble åtte 384-brønners plater med totalt 3072 BAC-kloner dyrket. For hver plate med 384 brønner konstruerte vi først 16 rad bassenger (rad A til P) og 24 kolonne bassenger (kolonne 1–24) for hver 384-brønns plate ved å blande like volum kultur for hver enkelt klon, og deretter blandet 16 rad bassenger og 24 kolonne basseng sammen med like store volumer fra hver rad og kolonne basseng som en plate basseng eller superpool. I denne studien fokuserte vi på screening av åtte 384-brønners plater. Dermed ble åtte superpools forberedt for de åtte platene hver for seg. Den positive 384-brønners plate fra de åtte platene ble identifisert, og deretter ble den positive raden blant de 16 blandingene screenet. Til slutt ble de PCR-positive kolonnene valgt blant 24 kolonneblandinger. Tjuefire PCR-reaksjoner ble utført for å identifisere positive kloner fra de åtte 384-brønnsplatene.
PCR-amplifikasjoner ble utført som tidligere beskrevet (Bouzidi et al., 2006). Hver reaksjon inkluderte rTaq forblanding 7,5 ul, 0,6 ul av hver grunning (10 mikrometer), 1,0 ul bakterievæskemal, til et sluttvolum på 20 ul. Etter initial denaturering ved 95 ° C i 5 minutter ble 35 sykluser ved 95 ° C i 30 s, 55 ° C i 30 s og 72 ° C i 1 min utført. PCR-produkter ble analysert på en 1,5% agarosegel.
Metaphase Chromosome Preparation
Kromosompreparat ble utført som tidligere beskrevet (Lou et al., 2010) med mindre modifikasjoner. Kort sagt ble S. spontaneum-planter dyrket under drivhusforholdene for høsting av rotspisser. Skårne rotspisser ca. 1–2 cm ble behandlet med 0,002 mol / l 8-hydroksykinolin ved romtemperatur i 2-4 timer, skyllet i vann i 15 minutter og fiksert i etanol: eddiksyre (3: 1) i minst 24 h ved romtemperatur. Rotspissene ble deretter fordøyd i en enzymløsning (4% cellulose R-10 og 2% pektolyase i 0,1 M citratbuffer) ved 37 ° C i 1 time, vasket med isavionisert vann i 30 minutter og til slutt inkubert i etanol: eddiksyre (3: 1) i ca. 30 minutter. Objekter ble fremstilt i henhold til «flammetørket» -metoden (Iovene et al., 2008).
BAC DNA-rensing og probemerking
BAC-DNA ble ekstrahert med PhasePrepTM BAC DNA-sett i henhold til produsentens håndbok. Renset BAC DNA ble merket ved standard nick-translasjonsreaksjon, inkludert fortynnet DNase I, 10 × nick-translasjonsbuffer, DNA Polymerase I, dNTP, biotin- / digoksigenin-merkede dUTPer, og BAC DNA. Blandingen ble inkubert ved 15 ° C i 1,5 timer. De kuttede produktene ble deretter undersøkt på en 1,5% agarosegel for tilstedeværelse av et smøre mellom 300 og 500 bp. De oppnådde sonder ble lagret ved -20 ° C til de ble brukt.
In situ-hybridisering og påvisning
Probemiks og hybridisering: Først probeblandingen (50% deionisert formamid, 2 × SSC, 80 ng digoksygenin- / biotin-merket DNA, 10% dekstransulfat , > 1 μg C ° t-100) ble plassert i en 90 ° C varm blokk i 5 minutter, og deretter umiddelbart overført på is til den var klar til sonde for hybridizati på. De tidligere tilberedte flammetørkede lysbildene ble behandlet med 70% deionisert formamid, denaturert på en varmeblokk ved 80 ° C i 1.5 min, umiddelbart nedsenket sekvensielt i iskald 70% etanol, 90% etanol, og deretter 100% etanol hver i 5 minutter, og deretter lufttørket på benken. Til slutt ble denaturerte sonder lagt til hvert lysbilde, og dekket med 24 × 32 mm dekkglass. Objektglass ble plassert i et fuktig kammer ved 37 ° C over natten.
Sondedeteksjon: dekkglassene ble fjernet og lysbildene ble vasket ved romtemperatur i 2 x SSC i 5 minutter, ved 42 ° C i 2 x SSC i 10 minutter og ved romtemperatur i 1 × PBS i 5 minutter i denne sekvensen. Biotin-merkede probesignaler ble påvist med 2 mg / ml Alexa Fluor 488 streptavidin og digoksygenin-merkede probesignaler ble påvist med 2% anti-digoksigenin-rodamin fra sau. Antistoffcocktailen (100 ul TNB-buffer, 1 ul Alexa Fluor 488 streptavidin, 1 ul rodamin anti-gravesau) ble tilsatt lysbildene, som ble dekket med 24 × 32 mm dekkglass, inkubert i 1 time ved 37 ° C i et fuktig kammer, og vasket ved romtemperatur i 1 × PBS tre ganger i 5 minutter hver. Overskudd av væske ble fjernet og 4 ′, 6-diamidino-2-fenylindol (DAPI) (Sigma, St. Louis, MO, USA) i antifadeoppløsningen Vectashield (Vector, Burlingame, CA, USA) ble tilsatt for å motbe kromosomer. Bilder ble tatt med et Olympus BX63 epifluorescensmikroskop. FISH-signalbilder ble analysert ved hjelp av CellSens Dimension-programvaren.
Resultater
Screening Low-Copy BAC Clones
Å skjerme BAC-klonene som potensielt har lavt kopiantal sekvenser i S. spontaneum genomet, vi BLAST søkte de sekvenserte AP85-441 BAC bibliotekene mot sorghum genomet. Totalt ble 2000 BAC-sekvenser tilsvarende 10 sorghum-kromosomer med god kollinearitet og viser sekvensspesifisitet screenet. 2000 AP85-441 BAC-sekvensene ble maskert av REPEATMASKER mot den høy-gjentatte sorghum DNA-databasen og TIGR gramineae-databasen for å filtrere gjentatte sekvenser. Til slutt ble 114 BACer fordelt på 10 sorghumkromosomer valgt for FISH-analyse (tilleggstabell 1) med 7–16 BACer på hver av de 10 sorghumkromosomene (figur 1). For å skjerme positive BAC-kloner, ble 114 spesifikke primere designet basert på BAC-klonsekvensene (tilleggstabell 2) og 49 positive BAC-kloner ble screenet fra BAC-bibliotekene (tilleggstabell 3).
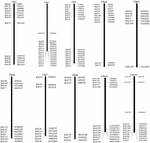
FIGUR 1. Fordelingen av 114 BAC-kloner fra AP85-441 i kromosomene av sorghum. BAC1-114 representerer BAC-nummeret, dataene til høyre er den fysiske plasseringen av BAC i tilsvarende region av sorghum-kromosomene. Enhet = bp.
BAC-FISH Signalstyrke og distribusjon
DNA-prøver isolert fra de 49 positive BAC-kloner ble merket med biotin eller digoksigenin. 5S rDNA- og 45S rDNA-prober ble brukt som kontroll og ble hybridisert til S. spontaneum somatiske metafasekromosomer etter FISH-prosedyren. På grunn av korrelasjonen mellom signalstyrke og variasjonen av gjentatt sekvensinnhold i BAC-sekvensene, brukte vi C ° t-100 som en konkurrent (tabell 1). BAC-kloner som viser sterke og stabile hybridiseringssignaler ble valgt for videre FISH-analyse.

TABELL 1. Klassifiseringen av 27 positive kloner med blokkerende DNA.
Totalt 64 forskjellige kromosomer kan observeres i metafasen til S. spontaneum. Hver BAC-FISH ble utført i fire uavhengige eksperimenter (eller lysbilder). Minst 10 spredning av somatiske metafasekromosomer ble analysert i hvert lysbilde. Resultatene viste at de 27 spesifikke sonderne (tilleggstabell 4) kan klassifiseres i fem grupper basert på signalet om hybridisering (tabell 1). I gruppe I (inkludert BAC-18, BAC-20, BAC-29 og BAC-84), viste sonder åtte særegne steder uten konkurransedyktig in situ-undertrykkelse (CISS) ved bruk av C ° t-100 (figur 2A), noe som tyder på noen gjentatte sekvenser eksisterte i disse BAC-sekvensene. I gruppe II (inkludert BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 og BAC-74), ° C -100 ble brukt som konkurrent for BAC-probe hybridisering, og seks til syv ganske forskjellige steder ble vist i karyotyper av denne gruppen (figur 2B). Hybridiseringsresultatet var lik 45S rDNA med syv signalsteder, som kan være forårsaket av variasjon i kromosomstruktur i det homologe kromosomet. I gruppe III (inkludert BAC-26, BAC-66, BAC-76 og BAC-78) ble C ° t-100 DNA brukt som en konkurrent, og åtte forskjellige steder kunne observeres i karyotypen, noe som tyder på denne gruppen BAC kloner har repeterende DNA-sekvenser (figur 2C). I gruppe IV (inkludert BAC-24) ble mer enn åtte signalsteder observert i karyotypen og dukket opp i de pericentromere og telomere regionene (figur 2D), noe som tyder på at disse BAC-probene hadde repeterende sekvenser i S. spontaneum.I gruppe V (inkludert BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 og BAC-81), viste probene spredte signaler i alle kromosomer med begrensede regioner av individuelle kromosomer som viser spesifikke signaler (figur 2E). Hybridiseringsresultatet indikerte at denne gruppen av BAC-kloner inneholdt store mengder repeterende DNA i S. spontaneum, som kan være forårsaket av en rask evolusjonær divergens for de repeterende regionene etter splittelsen av sorghum og S. spontaneum. Derfor viste et sett med 19 BAC-prober (tilleggstabell 3) fra gruppe I, II og III spesifikke signaler på S. spontaneum-kromosomer som tilsvarer ni av sorghum-kromosomene ved siden av Sb05, av de 49 BAC-probene. Signalene fra de andre 22 sonder ble spredt i alle kromosomer. Vi valgte et sett med 19 BAC-sonder som S. spontaneum kromosomspesifikke signaler (figur 3). Av de 19 BAC-probene var en probe lokalisert på Sb02, Sb04 og Sb09; to sonder på Sb07, Sb08 og Sb10; tre sonder på Sb01 og Sb06; og fire sonder på Sb03. Dette sondesettet ga et nyttig verktøy for komparativ analyse av S. spontaneum og sorghum (tabell 2).
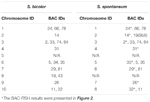
TABELL 2. Fordelingen av BAC-FISH-prober i kromosomer av S. bicolor og S. spontaneum.
Konstruksjon av et BAC-bibliotek spesifikt for S. spontaneum-kromosomer
Sb01 og Sb02 er de to største sorghum-kromosomene. For å oppdage genomets syntese mellom sorghum og S. spontaneum ble 10 kromosomspesifikke BAC-kloner valgt fra gruppe I, II og III. Av disse 10 BAC-probene var tre sonder (BAC-klon-ID: 24, 66, 78) homologe med Sb01, og syv sonder (BAC-klon-ID: 14, 18, 20, 69, 71, 73, 77) tilsvarte Sb02. Vi benyttet dobbeltfargedeteksjon av FISH for komplementært merkede BAC-par og sonder for å kartlegge kjedens cytogenetiske forhold for BAC-klonene. Ved å gjøre dette konstruerte vi det cytogenetiske kartet over S. spontaneum basert på de 10 BAC-probene.
For tre sonder som tilsvarer Sb01, viste FISH-analyse av S. spontaneum (SES208) at BAC-24 og BAC -78 sonder var hovedsakelig lokalisert på den distale regionen av S. spontaneum kromosom 1, og BAC-66 ble kartlagt i nærheten av den sentromere regionen (figur 4). BAC-24 og BAC-66 lokalisert til samme kromosom (S. spontaneum kromosom 1); samtidig mens probe 66 og probe 78 også befinner seg på samme kromosom, og viser at de tre BAC-probene fordelte seg til de samme kromosomene i S. spontaneum, noe som tyder på syntese mellom S. spontaneum og sorghum for kromosom 1. FISK-resultater avslørte at de syv BAC-probene justert til Sb02; bare sonder 69, 71, 73 og 77 av de syv BACene genererte intense signaler på ett S. spontaneum-kromosom (SsChr2) (figur 5); mens sonder 14, 18 og 20 ikke kunne påvises på S. spontaneum-kromosom 2, men ble observert på andre S. spontaneum-kromosomer. Disse resultatene viste at kromosomomdannelsene skjedde i den tilsvarende Sb02 mellom S. spontaneum og sorghum.
Videre delte sekvensen av BAC-2 høy likhet (97%) til et fragment på kromosom 3 av sorghum. Dobbelfarget FISH-analyse viste at både BAC-2 og 45s rDNA ble kartlagt til samme sted som S. spontaneum-kromosomet (Figur 6).
Kromosomomdannelse på Sb02 mellom Sorghum og S. spontaneum
For å videre undersøke kromosomomdannelsen på Sb02 mellom sorghum og S. spontaneum, kromosom -spesifikke prober tilsvarende Sb02 (BAC-14, BAC-18 og BAC-20) og tilsvarende Sb08 (BAC-19 og BAC-43) ble brukt for FISH-analyse. Resultatene viste at BAC-14 og BAC-19 ble kartlagt til forskjellige armer av de samme S. spontaneum-kromosomene, noe som indikerer at kromosomomdannelser fant sted mellom kromosomer 2 og 8 i S. spontaneum og sorghum (Figur 7).
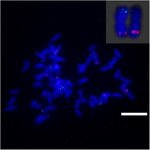
FIGUR 7. Omlegging av kromosom i sukkerrør og sorghum som avslørt av FISH. BAC-14 og BAC-19 sonder merket med biotin (grønn) og digoksigenin (rød). Skalestenger = 10 μm.
Diskusjon
Saccharum er en kompleks slekt preget av høye polyploidinivåer, med små kromosomer (Ha et al., 1999; Dhont, 2005). Å skille individuelle Saccharum-kromosomer ut fra deres morfologi er veldig utfordrende. Ved å utnytte sekvenser som er sterkt dekket av BAC-biblioteket til S. spontaneum, var vi i stand til å skjerme for de potensielle BAC-klonene med lavt antall kopier i S. spontaneum ved å bruke sorghumgenomet som referanse. Som en høy polyploid art kan S. spontaneum inneholde flere genomomlegginger enn diploider.Forskjellene i genomene mellom S. spontaneum og sorghum kan være årsaken til inexactitude i spådommene for de lave kopiantal BAC-sekvensene i S. spontaneum-genomet. I denne studien har vi opprinnelig fått 114 potensielle BAC-sekvenser med enkeltkopi basert på sorghumgenomet. Imidlertid kunne bare 49 identifiseres i de delvise BAC-bibliotekene, og 27 ble funnet å være kromosomspesifikke BAC-FISH-prober. Det er minst to grunner til å forklare disse resultatene. For det første var de 114 BAC-sekvensene delvise fragmenter av BAC-innsettingen på grunn av begrensningene for NGS-sekvensmontering, og kan inneholde gjentatte sekvenser i de andre regionene som ikke har sekvensinformasjon. Disse potensielt avdekkede gjentakelsessekvensene kan villede påvisningen av sekvenser med lav kopi gjennom sekvenssammenligning. For det andre kan den nylige polyploidiseringen i S. spontaneum forårsake variasjon av gjentatte sekvenser mellom S. spontaneum og sorghum, og dermed resultere i det uspesifikke FISH-signalet i S. spontaneum. I en tidligere studie genererte sekvensering av 20 BAC i sukkerrørshybrider 1,45 Mb contig-sekvenser, sekvensene på linje med sorghumgenom spente 0,99 Mb, som utgjorde omtrent halvparten av sukkerrørets BAC-sekvenser (Wang et al., 2010). Åpenbart eksisterte slettinger / innsettinger mellom sorghum og Saccharum, og bruken av sorghumgenomet som referanse kan ikke erstatte de unike egenskapene til Saccharum-genomet, og kan derfor forårsake uforutsagte FISH-resultater.
Noen FISH-lysbilder hadde sterke bakgrunnsstøy, som kan være på grunn av små regioner med gjentatte sekvenser (slik som SSR-sekvenser) i BAC-klonen, som følgelig gir interferens under hybridisering. Sorghumgenomet har et gjentatt innhold på omtrent 61% (Paterson et al., 2009), mens omtrent halvparten av genomet består av gjentatte sekvenser i Saccarrum-hybrider (De et al., 2014). I disse repeterende genomene er det vanskelig å utvikle signal-spesifikke FISH-prober, som skiller kromosomer cytogenetisk med lignende morfologier. CISS med blokkerende DNA C ° t-100 kan effektivt utelukke gjentatte sekvenser. I denne studien ble blokkering av DNA C ° t-100 brukt til FISH-analyse av BAC-sonder i gruppe II og III (figur 2D-F), som ble bekreftet som kromosomspesifikke BAC-sonder, mens FISK av BAC-sonder i gruppe IV og V produserte sterk bakgrunnsinterferens (figur 2D – F). Det er åpenbart at det er variasjoner av gjentatte sekvenser blant de undersøkte BAC-probene. Derfor ville optimalisering av eksperimenter være nødvendig for BAC-FISH-analyse i Saccharum. Seks til syv steder ble vist i gruppe II, noe som kunne være forårsaket av de homologe kromosomstrukturvariasjonene, slik som fragmentets sletting i en eller to homologe kromosomer. I heksaploid hvete hybridiserte BAC 676D4 sterkere til A-genom-kromosomene enn til B- og D-genom-kromosomene (Zhang et al., 2004). Disse som ikke kan oppdages av ett til to signaler i de homologe kromosomene, kan også være forårsaket av de tekniske problemene med BAC-FISH for så mange kromosomer med liten størrelse.
I denne studien identifiserte vi 27 kromosompesifikke BAC- FISH-sonder som tilsvarer 9 av de 10 sorghum-kromosomene (alle unntatt Sb05). I en studie av det genetiske kartet avledet fra et kryss mellom S. officinarum og sukkerrørsorter, ble Sb05 slått sammen med Sb06 i sukkerrør (Aitken et al., 2014). I vår studie avslørte et genetisk kart basert på F1-populasjonen i S. spontaneum at Sb05 ble delt inn i to segmenter, og de to segmentene ble slått sammen med henholdsvis Sb06 og Sb07 (Zhang og Ming, upubliserte data). Nylig viste det genetiske kartet til et medlem av Andropogoneae, Miscanthus sinensis, at Sb05 har dårlig kollinearitet med den tilsvarende koblingsgruppen i M. sinensis (Ma et al., 2012). Tilsvarende beholder de to forfedre maiskromosomene ortologe til Sb05 det minste antall synteniske ortologer til sorghumgener (Schubert og Lysak, 2011). Derfor kan fraværet av BAC-FISH tilsvarende Sb05 indikere kromosomfusjon i S. spontaneum.
Sorghum og S. spontaneum divergerte for 12 millioner år siden (MYA), det grunnleggende kromosontallet ble redusert fra x = 10 til x = 8. I Aitken et al. (2014), HG2 (homolog gruppe 2) av sukkerrør justert til Sb05 og Sb06, og HG8 (homolog gruppe 8) til Sb02 og Sb08, og gir bevis for den grunnleggende kromosomreduksjonshendelsen (Aitken et al., 2014). I denne studien observerte vi at sorghumkromosomene Sb02 og Sb08 hadde interkromosomal omlegging i S. spontaneum som demonstrert av beviset for at BAC-14 justert til Sb02, og BAC-19 justert til Sb08 (figur 7). Studien vår ga det første fysiske og cytogenetiske beviset for sorghum inter-kromosom omlegging i S. spontaneum. Upubliserte data fra vår gruppe avslørte også at Sb08 er delt inn i to segmenter, og ble slått sammen med av Sb02 og Sb09 i henholdsvis S. spontaneum (Zhang og Ming, upubliserte data).Sonder som tilsvarer Sb09, Sb08, Sb05 og Sb06 kan brukes videre til å undersøke de inter-kromosomale hendelsene mellom sorghum og S. spontaneum. S. spontaneum har et bredt spekter av ploidinivåer (2n = 40–128) (Irvine, 1999). Disse BAC-probene kan brukes til å bekrefte inter-kromosomal omlegging av S. spontaneum med de forskjellige polyploidinivåene.
På grunn av forskjellig grunnleggende kromosontall mellom sorghum (x = 10) og S. spontaneum (x = 8) var sorghumkromosomene ikke en en-til-en korrespondanse med S. spontaneum. Som vår genetiske kartleggingsstudie nevnt her tidligere, ble Sb08 delt inn i to segmenter som fusjonerte med segmenter av Sb02 og segmenter av Sb09 i S. spontaneum; Sb05 ble delt inn i to segmenter som fusjonerte med segmenter av Sb06 og segmenter av Sb07 S. spontaneum. Derfor var sonder som tilsvarer Sb08 og Sb05 ikke spesifikke for enkelt S. spontaneum-kromosomer, mens de andre sonder som tilsvarer de andre åtte sorghum-kromosomene kan brukes som kromosom-spesifikke cytogenetiske BAC-FISH-sonder for S. spontaneum. Dermed var de testede BAC-probene basert på de åtte sorghumkromosomene (tabell 2) tilstrekkelig for kromosomidentifikasjon ved bruk av BAC-FISH. Et satellittkromosom ble funnet i ett S. spontaneum-kromosom (kromosom 3) (Ha et al., 1999). Tidligere avslørte samtidig FISH at signalene til 45S rDNA var lokalisert på de sekundære innsnevringene av satellittkromosomene i kromosomet 3 fra den antherkulturavledede S. spontaneum-klonen (AP85-361) (Ha et al., 1999). I denne studien ble BAC-2 som tilsvarer Sb03 og 45s rDNA kartlagt til samme sted som S. spontaneum-kromosomet, og støtter dermed ytterligere tidligere funn (Ha et al., 1999). Resultatene våre ga også direkte bevis for at kromosom 3 av S. spontaneum oppkalt i forrige studie er homologt med Sb03.
Konklusjon
I denne studien utviklet vi kromosomspesifikke BACer av S spontaneum som et skritt mot utviklingen av en enkel og reproduserbar metode for kromosomidentifikasjon ved bruk av BAC-FISH cytogenetiske markører, som bekrefter muligheten for å isolere kromosomspesifikke BACer basert på sorghumgenomet for å konstruere et fysisk kart over sukkerrør. Vi gir også det første cytogenetiske beviset på inter-kromosomal omlegging mellom sorghum og S. spontaneum. Etableringen av sukkerrør BAC-FISH-teknologisystemet gir nye muligheter og midler for sukkerrørsmolekylær cytogenetikkforskning, inkludert karyotype-analyse, genlokalisering og fysisk kartkonstruksjon. Disse resultatene er essensielle for montering av S. spontaneum-genom som kreves for helgenomsekvensering.
Forfatterbidrag
GD, JS og JZ oppfattet studien og designet eksperimentene. GD, JS, QZ, JW, QY, RM, KW og JZ utførte eksperimentene og analyserte dataene. GD og JZ skrev manuskriptet. Alle forfattere leste og godkjente sluttoppgaven.
Finansiering
Dette prosjektet ble støttet av tilskudd fra 863-programmet (2013AA102604), NSFC (31201260), Program for New Century Excellent Talents in Fujian Province University, og finansieringen fra Fujian Agriculture and Forestry University.
Erklæring om interessekonflikter
Forfatterne erklærer at forskningen ble utført i fravær av kommersielle eller økonomiske forhold som kan tolkes som en potensiell interessekonflikt.
Anerkjennelser
Vi takker Dr. Xintan Zhang for å samle BAC-sekvensene og Dr. Irene Lavagi for redigering av engelsk.
Supplerende materiale
Dhont, A. (2005). Unraveling genomstrukturen til polyploids ved hjelp av FISH og GISH; eksempler på sukkerrør og banan. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
PubMed Abstract | CrossRef Fulltekst | Google Scholar
Piperidis, G., D’Hont, A. og Hogarth, D. M. (2001). «Kromosomsammensetningsanalyse av forskjellige interspesifikke Saccharum-hybrider etter genomisk in situ (hybridisering) (GISH),» i Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD: ASSCT), 565-566.
Google Scholar
