Introduction
La canne à sucre (Saccharum spp., Poaceae) est une culture annuelle ou pérenne cultivée dans les régions tropicales et subtropicales du monde entier. La canne à sucre est une culture importante pour la production de saccharose et représente 70% de la production mondiale de sucre. De plus, en tant qu’usine C4, la canne à sucre peut convertir efficacement l’énergie solaire en énergie chimique et constitue donc une culture de biocarburant idéale pour la production d’éthanol (Lam et al., 2009; Santchurn et al., 2014). Le genre Saccharum comprend six espèces: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense et S. edule, parmi lesquelles S. spontaneum est considérée comme l’espèce sauvage avec un ensemble chromosomique de base de x = 8. S. spontaneum est également l’une des deux principales espèces de Saccharum contribuant au développement des cultivars modernes de canne à sucre (Dhont et al., 1998; Ha et al., 1999). S. spontaneum a une forte adaptabilité environnementale et contient des traits génétiques importants pour la résistance aux maladies et à la sécheresse, contribuant ainsi à la tolérance au stress des hybrides de cultivars modernes (Grivet et al., 2004). S. spontaneum a la distribution géographique la plus large, et ses niveaux de ploïdie vont de 2n = 5x = 40 à 2n = 16x = 128 (Panje et Babu, 1960).
Les chromosomes de la canne à sucre ont des morphologies similaires et ont une petite taille de 1 à 6 μm au stade de la métaphase condensée (Ha et al., 1999; Dhont, 2005); par conséquent, il est très difficile de discerner entre les différents chromosomes sur la base des méthodes cytogénétiques traditionnelles. De plus, en tant qu’autopolyploïde, ses altérations structurelles chromosomiques causées par la polyploïdisation, la duplication, la suppression et la recombinaison sont très fréquentes (Piperidis et al., 2010). Chez S. spontaneum, des cartes de liaison génétique ont été développées (Silva et al., 1995), mais le développement d’une carte génétique cytologique a pris du retard par rapport aux autres espèces de graminées. Des outils cytologiques fiables pour identifier les chromosomes individuels peuvent être utilisés pour une recherche génomique efficace et une utilisation des ressources de germoplasme, en particulier avec le génome complexe des cultivars modernes de canne à sucre. Les clones de chromosomes artificiels bactériens spécifiques aux chromosomes (BAC) sont une ressource inestimable pour les recherches sur le génome de la canne à sucre, et auront de nombreuses applications dans la cartographie physique, l’identification des chromosomes et la sélection assistée par marqueurs de Saccharum spp., Et dans l’aide aux projets de séquençage et d’assemblage du génome de la canne à sucre. .
L’hybridation in situ par fluorescence (FISH) est un outil puissant pour la cytologie moléculaire (Jiang et Gill, 2006). Dans Saccharum spp., L’ADNr 45S et l’ADNr 5S ont été utilisés comme sondes pour détecter les chromosomes de base de S. spontaneum, S. robustum et S. officinarum, pour identifier les différents chromosomes de base, x = 8 dans S. spontaneum, et x = 10 chez S. robustum et S. officinarum (Dhont et al., 1998; Ha et al., 1999). D’après FISH, les cultivars modernes de canne à sucre contenaient 70 à 80% des chromosomes dérivés de S. officinarum et 10 à 23% des chromosomes de S. spontaneum, tandis que 5 à 17% semblent être le produit de la recombinaison entre S. spontaneum et S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). De plus, des événements d’élimination, de recombinaison et de translocation chromosomiques ont été détectés dans certains progéniteurs dérivés de l’hybridation entre Saccharum et Erianthus arundinaceus à l’aide de FISH (Dhont et al., 1995; Huang et al., 2015).
Sorgho a un petit génome diploïde (730 Mbp) avec une faible fréquence d’événements de recombinaison chromosomique. Le sorgho partage une haute synténie avec le génome de la canne à sucre, ce qui en fait une plante de référence idéale pour les analyses comparatives avec le génome de la canne à sucre (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014) ). Dans cette étude, sur la base des séquences disponibles pour les ressources BAC à couverture élevée, des sondes BAC fiables ont été développées pour l’identification des chromosomes FISH et la construction de cartes cytogénétiques. Les objectifs de cette étude étaient de: (1) développer un ensemble de sondes BAC-FISH spécifiques aux chromosomes sur le chromosome de S. spontaneum et (2) explorer le réarrangement chromosomique entre S. spontaneum et le sorgho.
Matériaux et méthodes
Matériels
Saccharum spontaneum SES208 (2n = 8x = 64) a été utilisé pour les analyses cytologiques dans cette étude. Les plantes SES208 ont été cultivées sur le terrain sur le campus de l’Université agricole et forestière du Fujian (Fuzhou, Chine) en février 2015 et maintenues dans des conditions de croissance régulières de la canne à sucre.
Une bibliothèque BAC a été construite à partir du génome haploïde de S. spontaneum AP85-441 (4x = 32), qui a été dérivée de S. spontaneum SES208 (2n = 8x = 64) via une culture d’anthère in vitro. La taille du génome d’AP85-441 est d’environ 3,2 Gbp. La banque se composait de 38 400 clones, avec une taille d’insert moyenne de 100 kb, couvrant 6 x du génome entier.Cette bibliothèque de BAC a été utilisée pour identifier les transporteurs de saccharose et les familles de gènes de fructokinase dans S. spontaneum (Zhang et al., 2016; Chen et al., 2017).
Criblage de la bibliothèque BAC
35 156 clones BAC des bibliothèques AP85-441 ont été regroupés dans 701 bibliothèques. Chaque bibliothèque contient en moyenne 50 clones BAC. Les bibliothèques d’ADN ont été préparées avec le kit d’ADN PhasePrepTM BAC (Sigma, États-Unis) en suivant les protocoles du fabricant. Les bibliothèques d’ADN BAC ont été séquencées en utilisant la plate-forme Illumina HiSeq 2500 avec le modèle PE250. Un total de 686 bibliothèques (18 bibliothèques ayant échoué pour le séquençage) ont été séquencées et 267,5 Gb de données nettoyées ont été générées après le découpage à l’aide de Trimmomatic version 0.36 (Bankevich et al., 2012). Les données de chaque pool BAC ont été assemblées à l’aide de SPAdes version 3.09 avec le paramètre par défaut. Un total de 2 611 145 contigs ont été assemblés avec un contig N50 de 7,38 kbp (Zhang et Ming, données non publiées).
Les séquences de la bibliothèque AP85-441 BAC ont été recherchées par BLAST contre le génome du sorgho avec une valeur E de 1e -4. Les séquences qui avaient des hits BLAST uniques dans le génome du sorgho ont été sélectionnées comme sondes FISH candidates. Pour identifier les clones BAC à faible nombre de copies à partir de la bibliothèque BAC, des amorces spécifiques à la séquence ont été conçues en utilisant le logiciel Primer 5.0 pour permettre le criblage spécifique des pools BAC par PCR. Des longueurs d’amorces de 18 à 25 pb, des valeurs Tm de 55 à 65 ° C et des compositions nucléotidiques de 40 à 60% de cytosine et de guanine ont été sélectionnées. Une méthode de PCR en deux étapes a été utilisée pour cribler les pools de dimensions 3D de la bibliothèque BAC (Asakawa et al., 1997; Crooijmans et al., 2000) avec quelques modifications. Chaque clone d’une plaque à 384 puits a été cultivé dans 80 ul de bouillon Lysogeny + 34 mg / ml de chloramphénicol pendant une nuit à 37 ° C dans une plaque de culture à 384 puits. Au total, huit plaques de 384 puits avec un total de 3072 clones BAC ont été cultivées. Pour chaque plaque de 384 puits, nous avons d’abord construit 16 pools de rangées (rangées A à P) et 24 pools de colonnes (colonnes 1 à 24) pour chaque plaque de 384 puits en mélangeant des volumes égaux de culture de chaque clone individuel, puis mélangé le 16 pools de lignes et 24 pools de colonnes avec des volumes égaux de chaque pool de lignes et de colonnes en tant que pool de plaques ou superpool. Dans cette étude, nous nous sommes concentrés sur le criblage de huit plaques de 384 puits. Ainsi, huit superpools ont été préparés séparément pour les huit plaques. La plaque positive à 384 puits des huit plaques a été identifiée et ensuite la rangée positive parmi les 16 mélanges a été criblée. Enfin, les colonnes positives pour la PCR ont été sélectionnées parmi 24 mélanges de colonnes. Vingt-quatre réactions PCR ont été réalisées pour identifier les clones positifs des huit plaques 384 puits.
Les amplifications PCR ont été réalisées comme décrit précédemment (Bouzidi et al., 2006). Chaque réaction comprenait 7,5 μl de prémélange rTaq, 0,6 μl de chaque amorce (10 μM), 1,0 μl de matrice liquide bactérienne, pour un volume final de 20 μl. Après la dénaturation initiale à 95 ° C pendant 5 min, 35 cycles de 95 ° C pendant 30 s, 55 ° C pendant 30 s et 72 ° C pendant 1 min ont été réalisés. Les produits de PCR ont été analysés sur un gel d’agarose à 1,5%.
Préparation du chromosome en métaphase
La préparation du chromosome a été réalisée comme décrit précédemment (Lou et al., 2010) avec des modifications mineures. En bref, les plantes de S. spontaneum ont été cultivées dans des conditions de serre pour la récolte des extrémités des racines. Les extrémités des racines excisées d’environ 1 à 2 cm ont été traitées avec 0,002 mol / l de 8-hydroxyquinoléine à température ambiante pendant 2 à 4 h, rincées à l’eau pendant 15 min et fixées dans de l’éthanol: acide acétique (3: 1) pendant au moins 24 heures. h à température ambiante. Les extrémités des racines ont ensuite été digérées dans une solution enzymatique (4% de cellulose R-10 et 2% de pectolyase dans un tampon citrate 0,1 M) à 37 ° C pendant 1 h, lavées avec de l’eau déminéralisée glacée pendant 30 min, et finalement incubées dans de l’éthanol: acide acétique (3: 1) pendant environ 30 min. Les lames ont été préparées selon la méthode «séchée à la flamme» (Iovene et al., 2008).
Purification de l’ADN BAC et marquage des sondes
L’ADN BAC a été extrait avec PhasePrepTM BAC Kit d’ADN selon le manuel du fabricant. L’ADN BAC purifié a été marqué par une réaction de traduction de coupure standard, y compris de la DNase I diluée, un tampon de traduction de coupure 10 ×, de l’ADN polymérase I, des dNTP, des dUTP marqués à la biotine / digoxigénine et de l’ADN BAC. Le mélange a été incubée à 15 ° C pendant 1,5 h. Les produits coupés ont ensuite été examinés sur un gel d’agarose à 1,5% pour la présence d’un frottis entre 300 et 500 pb. Les sondes obtenues ont été stockées à -20 ° C jusqu’à utilisation.
Hybridation et détection in situ
Mélange de sondes et hybridation: Premièrement, le mélange de sondes (50% formamide désionisé, 2 × SSC, 80 ng d’ADN marqué à la digoxygénine / biotine, 10% de sulfate de dextrane , > 1 μg C ° t-100) a été placé dans un bloc chaud à 90 ° C pendant 5 min, puis immédiatement transféré sur de la glace jusqu’à ce que vous soyez prêt à sonder pour hybridizati sur. Les lames séchées flammées préalablement préparées ont été traitées avec 70% de formamide désionisé, dénaturées sur un bloc thermique à 80 ° C pendant 1.5 min, immédiatement immergé séquentiellement dans de l’éthanol à 70% glacé, de l’éthanol à 90%, puis de l’éthanol à 100% chacun pendant 5 min, puis séché à l’air sur le banc. Enfin, des sondes dénaturées ont été ajoutées à chaque lame et recouvertes de lamelles 24 × 32 mm. Les lames ont été placées dans une chambre humide à 37 ° C pendant une nuit.
Détection par sonde: les lamelles ont été retirées et les lames ont été lavées à température ambiante dans 2 × SSC pendant 5 min, à 42 ° C dans 2 × SSC pendant 10 min et à température ambiante dans 1 x PBS pendant 5 min dans cette séquence. Les signaux de sonde marqués à la biotine ont été détectés avec 2 mg / ml de streptavidine Alexa Fluor 488 et les signaux de sonde marqués à la digoxygénine ont été détectés avec 2% d’anti-digoxigénine-rhodamine provenant de moutons. Le cocktail d’anticorps (100 μl de tampon TNB, 1 μl de streptavidine Alexa Fluor 488, 1 μl de rhodamine anti-dig-mouton) a été ajouté aux lames, qui ont été recouvertes de lamelles 24 × 32 mm, incubées pendant 1 h à 37 ° C dans une chambre humide, et lavé à température ambiante dans 1 × PBS trois fois pendant 5 min chacun. L’excès de liquide a été éliminé et du 4 ‘, 6-diamidino-2-phénylindole (DAPI) (Sigma, St. Louis, MO, États-Unis) dans la solution antifade Vectashield (Vector, Burlingame, CA, États-Unis) a été ajouté pour contre-colorer le chromosomes. Les images ont été capturées avec un microscope à épifluorescence Olympus BX63. Les images du signal FISH ont été analysées à l’aide du logiciel CellSens Dimension.
Résultats
Criblage des clones BAC à faible copie
Pour cribler les clones BAC qui ont potentiellement un faible nombre de copies séquences dans le génome de S. spontaneum, nous avons recherché BLAST dans les bibliothèques séquencées AP85-441 BAC contre le génome du sorgho. Un total de 2000 séquences BAC correspondant à 10 chromosomes de sorgho avec une bonne colinéarité et présentant une spécificité de séquence ont été criblés. Les séquences BAC de 2000 AP85-441 ont été masquées par REPEATMASKER contre la base de données ADN de sorgho à répétition élevée et la base de données TIGR gramineae pour filtrer les séquences répétées. Enfin, 114 BAC répartis sur 10 chromosomes de sorgho ont été sélectionnés pour l’analyse FISH (tableau supplémentaire 1) avec 7 à 16 BAC sur chacun des 10 chromosomes de sorgho (figure 1). Pour cribler les clones BAC positifs, 114 amorces spécifiques ont été conçues sur la base des séquences de clones BAC (tableau supplémentaire 2) et 49 clones BAC positifs ont été criblés à partir des bibliothèques BAC (tableau supplémentaire 3).
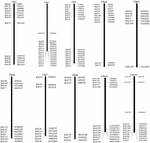
FIGURE 1. Les distributions de 114 clones BAC de AP85-441 dans les chromosomes du sorgho. BAC1-114 représente le numéro BAC, les données sur la droite sont l’emplacement physique du BAC dans la région correspondante des chromosomes du sorgho. Unit = bp.
Force et distribution du signal BAC-FISH
Échantillons d’ADN isolés des 49 échantillons positifs Les clones BAC ont été marqués avec de la biotine ou de la digoxigénine. Des sondes d’ADNr 5S et d’ADNr 45S ont été utilisées comme contrôle et ont été hybridées aux chromosomes métaphase somatique de S. spontaneum suivant la procédure FISH. En raison de la corrélation entre la force du signal et la variation du contenu des séquences répétées des séquences BAC, nous avons utilisé C ° t-100 comme concurrent (tableau 1). Les clones BAC affichant des signaux d’hybridation forts et stables ont été sélectionnés pour une analyse FISH plus approfondie.

TABLEAU 1. La classification de 27 clones positifs avec un ADN bloquant.
Un total de 64 chromosomes distincts peut être observé dans la métaphase du S. spontaneum. Chaque BAC-FISH a été réalisé dans quatre expériences indépendantes (ou lames). Au moins 10 étalements de chromosomes somatiques en métaphase ont été analysés dans chaque lame. Les résultats ont montré que les 27 sondes spécifiques (tableau supplémentaire 4) peuvent être classées en cinq groupes en fonction du signal d’hybridation (tableau 1). Dans le groupe I (y compris BAC-18, BAC-20, BAC-29 et BAC-84), les sondes ont affiché huit sites distinctifs sans suppression compétitive in situ (CISS) en utilisant C ° t-100 (figure 2A), suggérant peu de répétition des séquences existaient dans ces séquences BAC. Dans le groupe II (y compris BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 et BAC-74), C ° t -100 a été utilisé comme compétiteur pour l’hybridation de la sonde BAC, et six à sept sites assez distincts ont été affichés dans les caryotypes de ce groupe (figure 2B). Le résultat de l’hybridation était similaire à l’ADNr 45S avec sept sites de signal, ce qui peut être causé par une variation de la structure chromosomique du chromosome homologue. Dans le groupe III (y compris BAC-26, BAC-66, BAC-76 et BAC-78), l’ADN C ° t-100 a été utilisé comme concurrent et huit sites distincts ont pu être observés dans le caryotype, suggérant ce groupe de BAC les clones ont des séquences d’ADN répétitives (figure 2C). Dans le groupe IV (y compris BAC-24), plus de huit sites de signal ont été observés dans le caryotype et sont apparus dans les régions péricentromériques et télomériques (figure 2D), suggérant que ces sondes BAC avaient des séquences répétitives dans S. spontaneum.Dans le groupe V (y compris BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 et BAC-81), les sondes ont montré des signaux dispersés dans tous les chromosomes avec des régions limitées des chromosomes individuels affichant des signaux spécifiques (figure 2E). Le résultat de l’hybridation a indiqué que ce groupe de clones BAC contenait de grandes quantités d’ADN répétitif dans S. spontaneum, ce qui peut être causé par une divergence évolutive rapide pour les régions répétitives après la séparation du sorgho et de S. spontaneum. Par conséquent, parmi les 49 sondes BAC, un ensemble de 19 sondes BAC (tableau supplémentaire 3) des groupes I, II et III a montré des signaux spécifiques sur les chromosomes de S. spontaneum correspondant à neuf des chromosomes de sorgho à côté de Sb05. Les signaux des 22 autres sondes ont été dispersés dans tous les chromosomes. Nous avons sélectionné un ensemble de 19 sondes BAC comme signaux spécifiques du chromosome S. spontaneum (Figure 3). Sur les 19 sondes BAC, une sonde était localisée sur Sb02, Sb04 et Sb09; deux sondes sur Sb07, Sb08 et Sb10; trois sondes sur Sb01 et Sb06; et quatre sondes sur Sb03. Cet ensemble de sondes a fourni un outil utile pour l’analyse comparative de S. spontaneum et du sorgho (Tableau 2).
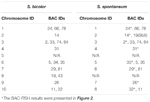
TABLEAU 2. La distribution des sondes BAC-FISH dans les chromosomes de S. bicolor et S. spontaneum.
Construction d’une bibliothèque BAC spécifique aux chromosomes de S. spontaneum
Sb01 et Sb02 sont les deux plus gros chromosomes du sorgho. Pour détecter la synténie génomique entre le sorgho et S. spontaneum, 10 clones BAC spécifiques du chromosome ont été sélectionnés dans les groupes I, II et III. Parmi ces 10 sondes BAC, trois sondes (BAC clone ID: 24, 66, 78) étaient homologues à Sb01 et sept sondes (BAC clone ID: 14, 18, 20, 69, 71, 73, 77) correspondaient à Sb02. Nous avons utilisé la détection bicolore de FISH pour les paires de BAC marquées de manière complémentaire et les sondes pour cartographier la relation cytogénétique de chaîne pour les clones BAC. Ce faisant, nous avons construit la carte cytogénétique de S. spontaneum basée sur les 10 sondes BAC.
Pour trois sondes correspondant à Sb01, l’analyse FISH de S. spontaneum (SES208) a montré que BAC-24 et BAC -78 sondes étaient principalement situées sur la région distale du chromosome 1 de S. spontaneum, et BAC-66 a été cartographiée à proximité de la région centromérique (figure 4). BAC-24 et BAC-66 localisés sur le même chromosome (chromosome 1 de S. spontaneum); simultanément alors que la sonde 66 et la sonde 78 se trouvaient également sur le même chromosome, démontrant que les trois sondes BAC se répartissaient sur les mêmes chromosomes chez S. spontaneum suggérant ainsi une synténie entre S. spontaneum et le sorgho pour le chromosome 1. Les résultats FISH ont révélé que les sept sondes BAC aligné sur Sb02; seules les sondes 69, 71, 73 et 77 des sept BAC ont généré des signaux intenses sur un chromosome de S. spontaneum (SsChr2) (figure 5); tandis que les sondes 14, 18 et 20 étaient indétectables sur le chromosome 2 de S. spontaneum mais ont été observées sur d’autres chromosomes de S. spontaneum. Ces résultats ont démontré que les réarrangements chromosomiques se sont produits dans le Sb02 correspondant entre S. spontaneum et le sorgho.
De plus, la séquence de BAC-2 partageait une forte similarité (97%) avec un fragment du chromosome 3 de sorgho. L’analyse FISH en double couleur a montré que les ADNr BAC-2 et 45s étaient cartographiés au même emplacement du chromosome de S. spontaneum (Figure 6).
Réarrangement chromosomique sur Sb02 entre Sorgho et S. spontaneum
Pour étudier plus avant le réarrangement chromosomique sur Sb02 entre le sorgho et S. spontaneum, chromosome -des sondes spécifiques correspondant à Sb02 (BAC-14, BAC-18 et BAC-20) et correspondant à Sb08 (BAC-19 et BAC-43) ont été utilisées pour l’analyse FISH. Les résultats ont montré que BAC-14 et BAC-19 ont été cartographiés à différents bras des mêmes chromosomes de S. spontaneum, indiquant que des réarrangements chromosomiques ont eu lieu entre les chromosomes 2 et 8 dans S. spontaneum et le sorgho (Figure 7).
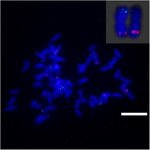
FIGURE 7. Le réarrangement du chromosome dans la canne à sucre et le sorgho tel que révélé par FISH. Sondes BAC-14 et BAC-19 marquées à la biotine (verte) et à la digoxigénine (rouge). Barres d’échelle = 10 μm.
Discussion
Le saccharum est un genre complexe caractérisé par des niveaux de polyploïdie élevés, avec de petits chromosomes (Ha et al., 1999; Dhont, 2005). Distinguer les chromosomes individuels de Saccharum en fonction de leur morphologie est très difficile. En tirant parti des séquences qui sont très couvertes par la bibliothèque BAC de S. spontaneum, nous avons pu cribler les clones potentiels de BAC à faible nombre de copies dans S. spontaneum en utilisant le génome du sorgho comme référence. En tant qu’espèce hautement polyploïde, S. spontaneum peut contenir plus de réarrangements génomiques que les diploïdes.Les différences de génomes entre S. spontaneum et le sorgho peuvent être la cause d’inexactitude dans les prédictions des séquences BAC à faible nombre de copies dans le génome de S. spontaneum. Dans cette étude, nous avons initialement obtenu 114 séquences BAC potentielles avec copie unique basée sur le génome du sorgho. Cependant, seuls 49 ont pu être identifiés dans les bibliothèques partielles de BAC, et 27 se sont avérés être des sondes BAC-FISH spécifiques des chromosomes. Il y a au moins deux raisons pour expliquer ces résultats. Premièrement, les 114 séquences BAC étaient des fragments partiels de l’insertion BAC en raison des limitations de l’assemblage de séquences NGS, et peuvent contenir des séquences répétées dans les autres régions qui n’ont aucune information de séquence. Ces séquences répétées potentiellement découvertes pourraient induire en erreur la détection d’une séquence à faible nombre de copies par comparaison de séquences. Deuxièmement, la polyploïdisation récente chez S. spontaneum peut provoquer la variation des séquences répétées entre S. spontaneum et le sorgho, et ainsi aboutir au signal FISH non spécifique chez S. spontaneum. Dans une étude précédente, le séquençage de 20 BAC dans des hybrides de canne à sucre a généré des séquences de contig de 1,45 Mb, les séquences alignées avec le génome du sorgho couvrant 0,99 Mb, ce qui représentait environ la moitié des séquences de BAC de la canne à sucre (Wang et al., 2010). De toute évidence, des suppressions / insertions existaient entre le sorgho et le saccharum, et l’utilisation du génome du sorgho comme référence ne peut pas remplacer les caractéristiques uniques du génome de Saccharum, et peut donc entraîner des résultats FISH imprévus.
Certaines diapositives FISH avaient de fortes bruit de fond, qui peut être dû à de petites régions de séquences répétées (telles que les séquences SSR) dans le clone BAC, qui produisent par conséquent des interférences lors de l’hybridation. Le génome du sorgho a une teneur en répétition d’environ 61% (Paterson et al., 2009), alors qu’environ la moitié du génome est composée de séquences répétées dans les hybrides de Saccarrum (De et al., 2014). Dans ces génomes riches en répétitions, il est difficile de développer des sondes FISH spécifiques au signal, qui distinguent cytogénétiquement les chromosomes de morphologies similaires. Le CISS avec l’ADN de blocage C ° t-100 peut empêcher efficacement la répétition des séquences. Dans cette étude, le blocage de l’ADN C ° t-100 a été utilisé pour l’analyse FISH des sondes BAC des groupes II et III (figures 2D-F), qui ont été vérifiées comme étant les sondes BAC spécifiques du chromosome, tandis que le FISH des sondes BAC dans les groupes IV et V a produit une forte interférence de fond (figures 2D – F). Il est évident qu’il existe des variations de séquences répétées parmi les sondes BAC examinées. Par conséquent, l’optimisation des expériences serait nécessaire pour l’analyse BAC-FISH dans Saccharum. Six à sept sites ont été affichés dans le groupe II, ce qui pourrait être causé par les variations de la structure du chromosome homologue, telles que la délétion de fragment dans un ou deux chromosomes homologues. Dans le blé hexaploïde, le BAC 676D4 s’hybridait plus fortement aux chromosomes du génome A qu’aux chromosomes des génomes B et D (Zhang et al., 2004). Ces indétectables d’un à deux signaux dans les chromosomes homologues pourraient également être causés par les problèmes techniques de BAC-FISH pour autant de chromosomes de petite taille.
Dans cette étude, nous avons identifié 27 BAC spécifiques aux chromosomes. Sondes FISH qui correspondent à 9 des 10 chromosomes du sorgho (tous sauf Sb05). Dans une étude de la carte génétique dérivée d’un croisement entre S. officinarum et un cultivar de canne à sucre, Sb05 a été fusionné avec Sb06 dans la canne à sucre (Aitken et al., 2014). Dans notre étude, une carte génétique basée sur la population F1 de S. spontaneum a révélé que Sb05 était divisé en deux segments et que les deux segments étaient fusionnés avec Sb06 et Sb07, respectivement (Zhang et Ming, données non publiées). Récemment, la carte génétique d’un membre des Andropogoneae, Miscanthus sinensis, a démontré que Sb05 a une mauvaise colinéarité avec le groupe de liaison correspondant chez M. sinensis (Ma et al., 2012). De même, les deux chromosomes ancestraux du maïs orthologues à Sb05 conservent le plus petit nombre d’orthologues synténiques aux gènes du sorgho (Schubert et Lysak, 2011). Par conséquent, l’absence du BAC-FISH correspondant à Sb05 peut indiquer une fusion chromosomique chez S. spontaneum.
Sorgho et S. spontaneum ont divergé il y a 12 millions d’années (MYA), le nombre de chromosomes de base a été réduit de x = 10 à x = 8. Dans Aitken et al. (2014), le HG2 (groupe homologue 2) de la canne à sucre aligné sur Sb05 et Sb06, et HG8 (groupe homologue 8) sur Sb02 et Sb08, fournissant des preuves de l’événement de réduction chromosomique de base (Aitken et al., 2014). Dans cette étude, nous avons observé que les chromosomes de sorgho Sb02 et Sb08 avaient un réarrangement interchromosomique dans S. spontaneum comme le démontre la preuve que BAC-14 s’alignait sur Sb02 et que BAC-19 était aligné sur Sb08 (Figure 7). Notre étude a fourni la première preuve physique et cytogénétique du réarrangement inter-chromosomique du sorgho chez S. spontaneum. Des données non publiées de notre groupe ont également révélé que Sb08 est divisé en deux segments, et ont été fusionnés avec Sb02 et Sb09 dans S. spontaneum, respectivement (Zhang et Ming, données non publiées).Les sondes correspondant à Sb09, Sb08, Sb05 et Sb06 pourraient en outre être utilisées pour étudier les événements interchromosomiques entre le sorgho et S. spontaneum. S. spontaneum a une large gamme de niveaux de ploïdie (2n = 40–128) (Irvine, 1999). Ces sondes BAC pourraient être utilisées pour confirmer le réarrangement inter-chromosomique de S. spontaneum avec les différents niveaux de polyploïdie.
En raison du nombre de chromosomes de base différent entre le sorgho (x = 10) et S. spontaneum (x = 8), les chromosomes du sorgho n’étaient pas une correspondance univoque avec S. spontaneum. Comme notre étude de cartographie génétique mentionnée ci-dessus, Sb08 a été divisé en deux segments qui ont fusionné avec des segments de Sb02 et des segments de Sb09 dans S. spontaneum; Sb05 a été divisé en deux segments qui ont fusionné avec des segments de Sb06 et des segments de Sb07 S. spontaneum. Par conséquent, les sondes correspondant à Sb08 et Sb05 n’étaient pas spécifiques de chromosomes uniques de S. spontaneum, tandis que les autres sondes correspondant aux huit autres chromosomes de sorgho peuvent être utilisées comme sondes cytogénétiques BAC-FISH spécifiques des chromosomes pour S. spontaneum. Ainsi, les sondes BAC testées étaient basées sur les huit chromosomes de sorgho (tableau 2) étaient suffisantes pour l’identification des chromosomes à l’aide de BAC-FISH. Un chromosome satellite a été trouvé dans un chromosome de S. spontaneum (chromosome 3) (Ha et al., 1999). Auparavant, le FISH simultané a révélé que les signaux de l’ADNr 45S étaient localisés sur les constrictions secondaires des chromosomes satellites dans le chromosome 3 du clone S. spontaneum dérivé de la culture d’anthère (AP85-361) (Ha et al., 1999). Dans cette étude, BAC-2 qui correspond à Sb03 et l’ADNr 45s ont été cartographiés au même emplacement du chromosome de S. spontaneum, confirmant ainsi les découvertes précédentes (Ha et al., 1999). Nos résultats ont également fourni des preuves directes que le chromosome 3 de S. spontaneum nommé dans l’étude précédente est homologue à Sb03.
Conclusion
Dans cette étude, nous avons développé des BAC spécifiques du chromosome de S . spontaneum comme une étape vers le développement d’une méthode simple et reproductible pour l’identification des chromosomes à l’aide de marqueurs cytogénétiques BAC-FISH, confirmant la faisabilité d’isoler des BAC spécifiques aux chromosomes basés sur le génome du sorgho pour construire une carte physique de la canne à sucre. Nous fournissons également la première preuve cytogénétique de réarrangement inter-chromosomique entre le sorgho et S. spontaneum. La mise en place du système technologique BAC-FISH de la canne à sucre offre de nouvelles opportunités et des moyens pour la recherche en cytogénétique moléculaire de la canne à sucre, y compris l’analyse des caryotypes, la localisation des gènes et la construction de cartes physiques. Ces résultats sont essentiels pour l’assemblage du génome de S. spontaneum requis pour le séquençage du génome entier.
Contributions des auteurs
GD, JS et JZ ont conçu l’étude et conçu les expériences. GD, JS, QZ, JW, QY, RM, KW et JZ ont réalisé les expériences et analysé les données. GD et JZ ont écrit le manuscrit. Tous les auteurs ont lu et approuvé l’article final.
Financement
Ce projet a été soutenu par des subventions du programme 863 (2013AA102604), NSFC (31201260), Programme for New Century Excellent Talents in Université de la province du Fujian, et le financement de l’Université d’agriculture et de foresterie du Fujian.
Déclaration de conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière cela pourrait être interprété comme un conflit d’intérêts potentiel.
Remerciements
Nous remercions le Dr Xintan Zhang pour l’assemblage des séquences BAC et le Dr Irene Lavagi pour l’édition de l’anglais.
Matériel supplémentaire
Dhont, A. (2005). Démêler la structure du génome des polyploïdes à l’aide de FISH et GISH; exemples de canne à sucre et de banane. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
Résumé PubMed | CrossRef Texte intégral | Google Scholar
Piperidis, G., D’Hont, A., et Hogarth, D. M. (2001). « Analyse de la composition chromosomique de divers hybrides interspécifiques de Saccharum par génomique in situ (hybridation) (GISH) », dans Actes de l’International Society of Sugar Cane Technologists, XXIV Congrès (Brisbane, QLD: ASSCT), 565-566.
Google Scholar
