Einführung
Zuckerrohr (Saccharum spp., Poaceae) ist eine einjährige oder mehrjährige Kultur, die in tropischen und subtropischen Regionen weltweit angebaut wird. Zuckerrohr ist eine wichtige Ernte für die Saccharoseproduktion und macht 70% der weltweiten Zuckerproduktion aus. Darüber hinaus kann Zuckerrohr als C4-Anlage Sonnenenergie effizient in chemische Energie umwandeln und ist daher eine ideale Biokraftstoffpflanze für die Ethanolproduktion (Lam et al., 2009; Santchurn et al., 2014). Die Gattung Saccharum hat sechs Arten: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense und S. edule, unter denen S. spontaneum als Wildart mit einem grundlegenden Chromosomensatz von x angesehen wird = 8. S. spontaneum ist auch eine der beiden Hauptarten von Saccharum, die zur Entwicklung moderner Zuckerrohrsorten beitragen (Dhont et al., 1998; Ha et al., 1999). S. spontaneum hat eine starke Umweltanpassungsfähigkeit und enthält wichtige genetische Merkmale für Krankheits- und Trockenresistenz, was zur Stresstoleranz moderner Sortenhybriden beiträgt (Grivet et al., 2004). S. spontaneum hat die breiteste geografische Verteilung und seine Ploidiewerte reichen von 2n = 5x = 40 bis 2n = 16x = 128 (Panje und Babu, 1960).
Zuckerrohrchromosomen haben ähnliche Morphologien und eine kleine Größe von 1–6 μm im Stadium der kondensierten Metaphase (Ha et al., 1999; Dhont, 2005); Daher ist es sehr schwierig, auf der Grundlage traditioneller zytogenetischer Methoden zwischen den verschiedenen Chromosomen zu unterscheiden. Darüber hinaus sind als Autopolyploid seine chromosomalen Strukturveränderungen, die durch Polyploidisierung, Duplikation, Deletion und Rekombination verursacht werden, sehr häufig (Piperidis et al., 2010). In S. spontaneum wurden genetische Verknüpfungskarten entwickelt (Silva et al., 1995), aber die Entwicklung einer zytologischen genetischen Karte blieb hinter anderen Grasarten zurück. Zuverlässige zytologische Werkzeuge zur Identifizierung einzelner Chromosomen können für eine effektive Genomforschung und die Nutzung von Keimplasma-Ressourcen verwendet werden, insbesondere mit dem komplexen Genomhintergrund moderner Zuckerrohrsorten. Chromosomenspezifische bakterielle künstliche Chromosomenklone (BAC) sind eine unschätzbare Ressource für die Erforschung des Zuckerrohrgenoms und werden viele Anwendungen bei der physikalischen Kartierung, Chromosomenidentifizierung und markergestützten Züchtung der Saccharum spp. Sowie bei der Unterstützung von Sequenzierungs- und Assemblierungsprojekten für das Zuckerrohrgenom haben
Die Fluoreszenz-in-situ-Hybridisierung (FISH) ist ein leistungsfähiges Werkzeug für die molekulare Zytologie (Jiang und Gill, 2006). In Saccharum spp. Wurden 45S-rDNA und 5S-rDNA als Sonden verwendet, um die Grundchromosomen von S. spontaneum, S. robustum und S. officinarum nachzuweisen, um die verschiedenen Grundchromosomen zu identifizieren, x = 8 in S. spontaneum und x = 10 in S. robustum und S. officinarum (Dhont et al., 1998; Ha et al., 1999). Basierend auf FISH wurde festgestellt, dass moderne Zuckerrohrsorten 70–80% der von S. officinarum abgeleiteten Chromosomen und 10–23% der Chromosomen von S. spontaneum enthalten, während 5–17% das Produkt der Rekombination zwischen zu sein scheinen S. spontaneum und S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). Darüber hinaus wurden Chromosomeneliminierungs-, Rekombinations- und Translokationsereignisse in einigen Vorläufern nachgewiesen, die aus der Hybridisierung zwischen Saccharum und Erianthus arundinaceus unter Verwendung von FISH stammen (Dhont et al., 1995; Huang et al., 2015).
Sorghum hat ein kleines diploides Genom (730 Mbp) mit einer geringen Häufigkeit von Chromosomenrekombinationsereignissen. Sorghum teilt eine hohe Syntenie mit dem Zuckerrohrgenom, was es zu einer idealen Referenzpflanze für vergleichende Analysen mit dem Zuckerrohrgenom macht (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014) ). In dieser Studie wurden basierend auf den verfügbaren Sequenzen für BAC-Ressourcen mit hoher Abdeckung zuverlässige BAC-Sonden für die Identifizierung von FISH-Chromosomen und die zytogenetische Kartenkonstruktion entwickelt. Die Ziele dieser Studie waren: (1) Entwicklung eines Satzes chromosomenspezifischer BAC-FISH-Sonden auf S. spontaneum-Chromosomen und (2) Untersuchung der Chromosomenumlagerung zwischen S. spontaneum und Sorghum.
Materialien und Methoden
Materialien
Saccharum spontaneum SES208 (2n = 8x = 64) wurde in dieser Studie für zytologische Analysen verwendet. Die SES208-Pflanzen wurden im Februar 2015 auf dem Campus der Fujian Agricultural and Forestry University (Fuzhou, China) auf dem Feld gezüchtet und unter regulären Zuckerrohrwachstumsbedingungen gehalten.
Aus dem haploiden Genom wurde eine BAC-Bibliothek konstruiert von S. spontaneum AP85-441 (4x = 32), das von S. spontaneum SES208 (2n = 8x = 64) über eine andere In-vitro-Kultur abgeleitet wurde. Die Genomgröße von AP85-441 beträgt etwa 3,2 Gbp. Die Bibliothek bestand aus 38.400 Klonen mit einer durchschnittlichen Insertgröße von 100 kb, die 6 × des gesamten Genoms abdeckten.Diese BAC-Bibliothek wurde zur Identifizierung der Saccharosetransporter und Fructokinase-Genfamilien in S. spontaneum verwendet (Zhang et al., 2016; Chen et al., 2017).
Screening der BAC-Bibliothek
35.156 BAC-Klone aus den AP85-441-Bibliotheken wurden in 701 Bibliotheken zusammengefasst. Jede Bibliothek enthält durchschnittlich 50 BAC-Klone. Die DNA-Bibliotheken wurden mit dem PhasePrepTM BAC DNA Kit (Sigma, USA) gemäß den Protokollen des Herstellers hergestellt. BAC-DNA-Bibliotheken wurden unter Verwendung der Illumina HiSeq 2500-Plattform mit PE250-Modell sequenziert. Insgesamt 686 Bibliotheken (18 Bibliotheken konnten nicht sequenziert werden) wurden sequenziert, und 267,5 GB gereinigte Daten wurden nach dem Trimmen unter Verwendung von Trimmomatic Version 0.36 (Bankevich et al., 2012) erzeugt. Daten aus jedem BAC-Pool wurden mit SPAdes Version 3.09 mit Standardparameter zusammengestellt. Insgesamt 2.611.145 Contigs wurden mit Contig N50 von 7,38 kbp (Zhang und Ming, unveröffentlichte Daten) zusammengesetzt. Die Sequenzen der AP85-441-BAC-Bibliothek wurden BLAST gegen das Sorghum-Genom mit einem E-Wert von 1e durchsucht -4. Die Sequenzen, die einzelne BLAST-Treffer im Sorghum-Genom hatten, wurden als Kandidaten-FISH-Sonden ausgewählt. Um BAC-Klone mit niedriger Kopienzahl aus der BAC-Bibliothek zu identifizieren, wurden sequenzspezifische Primer unter Verwendung der Primer 5.0-Software entworfen, um das spezifische Screening von BAC-Pools durch PCR zu ermöglichen. Primerlängen von 18–25 bp, Tm-Werte von 55–65 ° C und Nukleotidzusammensetzungen von 40–60% Cytosin und Guanin wurden ausgewählt. Ein zweistufiges PCR-Verfahren wurde verwendet, um die 3D-Dimensionspools der BAC-Bibliothek (Asakawa et al., 1997; Crooijmans et al., 2000) mit einigen Modifikationen zu screenen. Jeder Klon von einer 384-Well-Platte wurde in 80 & mgr; l Lysogeny-Brühe + 34 mg / ml Chloramphenicol über Nacht bei 37 ° C in einer 384-Well-Kulturplatte kultiviert. Insgesamt wurden acht 384-Well-Platten mit insgesamt 3072 BAC-Klonen kultiviert. Für jede 384-Well-Platte konstruierten wir zuerst 16 Reihenpools (Reihe A bis P) und 24 Säulenpools (Spalte 1–24) für jede 384-Well-Platte, indem wir gleiche Kulturvolumina jedes einzelnen Klons mischten und dann die mischten 16 Zeilenpools und 24 Spaltenpools zusammen mit gleichen Volumina aus jedem Zeilen- und Spaltenpool als Einplattenpool oder Superpool. In dieser Studie konzentrierten wir uns auf das Screening von acht 384-Well-Platten. Somit wurden acht Superpools für die acht Platten getrennt hergestellt. Die positive 384-Well-Platte aus den acht Platten wurde identifiziert und anschließend die positive Reihe unter den 16 Gemischen gescreent. Schließlich wurden die PCR-positiven Säulen aus 24 Säulenmischungen ausgewählt. Vierundzwanzig PCR-Reaktionen wurden durchgeführt, um positive Klone aus den acht 384-Well-Platten zu identifizieren.
PCR-Amplifikationen wurden wie zuvor beschrieben durchgeführt (Bouzidi et al., 2006). Jede Reaktion umfasste rTaq-Vormischung 7,5 & mgr; l, 0,6 & mgr; l jedes Primers (10 & mgr; M), 1,0 & mgr; l Bakterienflüssigkeitstemplat bis zu einem Endvolumen von 20 & mgr; l. Nach anfänglicher Denaturierung bei 95 ° C für 5 min wurden 35 Zyklen von 95 ° C für 30 s, 55 ° C für 30 s und 72 ° C für 1 min durchgeführt. PCR-Produkte wurden auf einem 1,5% igen Agarosegel analysiert.
Metaphase-Chromosomen-Präparation
Die Chromosomen-Präparation wurde wie zuvor beschrieben (Lou et al., 2010) mit geringfügigen Modifikationen durchgeführt. Kurz gesagt, S. spontaneum-Pflanzen wurden unter Gewächshausbedingungen für die Wurzelspitzenernte gezüchtet. Herausgeschnittene Wurzelspitzen von etwa 1–2 cm wurden 2–4 h bei Raumtemperatur mit 0,002 mol / l 8-Hydroxychinolin behandelt, 15 min in Wasser gespült und mindestens 24 in Ethanol: Essigsäure (3: 1) fixiert h bei Raumtemperatur. Die Wurzelspitzen wurden dann in einer Enzymlösung (4% Cellulose R-10 und 2% Pektolyase in 0,1 M Citratpuffer) 1 h bei 37 ° C verdaut, 30 min mit eisentionisiertem Wasser gewaschen und schließlich in Ethanol inkubiert: Essigsäure (3: 1) für ca. 30 min. Die Objektträger wurden gemäß der „flammengetrockneten“ Methode (Iovene et al., 2008) hergestellt.
BAC-DNA-Reinigung und Sondenmarkierung
Die BAC-DNA wurde mit PhasePrepTM BAC extrahiert DNA-Kit gemäß Herstellerhandbuch. Gereinigte BAC-DNA wurde durch Standard-Nick-Translationsreaktion markiert, einschließlich verdünnter DNase I, 10 × Nick-Translationspuffer, DNA-Polymerase I, dNTPs, Biotin / Digoxigenin-markierten dUTPs und BAC-DNA wurde 1,5 h bei 15 ° C inkubiert. Die geschnittenen Produkte wurden dann auf einem 1,5% igen Agarosegel auf das Vorhandensein eines Abstrichs zwischen 300 und 500 bp untersucht. Die erhaltenen Sonden wurden bis zur Verwendung bei -20 ° C gelagert.
In-situ-Hybridisierung und -Detektion
Sondenmischung und Hybridisierung: Zuerst die Sondenmischung (50% entionisiertes Formamid, 2 × SSC, 80 ng Digoxygenin / Biotin-markierte DNA, 10% Dextransulfat , > 1 & mgr; g C ° t-100) wurde 5 Minuten lang in einen 90 ° C heißen Block gegeben und dann sofort auf Eis übertragen, bis sie bereit waren, auf Hybridisierung zu prüfen auf. Die zuvor hergestellten flammgetrockneten Objektträger wurden mit 70% entionisiertem Formamid behandelt und 1 auf einem Wärmeblock bei 80 ° C denaturiert.5 min, sofort nacheinander in eiskaltes 70% iges Ethanol, 90% iges Ethanol und dann jeweils 5 min lang 100% iges Ethanol getaucht und dann auf der Bank luftgetrocknet. Schließlich wurden jedem Objektträger denaturierte Sonden zugesetzt und mit 24 × 32 mm Deckgläsern bedeckt. Die Objektträger wurden über Nacht in eine feuchte Kammer bei 37 ° C gestellt. Sondenerkennung: Die Deckgläser wurden entfernt und die Objektträger wurden 5 Minuten bei Raumtemperatur in 2 × SSC bei 42 ° C in 2 × gewaschen SSC für 10 min und bei Raumtemperatur in 1 × PBS für 5 min in dieser Sequenz. Biotin-markierte Sondensignale wurden mit 2 mg / ml Alexa Fluor 488 Streptavidin nachgewiesen, und Digoxygenin-markierte Sondensignale wurden mit 2% Anti-Digoxigenin-Rhodamin von Schafen nachgewiesen. Der Antikörpercocktail (100 & mgr; l TNB-Puffer, 1 & mgr; l Alexa Fluor 488 Streptavidin, 1 & mgr; l Rhodamin-Anti-Dig-Schaf) wurde zu den Objektträgern gegeben, die mit 24 × 32 mm Deckgläsern bedeckt waren und 1 h bei 37 ° C in inkubiert wurden eine feuchte Kammer und dreimal bei Raumtemperatur in 1 × PBS jeweils 5 Minuten lang gewaschen. Überschüssige Flüssigkeit wurde entfernt und 4 ‚, 6-Diamidino-2-phenylindol (DAPI) (Sigma, St. Louis, MO, USA) in der Antifade-Lösung Vectashield (Vector, Burlingame, CA, USA) wurde zugegeben, um das zu färben Chromosomen. Die Bilder wurden mit einem Olympus BX63-Epifluoreszenzmikroskop aufgenommen. FISH-Signalbilder wurden mit der CellSens Dimension-Software analysiert.
Ergebnisse
Screening von BAC-Klonen mit geringer Kopie
Zum Screening der BAC-Klone, die möglicherweise eine niedrige Kopienzahl aufweisen Sequenzen im S. spontaneum-Genom haben wir BLAST die sequenzierten AP85-441-BAC-Bibliotheken gegen das Sorghum-Genom durchsucht. Insgesamt 2000 BAC-Sequenzen, die 10 Sorghum-Chromosomen mit guter Kollinearität und Sequenzspezifität entsprachen, wurden gescreent. Die 2000 AP85-441 BAC-Sequenzen wurden von REPEATMASKER gegen die Sorghum-DNA-Datenbank mit hoher Wiederholung und die TIGR-Gramineae-Datenbank maskiert, um Wiederholungssequenzen zu filtern. Schließlich wurden 114 auf 10 Sorghum-Chromosomen verteilte BACs für die FISH-Analyse (Ergänzungstabelle 1) mit 7–16 BACs auf jedem der 10 Sorghum-Chromosomen ausgewählt (Abbildung 1). Um positive BAC-Klone zu screenen, wurden 114 spezifische Primer basierend auf den BAC-Klonsequenzen (Ergänzungstabelle 2) entworfen und 49 positive BAC-Klone wurden aus den BAC-Bibliotheken (Ergänzungstabelle 3) gescreent.
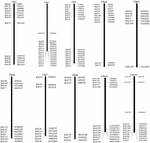
ABBILDUNG 1. Die Verteilungen von 114 BAC-Klonen von AP85-441 in den Chromosomen von Sorghum. BAC1-114 stellt die BAC-Nummer dar, die Daten rechts sind der physikalische Ort von BAC in der entsprechenden Region der Sorghum-Chromosomen. Einheit = bp.
BAC-FISH-Signalstärke und -verteilung
DNA-Proben, die aus den 49 positiven isoliert wurden BAC-Klone wurden mit Biotin oder Digoxigenin markiert. 5S-rDNA- und 45S-rDNA-Sonden wurden als Kontrolle verwendet und nach dem FISH-Verfahren mit somatischen Metaphasenchromosomen von S. spontaneum hybridisiert. Aufgrund der Korrelation zwischen der Signalstärke und der Variation des wiederholten Sequenzgehalts der BAC-Sequenzen verwendeten wir C ° t-100 als Konkurrent (Tabelle 1). BAC-Klone, die starke und stetige Hybridisierungssignale zeigten, wurden für die weitere FISH-Analyse ausgewählt.

TABELLE 1. Die Klassifizierung von 27 positiven Klonen mit blockierender DNA.
Insgesamt können 64 verschiedene Chromosomen beobachtet werden in der Metaphase des S. spontaneum. Jeder BAC-FISH wurde in vier unabhängigen Experimenten (oder Objektträgern) durchgeführt. In jedem Objektträger wurden mindestens 10 Ausbreitungen somatischer Metaphasenchromosomen analysiert. Die Ergebnisse zeigten, dass die 27 spezifischen Sonden (Ergänzungstabelle 4) basierend auf dem Hybridisierungssignal (Tabelle 1) in fünf Gruppen eingeteilt werden können. In Gruppe I (einschließlich BAC-18, BAC-20, BAC-29 und BAC-84) zeigten Sonden acht charakteristische Stellen ohne kompetitive In-situ-Unterdrückung (CISS) unter Verwendung von C ° t-100 (Fig. 2A), was darauf hindeutet, dass nur wenige wiederholt wurden Sequenzen existierten in diesen BAC-Sequenzen. In Gruppe II (einschließlich BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 und BAC-74) wurde C ° t -100 wurde als Konkurrent für die BAC-Sondenhybridisierung verwendet, und sechs bis sieben ziemlich unterschiedliche Stellen wurden in Karyotypen dieser Gruppe angezeigt (2B). Das Hybridisierungsergebnis war ähnlich wie bei 45S-rDNA mit sieben Signalstellen, was durch Variation der Chromosomenstruktur im homologen Chromosom verursacht werden kann. In Gruppe III (einschließlich BAC-26, BAC-66, BAC-76 und BAC-78) wurde C ° t-100-DNA als Konkurrent verwendet, und acht verschiedene Stellen konnten im Karyotyp beobachtet werden, was auf diese Gruppe von BAC hinweist Klone haben repetitive DNA-Sequenzen (2C). In Gruppe IV (einschließlich BAC-24) wurden mehr als acht Signalstellen im Karyotyp beobachtet und traten in den perizentromeren und telomeren Regionen auf (2D), was darauf hindeutet, dass diese BAC-Sonden repetitive Sequenzen in S. spontaneum hatten.In Gruppe V (einschließlich BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 und BAC-81) zeigten die Sonden dispergierte Signale in allen Chromosomen mit begrenzten Regionen von einzelnen Chromosomen, die spezifische Signale anzeigen (Abbildung 2E). Das Hybridisierungsergebnis zeigte, dass diese Gruppe von BAC-Klonen große Mengen repetitiver DNA in S. spontaneum enthielt, was durch eine schnelle evolutionäre Divergenz der repetitiven Regionen nach der Spaltung von Sorghum und S. spontaneum verursacht werden kann. Daher zeigte von den 49 BAC-Sonden ein Satz von 19 BAC-Sonden (Ergänzungstabelle 3) aus den Gruppen I, II und III spezifische Signale auf S. spontaneum-Chromosomen, die neun der Sorghum-Chromosomen neben Sb05 entsprachen. Die Signale der anderen 22 Sonden waren in allen Chromosomen verteilt. Wir haben einen Satz von 19 BAC-Sonden als chromosomenspezifische S. spontaneum-Signale ausgewählt (Abbildung 3). Von den 19 BAC-Sonden befand sich eine Sonde an Sb02, Sb04 und Sb09; zwei Sonden an Sb07, Sb08 und Sb10; drei Sonden an Sb01 und Sb06; und vier Sonden an Sb03. Dieser Sondensatz bot ein nützliches Werkzeug für die vergleichende Analyse von S. spontaneum und Sorghum (Tabelle 2).
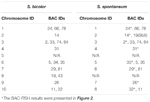
TABELLE 2. Die Verteilung von BAC-FISH-Sonden in Chromosomen von S. bicolor und S. spontaneum.
Aufbau einer für S. spontaneum-Chromosomen spezifischen BAC-Bibliothek
Sb01 und Sb02 sind die beiden größten Sorghum-Chromosomen. Um die Genomsyntenie zwischen Sorghum und S. spontaneum nachzuweisen, wurden 10 chromosomenspezifische BAC-Klone aus den Gruppen I, II und III ausgewählt. Von diesen 10 BAC-Sonden waren drei Sonden (BAC-Klon-ID: 24, 66, 78) homolog zu Sb01 und sieben Sonden (BAC-Klon-ID: 14, 18, 20, 69, 71, 73, 77) entsprachen Sb02. Wir verwendeten den zweifarbigen Nachweis von FISH für komplementär markierte BAC-Paare und die Sonden zur Kartierung der kettenzytogenetischen Beziehung für die BAC-Klone. Auf diese Weise konstruierten wir die zytogenetische Karte von S. spontaneum basierend auf den 10 BAC-Sonden.
Für drei Sonden, die Sb01 entsprechen, zeigte die FISH-Analyse von S. spontaneum (SES208), dass BAC-24 und BAC -78 Sonden befanden sich hauptsächlich in der distalen Region von S. spontaneum Chromosom 1, und BAC-66 wurde in unmittelbarer Nähe der zentromeren Region kartiert (Abbildung 4). BAC-24 und BAC-66 befinden sich auf demselben Chromosom (S. spontaneum-Chromosom 1); gleichzeitig, während sich Sonde 66 und Sonde 78 ebenfalls auf demselben Chromosom befanden, was zeigt, dass die drei BAC-Sonden in S. spontaneum auf dieselben Chromosomen verteilt waren, was auf eine Syntenie zwischen S. spontaneum und Sorghum für Chromosom 1 hindeutet. Die FISH-Ergebnisse zeigten, dass die sieben BAC-Sonden ausgerichtet auf Sb02; Nur die Sonden 69, 71, 73 und 77 der sieben BACs erzeugten intensive Signale auf einem S. spontaneum-Chromosom (SsChr2) (Abbildung 5). Die Sonden 14, 18 und 20 waren auf S. spontaneum-Chromosom 2 nicht nachweisbar, wurden jedoch auf anderen S. spontaneum-Chromosomen beobachtet. Diese Ergebnisse zeigten, dass die Chromosomenumlagerungen im entsprechenden Sb02 zwischen S. spontaneum und Sorghum auftraten.
Darüber hinaus hatte die Sequenz von BAC-2 eine hohe Ähnlichkeit (97%) mit einem Fragment auf Chromosom 3 von Sorghum. Eine zweifarbige FISH-Analyse zeigte, dass sowohl BAC-2- als auch 45s-rDNA auf die gleiche Stelle des S. spontaneum-Chromosoms abgebildet wurden (6).
Chromosomenumlagerung auf Sb02 zwischen Sorghum und S. spontaneum
Um die Chromosomenumlagerung auf Sb02 zwischen Sorghum und S. spontaneum, Chromosom, weiter zu untersuchen -spezifische Sonden entsprechend Sb02 (BAC-14, BAC-18 und BAC-20) und entsprechend Sb08 (BAC-19 und BAC-43) wurden für die FISH-Analyse verwendet. Die Ergebnisse zeigten, dass BAC-14 und BAC-19 auf verschiedene Arme derselben S. spontaneum-Chromosomen abgebildet wurden, was darauf hinweist, dass Chromosomenumlagerungen zwischen den Chromosomen 2 und 8 in S. spontaneum und Sorghum stattfanden (7)
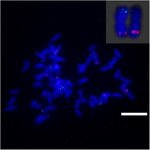
ABBILDUNG 7. Die Umlagerung des Chromosoms in Zuckerrohr und Sorghum, wie durch FISH gezeigt. Mit Biotin (grün) und Digoxigenin (rot) markierte BAC-14- und BAC-19-Sonden. Maßstabsbalken = 10 μm.
Diskussion
Saccharum ist eine komplexe Gattung, die durch hohe Polyploidie-Niveaus gekennzeichnet ist. mit kleinen Chromosomen (Ha et al., 1999; Dhont, 2005). Die Unterscheidung einzelner Saccharum-Chromosomen anhand ihrer Morphologie ist sehr schwierig. Unter Verwendung von Sequenzen, die stark von der BAC-Bibliothek von S. spontaneum abgedeckt sind, konnten wir anhand des Sorghum-Genoms als Referenz nach potenziellen BAC-Klonen mit niedriger Kopienzahl in S. spontaneum suchen. Als hochpolyploide Spezies kann S. spontaneum mehr Genomumlagerungen enthalten als Diploide.Die Unterschiede der Genome zwischen S. spontaneum und Sorghum können die Ursache für die Ungenauigkeit der Vorhersagen der BAC-Sequenzen mit niedriger Kopienzahl im S. spontaneum-Genom sein. In dieser Studie haben wir zunächst 114 potenzielle BAC-Sequenzen mit Einzelkopie basierend auf dem Sorghum-Genom erhalten. In den partiellen BAC-Bibliotheken konnten jedoch nur 49 identifiziert werden, und 27 erwiesen sich als chromosomenspezifische BAC-FISH-Sonden. Es gibt mindestens zwei Gründe, um diese Ergebnisse zu erklären. Erstens waren die 114 BAC-Sequenzen aufgrund der Einschränkungen der NGS-Sequenzanordnung Teilfragmente der BAC-Insertion und können Wiederholungssequenzen in den anderen Regionen enthalten, die keine Sequenzinformationen aufweisen. Diese möglicherweise nicht abgedeckten Wiederholungssequenzen könnten den Nachweis einer Sequenz mit geringer Kopie durch Sequenzvergleich irreführen. Zweitens kann die kürzliche Polyploidisierung in S. spontaneum die Variation von Wiederholungssequenzen zwischen S. spontaneum und Sorghum verursachen und somit zu dem unspezifischen FISH-Signal in S. spontaneum führen. In einer früheren Studie erzeugte die Sequenzierung von 20 BACs in Zuckerrohrhybriden 1,45-Mb-Contig-Sequenzen, wobei die mit dem Sorghum-Genom übereinstimmenden Sequenzen 0,99 Mb überspannten, was etwa die Hälfte der Zuckerrohr-BAC-Sequenzen ausmachte (Wang et al., 2010). Offensichtlich gab es Deletionen / Insertionen zwischen Sorghum und Saccharum, und die Verwendung des Sorghum-Genoms als Referenz kann die einzigartigen Merkmale des Saccharum-Genoms nicht ersetzen und kann daher zu unvorhergesehenen FISH-Ergebnissen führen.
Einige FISH-Objektträger waren stark Hintergrundrauschen, das auf kleine Regionen von Wiederholungssequenzen (wie SSR-Sequenzen) im BAC-Klon zurückzuführen sein kann, die folglich während der Hybridisierung Interferenzen erzeugen. Das Sorghum-Genom hat einen Wiederholungsgehalt von ungefähr 61% (Paterson et al., 2009), während ungefähr die Hälfte des Genoms aus Wiederholungssequenzen in Saccarrum-Hybriden besteht (De et al., 2014). In diesen wiederholungsreichen Genomen ist es schwierig, signalspezifische FISH-Sonden zu entwickeln, die Chromosomen zytogenetisch mit ähnlichen Morphologien unterscheiden. CISS mit blockierender DNA C ° t-100 kann Wiederholungssequenzen effizient ausschließen. In dieser Studie wurde blockierende DNA C ° t-100 für die FISH-Analyse der BAC-Sonden in den Gruppen II und III (Abbildungen 2D – F) verwendet, die als chromosomenspezifische BAC-Sonden verifiziert wurden, während die FISH von BAC-Sonden In den Gruppen IV und V traten starke Hintergrundstörungen auf (Abbildungen 2D – F). Es ist offensichtlich, dass es Variationen der Wiederholungssequenzen unter den untersuchten BAC-Sonden gibt. Daher wäre eine Optimierung der Experimente für die BAC-FISH-Analyse in Saccharum erforderlich. In Gruppe II wurden sechs bis sieben Stellen angezeigt, die durch homologe Chromosomenstrukturvariationen wie die Fragmentdeletion in einem oder zwei homologen Chromosomen verursacht werden könnten. In hexaploiden Weizen hybridisierte BAC 676D4 stärker mit den Chromosomen des A-Genoms als mit den Chromosomen des B- und D-Genoms (Zhang et al., 2004). Diese nicht nachweisbaren ein bis zwei Signale in den homologen Chromosomen könnten auch durch die technischen Probleme von BAC-FISH für so viele Chromosomen mit geringer Größe verursacht werden.
In dieser Studie identifizierten wir 27 chromosomenspezifische BAC- FISH-Sonden, die 9 der 10 Sorghum-Chromosomen entsprechen (alle außer Sb05). In einer Studie der genetischen Karte, die aus einer Kreuzung zwischen S. officinarum und Zuckerrohrsorte stammt, wurde Sb05 mit Sb06 in Zuckerrohr fusioniert (Aitken et al., 2014). In unserer Studie ergab eine genetische Karte, die auf der F1-Population von S. spontaneum basierte, dass Sb05 in zwei Segmente unterteilt war und die beiden Segmente mit Sb06 bzw. Sb07 zusammengeführt wurden (Zhang und Ming, unveröffentlichte Daten). Kürzlich hat die genetische Karte eines Mitglieds der Andropogoneae, Miscanthus sinensis, gezeigt, dass Sb05 eine schlechte Kollinearität mit der entsprechenden Verknüpfungsgruppe in M. sinensis aufweist (Ma et al., 2012). In ähnlicher Weise behalten die beiden zu Sb05 orthologen Mais-Chromosomen der Vorfahren die geringste Anzahl syntenischer Orthologe zu Sorghum-Genen bei (Schubert und Lysak, 2011). Daher kann das Fehlen des BAC-FISH, das Sb05 entspricht, auf eine Chromosomenfusion in S. spontaneum hinweisen. Sorghum und S. spontaneum gingen vor 12 Millionen Jahren auseinander (MYA), die Grundchromosomenzahl wurde von x reduziert = 10 bis x = 8. In Aitken et al. (2014), das HG2 (homologe Gruppe 2) von Zuckerrohr, ausgerichtet an Sb05 und Sb06, und HG8 (homologe Gruppe 8) an Sb02 und Sb08, liefern Hinweise auf das grundlegende Chromosomenreduktionsereignis (Aitken et al., 2014). In dieser Studie beobachteten wir, dass die Sorghum-Chromosomen Sb02 und Sb08 eine interchromosomale Umlagerung in S. spontaneum aufwiesen, wie durch den Nachweis gezeigt wurde, dass BAC-14 an Sb02 und BAC-19 an Sb08 ausgerichtet war (7). Unsere Studie lieferte den ersten physikalischen und zytogenetischen Beweis für die Sorghum-Interchromosomen-Umlagerung in S. spontaneum. Unveröffentlichte Daten aus unserer Gruppe zeigten auch, dass Sb08 in zwei Segmente unterteilt ist und mit Sb02 und Sb09 in S. spontaneum zusammengeführt wurde (Zhang und Ming, unveröffentlichte Daten).Die Sonden, die Sb09, Sb08, Sb05 und Sb06 entsprechen, könnten weiter zur Untersuchung der interchromosomalen Ereignisse zwischen Sorghum und S. spontaneum verwendet werden. S. spontaneum weist einen weiten Bereich von Ploidie-Spiegeln auf (2n = 40–128) (Irvine, 1999). Diese BAC-Sonden könnten verwendet werden, um die interchromosomale Umlagerung von S. spontaneum mit den verschiedenen Polyploidie-Spiegeln zu bestätigen.
Aufgrund der unterschiedlichen Grundchromosomenzahl zwischen Sorghum (x = 10) und S. spontaneum (x =) 8) waren die Sorghum-Chromosomen keine Eins-zu-Eins-Entsprechung mit S. spontaneum. Wie in unserer zuvor erwähnten genetischen Kartierungsstudie wurde Sb08 in zwei Segmente unterteilt, die mit Segmenten von Sb02 und Segmenten von Sb09 in S. spontaneum verschmolzen; Sb05 wurde in zwei Segmente unterteilt, die mit Segmenten von Sb06 und Segmenten von Sb07 S. spontaneum verschmolzen. Daher waren die Sonden, die Sb08 und Sb05 entsprachen, nicht spezifisch für einzelne S. spontaneum-Chromosomen, während die anderen Sonden, die den anderen acht Sorghum-Chromosomen entsprachen, als chromosomenspezifische zytogenetische BAC-FISH-Sonden für S. spontaneum verwendet werden können. Somit basierten die getesteten BAC-Sonden auf den acht Sorghum-Chromosomen (Tabelle 2), die für die Chromosomenidentifikation unter Verwendung von BAC-FISH ausreichend waren. Ein Satellitenchromosom wurde in einem S. spontaneum-Chromosom (Chromosom 3) gefunden (Ha et al., 1999). Zuvor zeigte simultanes FISH, dass sich die Signale der 45S-rDNA auf den sekundären Verengungen der Satellitenchromosomen innerhalb des Chromosoms 3 des von der Antherenkultur abgeleiteten S. spontaneum-Klons (AP85-361) befanden (Ha et al., 1999). In dieser Studie wurde BAC-2, das Sb03- und 45s-rDNA entspricht, auf dieselbe Stelle des S. spontaneum-Chromosoms abgebildet, wodurch frühere Ergebnisse weiter gestützt wurden (Ha et al., 1999). Unsere Ergebnisse lieferten auch direkte Beweise dafür, dass das in der vorherigen Studie genannte Chromosom 3 von S. spontaneum homolog zu Sb03 ist.
Schlussfolgerung
In dieser Studie entwickelten wir chromosomenspezifische BACs von S. Spontaneum als Schritt zur Entwicklung einer einfachen und reproduzierbaren Methode zur Identifizierung von Chromosomen unter Verwendung von zytogenetischen BAC-FISH-Markern, die die Machbarkeit der Isolierung chromosomenspezifischer BACs auf der Basis des Sorghum-Genoms zur Erstellung einer physikalischen Karte von Zuckerrohr bestätigt. Wir liefern auch den ersten zytogenetischen Nachweis einer interchromosomalen Umlagerung zwischen Sorghum und S. spontaneum. Die Einrichtung des Zuckerrohr-BAC-FISH-Technologiesystems bietet neue Möglichkeiten und Mittel für die Forschung zur molekularen Zytogenetik von Zuckerrohr, einschließlich Karyotypanalyse, Genlokalisierung und physikalischer Kartenkonstruktion. Diese Ergebnisse sind für die Assemblierung des S. spontaneum-Genoms, das für die Sequenzierung des gesamten Genoms erforderlich ist, wesentlich.
Autorenbeiträge
GD, JS und JZ konzipierten die Studie und entwarfen die Experimente. GD, JS, QZ, JW, QY, RM, KW und JZ führten die Experimente durch und analysierten die Daten. GD und JZ haben das Manuskript geschrieben. Alle Autoren haben das Abschlusspapier gelesen und genehmigt.
Finanzierung
Dieses Projekt wurde durch Zuschüsse aus dem Programm 863 (2013AA102604), NSFC (31201260), Programm für hervorragende Talente des neuen Jahrhunderts in unterstützt Fujian Province University und die Finanzierung durch die Fujian Agriculture and Forestry University.
Erklärung zu Interessenkonflikten
Die Autoren erklären, dass die Forschung ohne kommerzielle oder finanzielle Beziehungen durchgeführt wurde Dies könnte als potenzieller Interessenkonflikt ausgelegt werden.
Danksagung
Wir danken Dr. Xintan Zhang für die Zusammenstellung der BAC-Sequenzen und Dr. Irene Lavagi für die Bearbeitung der englischen Sprache.
Ergänzungsmaterial
Dhont, A. (2005). Entschlüsselung der Genomstruktur von Polyploiden mit FISH und GISH; Beispiele für Zuckerrohr und Banane. Cytogenet. Genom Res. 109, 27–33. doi: 10.1159 / 000082378
PubMed Abstract | CrossRef Volltext | Google Scholar
Piperidis, G., D’Hont, A. und Hogarth, D. M. (2001). „Analyse der Chromosomenzusammensetzung verschiedener interspezifischer Saccharum-Hybride durch genomische in situ (Hybridisierung) (GISH)“ in Proceedings der International Society of Sugar Cane Technologists, XXIV. Kongress (Brisbane, QLD: ASSCT), 565–566.
Google Scholar
