Introducere
Trestia de zahăr (Saccharum spp., Poaceae) este o cultură anuală sau perenă cultivată în regiunile tropicale și subtropicale din întreaga lume. Trestia de zahăr este o cultură importantă pentru producția de zaharoză și reprezintă 70% din producția mondială de zahăr. În plus, ca plantă C4, trestia de zahăr poate converti eficient energia solară în energie chimică și, prin urmare, este o cultură ideală de biocombustibil pentru producerea etanolului (Lam și colab., 2009; Santchurn și colab., 2014). Genul Saccharum are șase specii: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense și S. edule, printre care S. spontaneum este considerată specia sălbatică cu un set de cromozomi de bază de x = 8. S. spontaneum este, de asemenea, una dintre cele două specii principale de Saccharum care contribuie la dezvoltarea modernă a soiurilor de zahăr (Dhont și colab., 1998; Ha și colab., 1999). S. spontaneum are o puternică adaptabilitate la mediu și conține trăsături genetice importante pentru rezistența la boli și secetă, contribuind astfel la toleranța la stres a hibrizilor de soiuri moderne (Grivet și colab., 2004). S. spontaneum are cea mai largă distribuție geografică, iar nivelurile sale de ploidie variază de la 2n = 5x = 40 la 2n = 16x = 128 (Panje și Babu, 1960).
Cromozomii din trestie de zahăr au morfologii similare și au o mică dimensiunea de 1–6 μm la stadiul de metafază condensată (Ha și colab., 1999; Dhont, 2005); prin urmare, este foarte dificil să discernem între diferiții cromozomi pe baza metodelor citogenetice tradiționale. Mai mult, ca autopoliploid, modificările sale structurale cromozomiale cauzate de poliploidizare, duplicare, ștergere și recombinare sunt foarte frecvente (Piperidis și colab., 2010). În S. spontaneum, au fost dezvoltate hărți de legătură genetică (Silva și colab., 1995), dar dezvoltarea unei hărți genetice citologice a rămas în urma celorlalte specii de iarbă. Unele instrumente citologice fiabile pentru identificarea cromozomilor individuali pot fi utilizate pentru cercetarea eficientă a genomului și utilizarea resurselor germoplasmei, în special pe baza genomului complex al soiurilor moderne de trestie de zahăr. Clonele cu cromozomi artificiali bacteriali specifici cromozomului (BAC) sunt o resursă neprețuită pentru cercetările genomului trestiei de zahăr și vor avea multe aplicații în cartografierea fizică, identificarea cromozomilor și reproducerea asistată de markeri a Saccharum spp. .
Hibridizarea in situ a fluorescenței (FISH) este un instrument puternic pentru citologia moleculară (Jiang și Gill, 2006). În Saccharum spp., 45S rADN și 5S rADN au fost utilizate ca sonde pentru a detecta cromozomii de bază ai S. spontaneum, S. robustum și S. officinarum, pentru a identifica diferiții cromozomi de bază, x = 8 în S. spontaneum și x = 10 în S. robustum și S. officinarum (Dhont și colab., 1998; Ha și colab., 1999). Pe baza FISH, s-a constatat că soiurile moderne de trestie de zahăr conțin 70-80% din cromozomii derivați din S. officinarum și 10-23% din cromozomii din S. spontaneum, în timp ce 5-17% pare a fi produsul recombinării dintre S. spontaneum și S. officinarum (Dhont și colab., 1996; Piperidis și colab., 2001; Cuadrado și colab., 2004). Mai mult, evenimentele de eliminare, recombinare și translocare a cromozomilor au fost detectate la unii progenitori derivați din hibridizarea dintre Saccharum și Erianthus arundinaceus folosind FISH (Dhont și colab., 1995; Huang și colab., 2015).
Sorghum are un genom diploid mic (730 Mbp) cu o frecvență scăzută a evenimentelor de recombinare a cromozomilor. Sorgul împărtășește o sinteză ridicată cu genomul trestiei de zahăr, făcându-l o plantă de referință ideală pentru analize comparative cu genomul trestiei de zahăr (Ming și colab., 1998; Nazeema și colab., 2007; Wang și colab., 2010; Aitken și colab., 2014 ). În acest studiu, pe baza secvențelor disponibile pentru resurse BAC cu acoperire ridicată, s-au dezvoltat sonde BAC fiabile pentru identificarea cromozomilor FISH și construcția hărții citogenetice. Obiectivele acestui studiu au fost: (1) dezvoltarea unui set de sonde BAC-FISH specifice cromozomilor pe cromozomul S. spontaneum și (2) explorarea rearanjării cromozomului dintre S. spontaneum și sorg.
Materiale și metode
Materiale
Saccharum spontaneum SES208 (2n = 8x = 64) a fost utilizat pentru analizele citologice în acest studiu. Plantele SES208 au fost cultivate pe teren în campusul Universității Agricole și Silvice Fujian (Fuzhou, China) în februarie 2015 și menținute în condiții regulate de creștere a trestiei de zahăr.
O bibliotecă BAC a fost construită din genomul haploid. de S. spontaneum AP85-441 (4x = 32), care a fost derivat din S. spontaneum SES208 (2n = 8x = 64) prin cultură anteră in vitro. Dimensiunea genomului AP85-441 este de aproximativ 3,2 Gbp. Biblioteca a constat din 38.400 de clone, cu o dimensiune medie a insertului de 100 kb, acoperind 6 × din întregul genom.Această bibliotecă BAC a fost utilizată pentru identificarea transportorilor de zaharoză și a familiilor de gene fructokinazice din S. spontaneum (Zhang și colab., 2016; Chen și colab., 2017).
Screening the BAC Library
35.156 clone BAC din bibliotecile AP85-441 au fost reunite în 701 biblioteci. Fiecare bibliotecă conține în medie 50 de clone BAC. Bibliotecile de ADN au fost pregătite cu PhasePrep ™ BAC DNA Kit (Sigma, Statele Unite) urmând protocoalele producătorului. Bibliotecile de ADN BAC au fost secvențiate utilizând platforma Illumina HiSeq 2500 cu model PE250. Au fost secvențiate un total de 686 de biblioteci (18 biblioteci nereușite pentru secvențiere) și s-au generat 267,5 Gb de date curățate după tăiere utilizând versiunea Trimmomatic 0.36 (Bankevich și colab., 2012). Datele din fiecare pool BAC au fost asamblate folosind SPAdes versiunea 3.09 cu parametrul implicit. Un total de 2.611.145 contigs au fost asamblate cu contig N50 de 7,38 kbp (Zhang și Ming, date nepublicate).
Secvențele bibliotecii AP85-441 BAC au fost căutate BLAST împotriva genomului sorgului cu o valoare E de 1e -4. Secvențele care au avut lovituri BLAST unice în genomul sorgului au fost selectate ca probe FISH candidate. Pentru a identifica clonele BAC cu număr redus de copii din biblioteca BAC, primeri specifici secvenței au fost proiectați utilizând software-ul Primer 5.0 pentru a permite piscinelor BAC să fie ecranate în mod specific de PCR. Au fost selectate lungimi primare de 18-25 bp, valori Tm de 55-65 ° C și compoziții de nucleotide de 40-60% citozină și guanină. O metodă PCR în doi pași a fost utilizată pentru a ecraniza grupurile de dimensiuni 3D ale bibliotecii BAC (Asakawa și colab., 1997; Crooijmans și colab., 2000) cu unele modificări. Fiecare clonă dintr-o placă de 384 de godeuri a fost cultivată în 80 pl bulion de lizogenie + 34 mg / ml cloramfenicol peste noapte la 37 ° C într-o placă de cultură de 384 de godeuri. În total, au fost cultivate opt plăci cu 384 de godeuri cu un total de 3072 clone BAC. Pentru fiecare placă de 384 de godeuri, am construit mai întâi 16 grupuri de rânduri (rândul A până la P) și 24 de piscine de coloane (coloana 1-24) pentru fiecare placă de 384 de godeuri, amestecând volume egale de cultură pentru fiecare clonă individuală, apoi am amestecat 16 grupuri de rânduri și 24 de coloane, împreună cu volume egale din fiecare rând și coloană, ca un pool de plăci sau superpool. În acest studiu, ne-am concentrat asupra screening-ului a opt plăci cu 384 de godeuri. Astfel, s-au pregătit opt super-piscine pentru cele opt plăci separat. Placa pozitivă de 384 de godeuri din cele opt plăci a fost identificată și ulterior a fost selectat rândul pozitiv dintre cele 16 amestecuri. În cele din urmă, coloanele pozitive PCR au fost selectate dintre 24 de amestecuri de coloane. Douăzeci și patru de reacții PCR au fost efectuate pentru a identifica clone pozitive din cele opt plăci cu 384 de godeuri.
Amplificările PCR au fost efectuate așa cum s-a descris anterior (Bouzidi și colab., 2006). Fiecare reacție a inclus rTaq premix 7,5 μl, 0,6 μl din fiecare primer (10 μM), 1,0 μl șablon lichid de bacterii, până la un volum final de 20 μl. După denaturarea inițială la 95 ° C timp de 5 min, s-au efectuat 35 de cicluri de 95 ° C timp de 30 s, 55 ° C timp de 30 s și 72 ° C timp de 1 min. Produsele PCR au fost analizate pe un gel de agaroză 1,5%.
Prepararea cromozomului metafazic
Pregătirea cromozomială a fost efectuată așa cum s-a descris anterior (Lou și colab., 2010) cu modificări minore. Pe scurt, plantele S. spontaneum au fost cultivate în condiții de seră pentru recoltarea vârfurilor rădăcinii. Vârfurile rădăcinii excizate de aproximativ 1-2 cm au fost tratate cu 0,002 mol / l 8-hidroxichinolină la temperatura camerei timp de 2-4 ore, clătite în apă timp de 15 minute și fixate în etanol: acid acetic (3: 1) timp de cel puțin 24 h la temperatura camerei. Vârfurile rădăcinii au fost apoi digerate într-o soluție enzimatică (4% celuloză R-10 și 2% pectoliază în tampon citrat 0,1 M) la 37 ° C timp de 1 oră, spălate cu apă deionizată cu gheață timp de 30 min și, în cele din urmă, incubate în etanol: acid acetic (3: 1) timp de aproximativ 30 min. Diapozitivele au fost preparate conform metodei „uscate cu flacără” (Iovene și colab., 2008).
Purificarea ADN BAC și etichetarea sondei
ADN-ul BAC a fost extras cu PhasePrep ™ BAC ADN Kit conform manualului producătorului. ADN-ul BAC purificat a fost marcat prin reacție standard de traducere nick, incluzând DNază I diluată, 10 × tampon de traducere nick, ADN Polimerază I, dNTPs, dUTP-uri marcate cu biotină / digoxigenină și ADN BAC. a fost incubat la 15 ° C timp de 1,5 h. Produsele tăiate au fost apoi examinate pe un gel de agaroză 1,5% pentru prezența unui frotiu între 300 și 500 bp. Sondele obținute au fost depozitate la -20 ° C până la utilizare.
Hibridizare și detectare in situ
Amestec de sondă și hibridizare: Mai întâi, amestecul de sondă (50% formamidă deionizată, 2 × SSC, 80 ng ADN marcat cu digoxigenină / biotină, 10% sulfat de dextran , > 1 μg C ° t-100) a fost plasat într-un bloc fierbinte de 90 ° C timp de 5 minute, apoi a fost transferat imediat pe gheață până când a fost gata de sondare pentru hibridizare pe. Diapozitivele uscate cu flacără pregătite anterior au fost tratate cu 70% formamidă deionizată, denaturate pe un bloc de căldură la 80 ° C timp de 1.5 min, imersat imediat secvențial în etanol 70% răcit cu gheață, 90% etanol și apoi 100% etanol fiecare timp de 5 min, și apoi uscat la aer pe bancă. În cele din urmă, sonde denaturate au fost adăugate la fiecare lamă și acoperite cu lamele de acoperire de 24 × 32 mm. Lamele au fost plasate într-o cameră umedă la 37 ° C peste noapte.
Detectarea sondei: lamelele de acoperire au fost îndepărtate și lamele au fost spălate la temperatura camerei în 2 × SSC timp de 5 minute, la 42 ° C în 2 × SSC timp de 10 min și la temperatura camerei în 1 × PBS timp de 5 min în această secvență. Semnalele sondei marcate cu biotină au fost detectate cu 2 mg / ml Alexa Fluor 488 streptavidină și semnalele sondei marcate cu digoxigenină au fost detectate cu 2% anti-digoxigenină-rodamină de la oi. Cocktailul cu anticorpi (100 μl tampon TNB, 1 μl Alexa Fluor 488 streptavidin, 1 μl rodamină anti-dig-oi) a fost adăugat la lamele, care au fost acoperite cu lamele de acoperire de 24 × 32 mm, incubate 1 oră la 37 ° C în o cameră umedă și spălată la temperatura camerei în 1 × PBS de trei ori timp de 5 minute fiecare. Excesul de lichid a fost îndepărtat și a fost adăugat 4 ′, 6-diamidino-2-fenilindol (DAPI) (Sigma, St. Louis, MO, Statele Unite) în soluția antifade Vectashield (Vector, Burlingame, CA, Statele Unite) pentru a contracara cromozomi. Imaginile au fost capturate cu un microscop de epifluorescență Olympus BX63. Imaginile de semnal FISH au fost analizate folosind software-ul CellSens Dimension.
Rezultate
Screening Clones BAC Low-Copy
Pentru a ecraniza clonele BAC care pot avea un număr redus de copii secvențe în genomul S. spontaneum, am explorat BLAST bibliotecile secvențiate AP85-441 BAC împotriva genomului sorgului. Au fost selectate un total de 2000 de secvențe BAC corespunzătoare a 10 cromozomi de sorg cu o bună colinearitate și care prezintă specificitatea secvenței. Secvențele BAC AP85-441 din 2000 au fost mascate de REPEATMASKER împotriva bazei de date ADN de sorg cu repetare ridicată și a bazei de date cu grame TIGR pentru a filtra secvențele repetate. În cele din urmă, 114 BAC distribuiți pe 10 cromozomi de sorg au fost selectați pentru analiza FISH (Tabelul 1 suplimentar) cu 7-16 BAC pe fiecare dintre cei 10 cromozomi de sorg (Figura 1). Pentru a selecta clonele BAC pozitive, au fost proiectați 114 primeri specifici pe baza secvențelor de clone BAC (Tabelul 2 suplimentar) și 49 de clone BAC pozitive au fost selectate din bibliotecile BAC (Tabelul suplimentar 3).
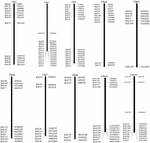
FIGURA 1. Distribuțiile a 114 clone BAC de la AP85-441 în cromozomii sorgului. BAC1-114 reprezintă numărul BAC, datele din dreapta sunt locația fizică a BAC în regiunea corespunzătoare a cromozomilor de sorg. Unit = bp.
Puterea și distribuția semnalului BAC-FISH
Probele de ADN izolate din cele 49 pozitive Clonele BAC au fost marcate cu biotină sau digoxigenină. Sondele 5S rADN și 45S rADN au fost utilizate ca martor și au fost hibridizate cu cromozomi somatici ai metafazei S. spontaneum în urma procedurii FISH. Datorită corelației dintre puterea semnalului și variația conținutului de secvențe repetate ale secvențelor BAC, am folosit C ° t-100 ca concurent (Tabelul 1). Clonele BAC care afișează semnale de hibridizare puternice și constante au fost selectate pentru analiza ulterioară FISH.

TABEL 1. Clasificarea a 27 de clone pozitive cu ADN blocant.
Se pot observa un total de 64 de cromozomi diferiți în metafaza S. spontaneum. Fiecare BAC-FISH a fost efectuat în patru experimente independente (sau diapozitive). Cel puțin 10 răspândiri de cromozomi metafazici somatici au fost analizate în fiecare lamă. Rezultatele au arătat că cele 27 de sonde specifice (tabelul suplimentar 4) pot fi clasificate în cinci grupuri pe baza semnalului de hibridizare (tabelul 1). În grupa I (inclusiv BAC-18, BAC-20, BAC-29 și BAC-84), sondele au afișat opt situri distinctive fără supresie in situ competitivă (CISS) folosind C ° t-100 (Figura 2A), sugerând câteva repetări secvențe au existat în aceste secvențe BAC. În grupa II (inclusiv BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 și BAC-74), C ° t -100 a fost utilizat ca concurent pentru hibridizarea sondei BAC și șase până la șapte situri destul de distincte au fost afișate în cariotipurile acestui grup (Figura 2B). Rezultatul hibridizării a fost similar cu ADNr 45S cu șapte situsuri de semnal, care pot fi cauzate de variația structurii cromozomiale în cromozomul omolog. În grupa III (inclusiv BAC-26, BAC-66, BAC-76 și BAC-78), ADN C ° t-100 a fost utilizat ca concurent și opt situri distincte au putut fi observate în cariotip, sugerând acest grup de BAC clonele au secvențe repetitive de ADN (Figura 2C). În grupa IV (inclusiv BAC-24), mai mult de opt situri de semnal au fost observate în cariotip și au apărut în regiunile pericentromerice și telomerice (Figura 2D), sugerând că aceste sonde BAC aveau secvențe repetitive în S. spontaneum.În grupa V (inclusiv BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 și BAC-81), sondele au prezentat semnale dispersate în toți cromozomii cu regiuni limitate a cromozomilor individuali care prezintă semnale specifice (Figura 2E). Rezultatul hibridizării a indicat faptul că acest grup de clone BAC conținea cantități mari de ADN repetitiv în S. spontaneum, care poate fi cauzat de o divergență evolutivă rapidă pentru regiunile repetitive după împărțirea sorgului și S. spontaneum. Prin urmare, din cele 49 de sonde BAC, un set de 19 sonde BAC (Tabelul suplimentar 3) din grupele I, II și III au arătat semnale specifice pe cromozomii S. spontaneu corespunzător cu nouă dintre cromozomii de sorg lângă Sb05. Semnalele celorlalte 22 de sonde au fost dispersate în toți cromozomii. Am selectat un set de 19 sonde BAC ca semnale specifice cromozomului S. spontaneu (Figura 3). Dintre cele 19 sonde BAC, o sondă a fost localizată pe Sb02, Sb04 și Sb09; două sonde pe Sb07, Sb08 și Sb10; trei sonde pe Sb01 și Sb06; și patru sonde pe Sb03. Acest set de sonde a furnizat un instrument util pentru analiza comparativă a S. spontaneum și sorg (Tabelul 2).
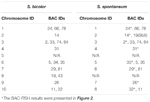
TABELUL 2. Distribuția sondelor BAC-FISH în cromozomii S. bicolor și S. spontaneum.
Construcția unei biblioteci BAC specifice pentru cromozomii S. spontaneum
Sb01 și Sb02 sunt cei mai mari doi cromozomi de sorg. Pentru a detecta sinteza genomului dintre sorg și S. spontaneum, au fost selectate 10 clone BAC specifice cromozomilor din grupurile I, II și III. Dintre aceste 10 sonde BAC, trei sonde (ID clonă BAC: 24, 66, 78) au fost omoloage cu Sb01 și șapte sonde (ID clonă BAC: 14, 18, 20, 69, 71, 73, 77) au corespuns Sb02. Am folosit detectarea cu două culori a FISH pentru perechile BAC etichetate complementar și sondele pentru cartarea relației citogenetice a lanțului pentru clonele BAC. Procedând astfel, am construit harta citogenetică a S. spontaneum pe baza celor 10 sonde BAC.
Pentru trei sonde corespunzătoare Sb01, analiza FISH a S. spontaneum (SES208) a arătat că BAC-24 și BAC -78 sonde au fost localizate în principal pe regiunea distală a cromozomului 1 S. spontaneum și BAC-66 a fost cartografiat în imediata apropiere a regiunii centromerice (Figura 4). BAC-24 și BAC-66 localizate la același cromozom (S. spontaneum cromozom 1); simultan, în timp ce sonda 66 și sonda 78, de asemenea, situate pe același cromozom, demonstrând că cele trei sonde BAC distribuite la aceiași cromozomi în S. spontaneum sugerând astfel sintenie între S. spontaneum și sorg pentru cromozomul 1. Rezultatele FISH au arătat că cele șapte sonde BAC aliniat la Sb02; numai sondele 69, 71, 73 și 77 din cele șapte BAC au generat semnale intense pe un cromozom S. spontaneu (SsChr2) (Figura 5); în timp ce sondele 14, 18 și 20 au fost nedetectabile pe cromozomul S. spontaneum 2, dar au fost observate pe alți cromozomi S. spontaneu. Aceste rezultate au demonstrat rearanjările cromozomiale apărute în Sb02 corespunzător între S. spontaneum și sorg.
Mai mult, secvența BAC-2 a împărtășit o asemănare mare (97%) cu un fragment de pe cromozomul 3 al sorg. Analiza FISH cu dublă culoare a arătat că atât ADNc BAC-2, cât și 45s au fost mapate la aceeași locație a cromozomului S. spontaneum (Figura 6).
Rearanjarea cromozomului pe Sb02 între sorg și S. spontaneum
Pentru a investiga în continuare rearanjarea cromozomială pe Sb02 între sorg și S. spontaneum, cromozom -sondele specifice corespunzătoare Sb02 (BAC-14, BAC-18 și BAC-20) și corespunzătoare Sb08 (BAC-19 și BAC-43) au fost utilizate pentru analiza FISH. Rezultatele au arătat că BAC-14 și BAC-19 au fost mapate la diferite brațe ale aceluiași cromozom S. spontaneum, indicând faptul că rearanjările cromozomiale au avut loc între cromozomii 2 și 8 în S. spontaneum și sorg (Figura 7).
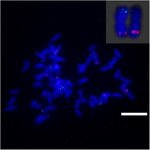
FIGURA 7. Reamenajarea cromozomului în trestie de zahăr și sorg, după cum a arătat FISH. Sonde BAC-14 și BAC-19 marcate cu biotină (verde) și digoxigenină (roșie). Scale bars = 10 μm.
Discuție
Saccharum este un gen complex caracterizat prin niveluri ridicate de poliploidie, cu cromozomi mici (Ha și colab., 1999; Dhont, 2005). Distingerea cromozomilor individuali Saccharum pe baza morfologiei lor este foarte provocatoare. Profitând de secvențe care sunt extrem de acoperite de biblioteca BAC a S. spontaneum, am reușit să analizăm potențialul număr de copii reduse ale clonelor BAC din S. spontaneum folosind ca referință genomul sorgului. Ca specie poliploidă înaltă, S. spontaneum poate conține mai multe rearanjări ale genomului decât diploizi.Diferențele genomilor dintre S. spontaneum și sorg pot fi cauza inexactitudinii în predicțiile secvențelor BAC cu număr mic de copii din genomul S. spontaneum. În acest studiu, am obținut inițial 114 secvențe potențiale de BAC cu o singură copie pe baza genomului sorgului. Cu toate acestea, doar 49 au putut fi identificate în bibliotecile parțiale BAC și 27 s-au dovedit a fi sonde BAC-FISH specifice cromozomilor. Există cel puțin două motive pentru a explica aceste rezultate. În primul rând, cele 114 secvențe BAC au fost fragmente parțiale ale inserției BAC datorită limitărilor asamblării secvenței NGS și pot conține secvențe repetate în celelalte regiuni care nu au informații de secvență. Aceste secvențe de repetare potențial descoperite ar putea induce în eroare detectarea secvenței de copiere redusă prin compararea secvenței. În al doilea rând, poliploidizarea recentă în S. spontaneum poate provoca variația secvențelor repetate între S. spontaneum și sorg și, astfel, poate duce la semnalul FISH nespecific în S. spontaneum. Într-un studiu anterior, secvențierea a 20 BAC în hibrizii trestiei de zahăr a generat secvențe contig de 1,45 Mb, secvențele aliniate cu genomul sorgului s-au întins pe 0,99 Mb, ceea ce a reprezentat aproximativ jumătate din secvențele BAC ale trestiei de zahăr (Wang și colab., 2010). Evident, ștergerile / inserțiile au existat între sorg și Saccharum, iar utilizarea genomului de sorg ca referință nu poate înlocui caracteristicile unice ale genomului Saccharum și, prin urmare, poate provoca rezultate FISH neprevăzute.
Unele diapozitive FISH au avut puternice zgomot de fond, care se poate datora unor regiuni mici de secvențe repetate (cum ar fi secvențe SSR) în clona BAC, care în consecință produc interferențe în timpul hibridizării. Genomul sorgului are un conținut de repetare de aproximativ 61% (Paterson și colab., 2009), în timp ce aproximativ jumătate din genom este compus din secvențe repetate în hibrizii Saccarrum (De și colab., 2014). În aceste genome bogate în repetări, este dificil să se dezvolte sonde FISH specifice semnalului, care disting cromozomii citogenetic cu morfologii similare. CISS cu ADN blocant C ° t-100 poate exclude în mod eficient secvențe repetate. În acest studiu, blocarea ADN-ului C ° t-100 a fost utilizată pentru analiza FISH a sondelor BAC din grupele II și III (Figurile 2D-F), care s-au verificat a fi sondele BAC specifice cromozomului, în timp ce sondele FISH ale BAC în grupurile IV și V a produs o puternică interferență de fond (figurile 2D-F). Este evident că există variații ale secvențelor repetate între sondele BAC examinate. Prin urmare, optimizarea experimentelor ar fi necesară pentru analiza BAC-FISH în Saccharum. Șase până la șapte site-uri au fost afișate în grupa II, care ar putea fi cauzată de variațiile omoloage ale structurii cromozomilor, cum ar fi ștergerea fragmentului într-unul sau doi cromozomi omologi. În grâul hexaploid, BAC 676D4 s-a hibridizat mai puternic cu cromozomii genomului A decât cu cromozomii genomului B și D (Zhang și colab., 2004). Aceste nedetectabile de unu până la două semnale în cromozomii omologi ar putea fi, de asemenea, cauzate de problemele tehnice ale BAC-FISH pentru atât de mulți cromozomi cu dimensiuni mici.
În acest studiu, am identificat 27 de BAC-cromozomi specifici Sonde FISH care corespund cu 9 din cei 10 cromozomi de sorg (toate cu excepția Sb05). Într-un studiu al hărții genetice derivat dintr-o încrucișare între S. officinarum și soiul de trestie de zahăr, Sb05 a fost fuzionat cu Sb06 în trestia de zahăr (Aitken și colab., 2014). În studiul nostru, o hartă genetică bazată pe populația F1 de S. spontaneum a dezvăluit că Sb05 a fost împărțit în două segmente, iar cele două segmente au fost îmbinate cu Sb06 și respectiv Sb07 (Zhang și Ming, date nepublicate). Recent, harta genetică a unui membru al Andropogoneae, Miscanthus sinensis, a demonstrat că Sb05 are o colinearitate slabă cu grupul de legătură corespunzător din M. sinensis (Ma și colab., 2012). În mod similar, cei doi cromozomi ancestrali de porumb ortologi Sb05 păstrează cel mai mic număr de ortologi sintetici ai genelor de sorg (Schubert și Lysak, 2011). Prin urmare, absența BAC-FISH corespunzătoare Sb05 poate indica fuziunea cromozomială în S. spontaneum.
Sorgul și S. spontaneum au divergut în urmă cu 12 milioane de ani (MYA), numărul cromozomilor de bază a fost redus de la x = 10 la x = 8. În Aitken și colab. (2014), HG2 (grupul omolog 2) al trestiei de zahăr aliniat la Sb05 și Sb06 și HG8 (grupul omolog 8) la Sb02 și Sb08, oferind dovezi pentru evenimentul de bază de reducere a cromozomilor (Aitken și colab., 2014). În acest studiu, am observat că cromozomii de sorg Sb02 și Sb08 au avut rearanjamente intercromozomiale în S. spontaneum, după cum a demonstrat dovezile că BAC-14 s-a aliniat la Sb02 și BAC-19 s-a aliniat la Sb08 (Figura 7). Studiul nostru a furnizat primele dovezi fizice și citogenetice pentru rearanjarea inter-cromozomială a sorgului în S. spontaneum. Datele nepublicate din grupul nostru au arătat, de asemenea, că Sb08 este împărțit în două segmente și au fost îmbinate cu Sb02 și Sb09 în S. spontaneum, respectiv (Zhang și Ming, date nepublicate).Sondele corespunzătoare Sb09, Sb08, Sb05 și Sb06 ar putea fi utilizate în continuare pentru investigarea evenimentelor inter-cromozomiale dintre sorg și S. spontaneum. S. spontaneum are o gamă largă de niveluri de ploidie (2n = 40-128) (Irvine, 1999). Aceste sonde BAC ar putea fi utilizate pentru a confirma rearanjarea inter-cromozomială a S. spontaneum cu diferitele niveluri de poliploidie.
Datorită numărului diferit de cromozomi de bază între sorg (x = 10) și S. spontaneum (x = 8), cromozomii de sorg nu au fost o corespondență unu-la-unu cu S. spontaneum. După cum am menționat anterior studiul nostru de cartografiere genetică, Sb08 a fost împărțit în două segmente care au fuzionat cu segmente de Sb02 și segmente de Sb09 în S. spontaneum; Sb05 a fost împărțit în două segmente care au fuzionat cu segmente de Sb06 și segmente de Sb07 S. spontaneum. Prin urmare, sondele corespunzătoare Sb08 și Sb05 nu au fost specifice cromozomilor S. spontaneum unici, în timp ce celelalte sonde corespunzătoare celorlalte opt cromozomi sorg pot fi folosite ca sonde citogenetice BAC-FISH specifice cromozomului pentru S. spontaneum. Astfel, sondele BAC testate s-au bazat pe cei opt cromozomi de sorg (Tabelul 2) au fost suficiente pentru identificarea cromozomilor utilizând BAC-FISH. Un cromozom satelit a fost găsit într-un cromozom S. spontaneu (cromozomul 3) (Ha și colab., 1999). Anterior, FISH-ul simultan a dezvăluit că semnalele ADNr 45S erau localizate pe constricțiile secundare ale cromozomilor satelit din cromozomul 3 din clona S. spontaneum derivată din cultura anterioară (AP85-361) (Ha și colab., 1999). În acest studiu, BAC-2, care corespunde Sb03 și 45s rADN, au fost mapate în aceeași locație a cromozomului S. spontaneum, susținând astfel în continuare descoperirile anterioare (Ha și colab., 1999). Rezultatele noastre au furnizat, de asemenea, dovezi directe că cromozomul 3 din S. spontaneum numit în studiul anterior este omolog cu Sb03.
Concluzie
În acest studiu, am dezvoltat BACs specifici pentru cromozomi ai S spontaneul ca un pas spre dezvoltarea unei metode simple și reproductibile pentru identificarea cromozomilor utilizând markeri citogenetici BAC-FISH, confirmând fezabilitatea izolării BAC-urilor specifice cromozomului pe baza genomului sorgului pentru a construi o hartă fizică a trestiei de zahăr. De asemenea, oferim primele dovezi citogenetice de rearanjare inter-cromozomială între sorg și S. spontaneum. Înființarea sistemului tehnologic BAC-FISH al trestiei de zahăr oferă noi oportunități și mijloace pentru cercetarea citogenetică moleculară a trestiei de zahăr, inclusiv analiza cariotipului, localizarea genelor și construirea hărții fizice. Aceste rezultate sunt esențiale pentru asamblarea genomului S. spontaneum necesar pentru secvențierea întregului genom.
Contribuțiile autorului
GD, JS și JZ au conceput studiul și au conceput experimentele. GD, JS, QZ, JW, QY, RM, KW și JZ au efectuat experimentele și au analizat datele. GD și JZ au scris manuscrisul. Toți autorii au citit și au aprobat lucrarea finală.
Finanțare
Acest proiect a fost sprijinit de subvenții din programul 863 (2013AA102604), NSFC (31201260), Programul pentru talentele excelente din secolul nou Universitatea din provincia Fujian și finanțarea de la Universitatea agricolă și forestieră din Fujian.
Declarația privind conflictul de interese
Autorii declară că cercetarea a fost efectuată în absența oricărei relații comerciale sau financiare care ar putea fi interpretat ca un potențial conflict de interese.
Mulțumiri
Mulțumim dr. Xintan Zhang pentru asamblarea secvențelor BAC și dr. Irene Lavagi pentru editarea limbii engleze.
Material suplimentar
Dhont, A. (2005). Descoperirea structurii genomului poliploidelor folosind FISH și GISH; exemple de trestie de zahăr și banane. Citogenet. Genom Res. 109, 27–33. doi: 10.1159 / 000082378
PubMed Abstract | Text integral CrossRef | Google Scholar
Piperidis, G., D’Hont, A. și Hogarth, D. M. (2001). „Analiza compoziției cromozomiale a diferiților hibrizi Saccharum interspecifici prin genomică in situ (hibridizare) (GISH)”, în Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD: ASSCT), 565-566.
Google Scholar
