소개
설탕 수수 (Saccharum spp., Poaceae)는 전 세계 열대 및 아열대 지역에서 재배되는 연간 또는 다년생 작물입니다. 사탕 수수는 자당 생산에 중요한 작물이며 전 세계 설탕 생산량의 70 %를 차지합니다. 또한 C4 식물로서 사탕 수수는 태양 에너지를 화학 에너지로 효율적으로 변환 할 수 있으므로 에탄올 생산을위한 이상적인 바이오 연료 작물입니다 (Lam et al., 2009; Santchurn et al., 2014). Saccharum 속은 S. spontaneum, S. robubumum, S. officinarum, S. barberi, S. sinense, S. edule의 6 종이 있으며, 그중 S. spontaneum은 기본 염색체 세트가 x 인 야생 종으로 간주됩니다. = 8. S. spontaneum은 또한 현대 사탕 수수 품종 개발에 기여하는 두 가지 주요 Saccharum 종 중 하나입니다 (Dhont et al., 1998; Ha et al., 1999). S. spontaneum은 환경 적응성이 강하고 질병 및 가뭄 저항성에 중요한 유전 적 특성을 포함하고있어 현대 품종 잡종의 스트레스 내성에 기여합니다 (Grivet et al., 2004). S. spontaneum은 지리적 분포가 가장 넓고 배수성 수준은 2n = 5x = 40 ~ 2n = 16x = 128입니다 (Panje and Babu, 1960).
사탕 수수 염색체는 비슷한 형태를 가지고 있으며 응축 중기 단계에서 1-6 μm 크기 (Ha et al., 1999; Dhont, 2005); 따라서 전통적인 세포 유전 학적 방법을 기반으로 다른 염색체를 식별하는 것은 매우 어렵습니다. 더욱이,자가 배수체로서, 배수 체화, 복제, 결실 및 재조합으로 인한 염색체 구조적 변화는 매우 흔합니다 (Piperidis et al., 2010). S. spontaneum에서는 유전 적 연결지도가 개발되었지만 (Silva et al., 1995), 세포 학적 유전지도의 개발은 다른 풀 종보다 뒤처졌습니다. 개별 염색체를 식별하는 신뢰할 수있는 세포학 도구는 특히 현대 사탕 수수 품종의 복잡한 게놈 배경과 함께 효과적인 게놈 연구 및 생식 질 자원 활용에 사용될 수 있습니다. 염색체 특정 박테리아 인공 염색체 (BAC) 클론은 사탕 수수 게놈 연구에 매우 귀중한 자원이며 Saccharum 종의 물리적 매핑, 염색체 식별 및 마커 지원 육종, 사탕 수수 게놈 시퀀싱 및 조립 프로젝트 지원에 많은 응용 분야가 있습니다. .
FISH (Fluorescence in situ hybridization)는 분자 세포학을위한 강력한 도구입니다 (Jiang and Gill, 2006). Saccharum spp.에서는 45S rDNA와 5S rDNA를 프로브로 사용하여 S. spontaneum, S. robertum 및 S. officinarum의 기본 염색체를 감지하여 다양한 기본 염색체를 식별합니다. S. spontaneum에서 x = 8, x = 10 in S. rovenum 및 S. officinarum (Dhont et al., 1998; Ha et al., 1999). FISH에 따르면 현대 사탕 수수 품종은 S. officinarum에서 유래 한 염색체의 70 ~ 80 %, S. spontaneum에서 유래 한 염색체의 10 ~ 23 %를 포함하는 것으로 나타 났으며, 5 ~ 17 %는 S. spontaneum 및 S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). 더욱이, 염색체 제거, 재조합 및 전좌 이벤트는 FISH를 사용하여 Saccharum과 Erianthus arundinaceus 간의 혼성화에서 파생 된 일부 선조에서 발견되었습니다 (Dhont et al., 1995; Huang et al., 2015).
Sorghum 염색체 재조합 사건의 빈도가 낮은 작은 2 배체 게놈 (730Mbp)을 가지고 있습니다. 수수는 사탕 수수 게놈과 높은 synteny를 공유하므로 사탕 수수 게놈과의 비교 분석을위한 이상적인 참조 식물입니다 (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ). 이 연구에서는 높은 커버리지 BAC 자원에 사용할 수있는 서열을 기반으로 FISH 염색체 식별 및 세포 유전 학적지도 구축을 위해 신뢰할 수있는 BAC 프로브를 개발했습니다. 이 연구의 목적은 (1) S. spontaneum 염색체에 대한 일련의 염색체 특이 적 BAC-FISH 프로브를 개발하고 (2) S. spontaneum과 수수 사이의 염색체 재 배열을 탐구하는 것입니다.
재료 및 방법
재료
Saccharum spontaneum SES208 (2n = 8x = 64)은이 연구에서 세포 학적 분석에 사용되었습니다. SES208 식물은 2015 년 2 월 Fujian Agricultural and Forestry University (중국 푸 저우) 캠퍼스의 밭에서 재배되었으며 정기적 인 사탕 수수 성장 조건에서 유지되었습니다.
반수체 게놈에서 BAC 라이브러리를 구축했습니다. S. spontaneum AP85-441 (4x = 32)의 S. spontaneum SES208 (2n = 8x = 64)에서 꽃밥 in vitro 배양을 통해 파생되었습니다. AP85-441의 게놈 크기는 약 3.2Gbp입니다. 라이브러리는 전체 게놈의 6 배를 포함하는 평균 삽입 크기가 100kb 인 38,400 개의 클론으로 구성되었습니다.이 BAC 라이브러리는 S. spontaneum (Zhang et al., 2016; Chen et al., 2017)에서 수 크로스 수송 체와 프럭 토키나 제 유전자 패밀리를 식별하는 데 사용되었습니다.
BAC 라이브러리 스크리닝
AP85-441 라이브러리의 35,156 개의 BAC 클론이 701 개의 라이브러리로 풀링되었습니다. 각 라이브러리에는 평균 50 개의 BAC 클론이 포함되어 있습니다. DNA 라이브러리는 제조업체의 프로토콜에 따라 PhasePrepTM BAC DNA 키트 (미국 시그마)로 준비되었습니다. BAC DNA 라이브러리는 PE250 모델과 함께 Illumina HiSeq 2500 플랫폼을 사용하여 시퀀싱되었습니다. 총 686 개의 라이브러리 (시퀀싱에 실패한 18 개의 라이브러리)가 시퀀싱되었으며 Trimmomatic 버전 0.36을 사용하여 트리밍 후 267.5Gb의 정리 된 데이터가 생성되었습니다 (Bankevich et al., 2012). 각 BAC 풀의 데이터는 기본 매개 변수와 함께 SPAdes 버전 3.09를 사용하여 어셈블되었습니다. 총 2,611,145 개의 contig가 7.38kbp의 contig N50 (Zhang and Ming, 미공개 데이터)으로 어셈블되었습니다.
AP85-441 BAC 라이브러리 서열은 E- 값 1e로 수수 게놈에 대해 BLAST 검색되었습니다. -4. 수수 게놈에서 단일 BLAST 히트를 가진 서열을 후보 FISH 프로브로 선택했습니다. BAC 라이브러리에서 낮은 카피 수 BAC 클론을 식별하기 위해 Primer 5.0 소프트웨어를 사용하여 서열 특이 적 프라이머를 설계하여 BAC 풀이 PCR에 의해 특별히 스크리닝 될 수 있도록했습니다. 프라이머 길이는 18–25 bp, Tm 값은 55–65 ° C, 40–60 % 사이토 신과 구아닌의 뉴클레오티드 조성이 선택되었습니다. 2 단계 PCR 방법을 사용하여 BAC 라이브러리 (Asakawa et al., 1997; Crooijmans et al., 2000)의 3D 차원 풀을 일부 수정하여 스크리닝했습니다. 384- 웰 플레이트의 각 클론을 384- 웰 배양 플레이트에서 37 ° C에서 밤새 80μl 리소 제니 브로스 + 34mg / ml 클로람페니콜에서 배양 하였다. 총 3072 개의 BAC 클론이있는 총 8 개의 384 웰 플레이트를 배양했습니다. 각 384- 웰 플레이트에 대해 먼저 각 개별 클론의 동일한 양의 배양 물을 혼합하여 각 384 웰 플레이트에 대해 16 개의 행 풀 (A ~ P 행)과 24 개의 열 풀 (1 ~ 24 열)을 구성한 다음 16 개의 행 풀과 24 개의 열 풀, 각 행 및 열 풀의 동일한 볼륨이 하나의 플레이트 풀 또는 수퍼 풀로 사용됩니다. 이 연구에서 우리는 8 개의 384 웰 플레이트를 스크리닝하는 데 중점을 두었습니다. 따라서 8 개의 플레이트에 대해 개별적으로 8 개의 수퍼 풀이 준비되었습니다. 8 개의 플레이트에서 양성 384- 웰 플레이트를 확인한 후 16 개의 혼합물 중 양성 열을 선별했습니다. 마지막으로 24 개의 컬럼 혼합물 중에서 PCR 양성 컬럼을 선택했습니다. 24 개의 PCR 반응을 수행하여 8 개의 384 웰 플레이트에서 양성 클론을 확인했습니다.
PCR 증폭은 이전에 설명한대로 수행되었습니다 (Bouzidi et al., 2006). 각 반응에는 rTaq 프리믹스 7.5μl, 각 프라이머 (10μM) 0.6μl, 박테리아 액체 템플릿 1.0μl가 포함되어 최종 부피가 20μl가되었습니다. 95 ° C에서 5 분 동안 초기 변성 후, 30 초 동안 95 ° C, 30 초 동안 55 ° C, 1 분 동안 72 ° C의 35 사이클을 수행했습니다. PCR 산물은 1.5 % 아가 로스 겔에서 분석되었습니다.
메타 페이즈 염색체 준비
염색체 준비는 이전에 설명 된대로 약간 수정하여 수행했습니다 (Lou et al., 2010). 간단히 말해, S. spontaneum 식물은 뿌리 끝 수확을 위해 온실 조건에서 재배되었습니다. 약 1-2cm의 절제된 뿌리 끝을 실온에서 2-4 시간 동안 0.002 mol / l 8- 하이드 록시 퀴놀린으로 처리하고, 15 분 동안 물로 헹구고, 적어도 24 시간 동안 에탄올 : 아세트산 (3 : 1)에 고정했습니다. h 실온에서. 그런 다음 뿌리 끝을 37 ° C에서 1 시간 동안 효소 용액 (0.1M 시트 레이트 완충액 중 4 % 셀룰로오스 R-10 및 2 % 펙톨 리아제)에서 분해하고, 얼음 탈 이온수로 30 분 동안 세척하고 마지막으로 에탄올에서 배양했습니다. 약 30 분 동안 아세트산 (3 : 1). 슬라이드는 “불꽃 건조”방법 (Iovene et al., 2008)을 사용하여 준비했습니다.
BAC DNA 정제 및 프로브 라벨링
BAC DNA는 PhasePrepTM BAC로 추출했습니다. 제조사 매뉴얼에 따른 DNA Kit. 정제 된 BAC DNA는 희석 된 DNase I, 10x nick translation buffer, DNA Polymerase I, dNTPs, biotin- / digoxigenin-labeled dUTPs 및 BAC DNA를 포함한 표준 nick translation 반응에 의해 표지되었습니다. 15 ° C에서 1.5 시간 동안 배양 한 다음, 절단 된 제품을 1.5 % 아가 로스 겔에서 300 ~ 500bp 사이의 얼룩이 있는지 검사 한 후 얻은 프로브를 사용할 때까지 -20 ° C에서 보관했습니다.
In Situ 혼성화 및 검출
프로브 혼합 및 혼성화 : 첫째, 프로브 혼합물 (50 % 탈 이온화 포름 아미드, 2X SSC, 80ng 디 옥시 게닌 / 비오틴 표지 DNA, 10 % 덱스 트란 설페이트 , > 1μg C ° t-100)을 90 ° C 핫 블록에 5 분 동안 넣은 다음 하이브리드 자티를 조사 할 준비가 될 때까지 즉시 얼음 위에 옮겼습니다. 의 위에. 이전에 준비된 화염 건조 된 슬라이드를 70 % 탈 이온화 포름 아미드로 처리하고 80 ° C에서 1 시간 동안 열 블록에서 변성합니다.5 분, 즉시 차가운 70 % 에탄올, 90 % 에탄올, 100 % 에탄올 각각에 5 분 동안 순차적으로 침지 한 다음 벤치에서 공기 건조합니다. 마지막으로 변성 프로브를 각 슬라이드에 추가하고 24 x 32mm 커버 슬립으로 덮었습니다. 슬라이드를 37 ° C의 축축한 챔버에 밤새 두었습니다.
프로브 감지 : 커버 슬립을 제거하고 슬라이드를 2x SSC에서 5 분 동안 실온에서, 42 ° C에서 2x로 세척했습니다. 이 순서로 1x PBS에서 10 분 동안 SSC 및 실온에서 5 분 동안. 비오틴 표지 프로브 신호는 2mg / ml Alexa Fluor 488 스트렙 타비 딘으로 검출되었고 digoxygenin 표지 프로브 신호는 양의 2 % 항-디곡 시게 닌-로다 민으로 검출되었습니다. 항체 칵테일 (100μl TNB 완충제, 1μl Alexa Fluor 488 스트렙 타비 딘, 1μl 로다 민 항 -dig-sheep)을 슬라이드에 첨가하고, 24 x 32mm 커버 슬립으로 덮고, 37 ° C에서 1 시간 동안 배양했습니다. 습한 챔버, 그리고 1x PBS에서 각각 5 분 동안 3 회 실온에서 세척 하였다. 과량의 액체를 제거하고 페이드 방지 용액 Vectashield (Vector, Burlingame, CA, United States)에 4 ‘, 6-diamidino-2-phenylindole (DAPI) (Sigma, St. Louis, MO, United States)을 첨가하여 염색체. Olympus BX63 epifluorescence 현미경으로 이미지를 캡처했습니다. FISH 신호 이미지는 CellSens Dimension 소프트웨어를 사용하여 분석되었습니다.
결과
저 복사 BAC 클론 스크리닝
잠재적으로 카피 수가 적은 BAC 클론 스크리닝 S. spontaneum 게놈의 염기 서열, 우리 BLAST는 수수 게놈에 대해 염기 서열 된 AP85-441 BAC 라이브러리를 검색했습니다. 공선 성이 양호하고 서열 특이성을 나타내는 10 개의 수수 염색체에 해당하는 총 2000 개의 BAC 서열을 선별 하였다. 2000 개의 AP85-441 BAC 서열은 반복 서열을 필터링하기 위해 고반복 수수 DNA 데이터베이스 및 TIGR gramineae 데이터베이스에 대해 REPEATMASKER에 의해 마스킹되었습니다. 마지막으로, 10 개의 수수 염색체에 분포 된 114 개의 BAC가 FISH 분석을 위해 선택되었습니다 (보충 표 1). 10 개의 수수 염색체 각각에 7-16 개의 BAC가 선택되었습니다 (그림 1). 양성 BAC 클론을 스크리닝하기 위해 BAC 클론 서열 (보충 표 2)을 기반으로 114 개의 특정 프라이머를 설계하고 BAC 라이브러리에서 양성 BAC 클론 49 개를 스크리닝했습니다 (보충 표 3).
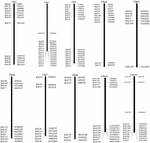
그림 1. 수수의 염색체에있는 AP85-441의 BAC 클론 114 개 분포. BAC1-114는 BAC 번호를 나타내고 오른쪽 데이터는 수수 염색체의 해당 영역에서 BAC의 물리적 위치입니다. 단위 = bp.
BAC-FISH 신호 강도 및 분포
49 개의 양성에서 분리 된 DNA 샘플 BAC 클론은 비오틴 또는 디곡시 제닌으로 표지되었습니다. 5S rDNA 및 45S rDNA 프로브를 대조군으로 사용하고 FISH 절차에 따라 S. spontaneum 체세포 중기 염색체에 혼성화했습니다. 신호 강도와 BAC 시퀀스의 반복되는 시퀀스 콘텐츠의 변화 사이의 상관 관계로 인해 C ° t-100을 경쟁자로 사용했습니다 (표 1). 강하고 꾸준한 혼성화 신호를 나타내는 BAC 클론은 추가 FISH 분석을 위해 선택되었습니다.

표 1. DNA를 차단하는 27 개의 양성 클론 분류
총 64 개의 별개의 염색체를 관찰 할 수 있습니다. S. spontaneum의 중기에서. 각 BAC-FISH는 4 개의 독립적 인 실험 (또는 슬라이드)에서 수행되었습니다. 각 슬라이드에서 체세포 중기 염색체의 최소 10 개 스프레드를 분석했습니다. 그 결과 27 개의 특정 프로브 (보충 표 4)가 혼성화 신호에 따라 5 개 그룹으로 분류 될 수 있음을 보여주었습니다 (표 1). 그룹 I (BAC-18, BAC-20, BAC-29 및 BAC-84 포함)에서 프로브는 C ° t-100 (그림 2A)을 사용하여 경쟁 현장 억제 (CISS)없이 8 개의 고유 한 사이트를 표시하여 거의 반복되지 않음을 시사합니다. 이들 BAC 서열에는 서열이 존재한다. 그룹 II (BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 및 BAC-74 포함), C ° t -100은 BAC 프로브 혼성화를위한 경쟁자로 사용되었으며,이 그룹의 핵형에서 6 ~ 7 개의 매우 뚜렷한 부위가 표시되었습니다 (그림 2B). 혼성화 결과는 상동 염색체의 염색체 구조 변화에 의해 발생할 수있는 7 개의 신호 부위를 가진 45S rDNA와 유사했습니다. 그룹 III (BAC-26, BAC-66, BAC-76 및 BAC-78 포함)에서는 C ° t-100 DNA가 경쟁자로 사용되었으며 핵형에서 8 개의 다른 부위가 관찰 될 수있어이 그룹의 BAC를 시사합니다. 클론에는 반복적 인 DNA 서열이 있습니다 (그림 2C). 그룹 IV (BAC-24 포함)에서 8 개 이상의 신호 부위가 핵형에서 관찰되었고 주변 중심 및 텔 로머 영역 (그림 2D)에 나타 났으며, 이는 이러한 BAC 프로브가 S. spontaneum에서 반복적 인 서열을 가졌음을 시사합니다.그룹 V (BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 및 BAC-81 포함)에서 프로브는 제한된 영역을 가진 모든 염색체에서 분산 된 신호를 나타냅니다. 특정 신호를 표시하는 개별 염색체의 (그림 2E). 혼성화 결과는이 그룹의 BAC 클론이 S. spontaneum에 다량의 반복 DNA를 포함하고 있음을 나타 냈는데, 이는 수수와 S. spontaneum이 분리 된 후 반복 영역에 대한 급속한 진화 적 분기로 인해 발생할 수 있습니다. 따라서 49 개의 BAC 프로브 중 그룹 I, II 및 III의 19 개 BAC 프로브 세트 (보충 표 3)는 Sb05 외에 수수 염색체 중 9 개에 해당하는 S. spontaneum 염색체에서 특정 신호를 나타 냈습니다. 다른 22 개의 프로브의 신호는 모든 염색체에 분산되었습니다. 우리는 S. spontaneum 염색체 특정 신호로 19 개의 BAC 프로브 세트를 선택했습니다 (그림 3). 19 개의 BAC 프로브 중 하나는 Sb02, Sb04 및 Sb09에 있습니다. Sb07, Sb08 및 Sb10에 대한 두 개의 프로브; Sb01 및 Sb06에 대한 세 개의 프로브; Sb03에 4 개의 프로브가 있습니다. 이 프로브 세트는 S. spontaneum과 수수를 비교 분석하는 데 유용한 도구를 제공했습니다 (표 2).
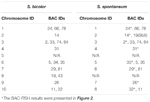
표 2. S. bicolor 및 S. spontaneum 염색체에서 BAC-FISH 프로브 분포.
S. spontaneum 염색체 전용 BAC 라이브러리 구축
Sb01과 Sb02는 두 개의 가장 큰 수수 염색체입니다. 수수와 S. spontaneum 사이의 게놈 합성을 검출하기 위해, 그룹 I, II 및 III에서 10 개의 염색체 특이 적 BAC 클론을 선택했습니다. 이 10 개의 BAC 프로브 중 3 개의 프로브 (BAC 클론 ID : 24, 66, 78)는 Sb01에 상동했고 7 개의 프로브 (BAC 클론 ID : 14, 18, 20, 69, 71, 73, 77)는 Sb02에 해당합니다. 우리는 보완 적으로 표지 된 BAC 쌍에 대한 FISH의 이중 색상 검출과 BAC 클론에 대한 사슬 세포 유전 학적 관계를 매핑하기위한 프로브를 사용했습니다. 이를 통해 10 개의 BAC 프로브를 기반으로 S. spontaneum의 세포 유전 학적지도를 구축했습니다.
Sb01에 해당하는 3 개의 프로브에 대해 S. spontaneum (SES208)의 FISH 분석 결과 BAC-24 및 BAC -78 프로브는 S. spontaneum 염색체 1의 원위 영역에 주로 위치했으며 BAC-66은 중심체 영역에 매우 근접하게 매핑되었습니다 (그림 4). 동일한 염색체에 위치한 BAC-24 및 BAC-66 (S. spontaneum 염색체 1); 동시에 프로브 66과 프로브 78도 동일한 염색체에 위치하여 세 개의 BAC 프로브가 S. spontaneum의 동일한 염색체에 분포되어 있음을 보여줌으로써 S. spontaneum과 수수 사이의 동기화를 시사합니다. FISH 결과는 7 개의 BAC 프로브가 있음을 나타냅니다. Sb02에 정렬 됨; 7 개의 BAC 중 69, 71, 73, 77 번 프로브 만이 S. spontaneum 염색체 (SsChr2) 하나에서 강력한 신호를 생성했습니다 (그림 5). 프로브 14, 18, 20은 S. spontaneum 염색체 2에서는 검출되지 않았지만 다른 S. spontaneum 염색체에서는 관찰되었습니다. 이 결과는 S. spontaneum과 수수 사이의 해당 Sb02에서 염색체 재 배열이 발생했음을 보여줍니다.
또한 BAC-2의 서열은 염색체 3의 단편과 높은 유사성 (97 %)을 공유했습니다. 수수. 이중 색상 FISH 분석은 BAC-2 및 45s rDNA가 S. spontaneum 염색체의 동일한 위치에 매핑 된 것으로 나타났습니다 (그림 6).
수수와 S. spontaneum 간의 Sb02 염색체 재 배열
수수와 S. spontaneum 간의 Sb02 염색체 재 배열을 추가로 조사하려면 염색체 Sb02 (BAC-14, BAC-18 및 BAC-20)에 해당하고 Sb08 (BAC-19 및 BAC-43)에 해당하는 특이 적 프로브를 FISH 분석에 사용했습니다. 결과는 BAC-14와 BAC-19가 동일한 S. spontaneum 염색체의 다른 암에 매핑되어 S. spontaneum과 수수에서 염색체 2와 8 사이에 염색체 재 배열이 발생했음을 나타냅니다 (그림 7).
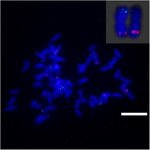
그림 7. FISH에 의해 밝혀진 사탕 수수와 수수의 염색체 재 배열. BAC-14 및 BAC-19 프로브는 비오틴 (녹색) 및 디곡 시게 닌 (빨간색)으로 표시됩니다. 축척 막대 = 10 μm.
토론
Saccharum은 높은 배수성 수준을 특징으로하는 복잡한 속입니다. 작은 염색체로 (Ha et al., 1999; Dhont, 2005). 형태에 따라 개별 Saccharum 염색체를 구별하는 것은 매우 어렵습니다. S. spontaneum의 BAC 라이브러리에 의해 고도로 커버되는 서열을 활용하여 수수 게놈을 참조로 사용하여 S. spontaneum에서 잠재적 인 낮은 카피 수 BAC 클론을 스크리닝 할 수있었습니다. 고 배수체 종으로서 S. spontaneum은 이배체보다 더 많은 게놈 재 배열을 포함 할 수 있습니다.S. spontaneum과 수수의 게놈 차이는 S. spontaneum 게놈에서 낮은 카피 수 BAC 서열의 예측에 부정확 한 원인이 될 수 있습니다. 이 연구에서 우리는 처음에 수수 게놈을 기반으로 한 단일 복사본으로 114 개의 잠재적 BAC 서열을 얻었습니다. 그러나 부분 BAC 라이브러리에서 49 개만이 식별 될 수 있었고 27 개는 염색체 특이 적 BAC-FISH 프로브 인 것으로 밝혀졌습니다. 이러한 결과를 설명하는 데는 최소한 두 가지 이유가 있습니다. 첫째, 114 개의 BAC 서열은 NGS 서열 조립의 한계로 인해 BAC 삽입의 일부 단편이었으며 서열 정보가없는 다른 영역에 반복 서열을 포함 할 수있다. 잠재적으로 발견되지 않은 이러한 반복 서열은 서열 비교를 통해 낮은 카피 서열의 검출을 오도 할 수 있습니다. 둘째, S. spontaneum의 최근 polyploidization은 S. spontaneum과 수수 사이의 반복 서열의 변화를 유발하여 S. spontaneum에서 비특이적 FISH 신호를 초래할 수 있습니다. 이전 연구에서 사탕 수수 잡종에서 20 개의 BAC를 시퀀싱 한 결과 1.45Mb contig 서열이 생성되었으며, 수수 게놈과 정렬 된 서열은 0.99Mb에 걸쳐 사탕 수수 BAC 서열의 약 절반을 차지했습니다 (Wang et al., 2010). 당연히 수수와 사카 룸 사이에 결실 / 삽입이 존재했고, 수수 게놈을 참조로 사용하는 것은 사카 룸 게놈의 고유 한 특징을 대체 할 수 없기 때문에 예상치 못한 FISH 결과를 초래할 수 있습니다.
일부 FISH 슬라이드는 강력했습니다. BAC 클론에서 반복 시퀀스 (예 : SSR 시퀀스)의 작은 영역으로 인해 발생할 수있는 배경 잡음으로, 결과적으로 혼성화 동안 간섭이 생성됩니다. 수수 게놈은 약 61 %의 반복 함량을 갖는 반면 (Paterson et al., 2009), 게놈의 약 절반은 Saccarrum 잡종의 반복 서열로 구성됩니다 (De et al., 2014). 이러한 반복이 풍부한 게놈에서는 유사한 형태로 염색체를 세포 유 전적으로 구별하는 신호 특이 적 FISH 프로브를 개발하기가 어렵습니다. DNA C ° t-100을 차단하는 CISS는 반복 서열을 효율적으로 배제 할 수 있습니다. 이 연구에서 블로킹 DNA C ° t-100은 그룹 II 및 III (그림 2D–F)의 BAC 프로브에 대한 FISH 분석에 사용되었으며, 염색체 특이 적 BAC 프로브 인 반면 BAC 프로브의 FISH는 그룹 IV 및 V에서는 강한 배경 간섭이 발생했습니다 (그림 2D–F). 검사 된 BAC 프로브간에 반복 시퀀스의 변형이 있음이 분명합니다. 따라서 Saccharum에서 BAC-FISH 분석을 위해서는 실험 최적화가 필요합니다. 그룹 II에서는 6 ~ 7 개의 부위가 나타 났는데, 이는 1 개 또는 2 개의 상동 염색체의 단편 결실과 같은 상동 염색체 구조 변이로 인해 발생할 수 있습니다. 6 배체 밀에서 BAC 676D4는 B- 및 D- 게놈 염색체보다 A- 게놈 염색체에 더 강하게 혼성화되었습니다 (Zhang et al., 2004). 상동 염색체에서 1 ~ 2 개의 신호를 감지 할 수없는 것은 크기가 작은 많은 염색체에 대한 BAC-FISH의 기술적 문제로 인해 발생할 수도 있습니다.
이 연구에서 27 개의 염색체 특이 BAC-를 확인했습니다. 10 개의 수수 염색체 중 9 개에 해당하는 FISH 프로브 (Sb05를 제외한 모두). S. officinarum과 사탕 수수 품종 사이의 교배에서 파생 된 유전자지도에 대한 연구에서 Sb05는 사탕 수수에서 Sb06과 병합되었습니다 (Aitken et al., 2014). 우리의 연구에서 S. spontaneum의 F1 집단을 기반으로 한 유전자지도는 Sb05가 두 개의 세그먼트로 나뉘었고 두 세그먼트가 각각 Sb06 및 Sb07과 병합 된 것으로 나타났습니다 (Zhang 및 Ming, 미공개 데이터). 최근 Andropogoneae의 일원 인 Miscanthus sinensis의 유전자지도는 Sb05가 M. sinensis의 해당 연결 그룹과 공선 성이 좋지 않음을 보여주었습니다 (Ma et al., 2012). 유사하게, Sb05에 직교하는 두 개의 조상 옥수수 염색체는 수수 유전자에 대한 가장 적은 수의 동조성 직교를 유지합니다 (Schubert and Lysak, 2011). 따라서 Sb05에 해당하는 BAC-FISH의 부재는 S. spontaneum에서 염색체 융합을 의미 할 수 있습니다.
수수와 S. spontaneum은 1,200 만년 전에 분기 된 (MYA), 기본 염색체 수는 x에서 감소했습니다. = 10에서 x = 8. In Aitken et al. (2014), Sb05 및 Sb06에 정렬 된 사탕 수수의 HG2 (동종 그룹 2), Sb02 및 Sb08에 정렬 된 HG8 (상동 그룹 8)은 기본 염색체 감소 사건에 대한 증거를 제공합니다 (Aitken et al., 2014). 이 연구에서 우리는 BAC-14가 Sb02에 정렬되고 BAC-19가 Sb08에 정렬되었다는 증거에 의해 입증 된 바와 같이 S. spontaneum에서 수수 염색체 Sb02 및 Sb08이 염색체 간 재 배열을 가짐을 관찰했습니다 (그림 7). 우리의 연구는 S. spontaneum에서 수수 염색체 간 재배 열에 대한 최초의 물리적 및 세포 유전 학적 증거를 제공했습니다. 우리 그룹의 미공개 데이터는 또한 Sb08이 두 개의 세그먼트로 나뉘어 S. spontaneum의 Sb02 및 Sb09와 각각 병합 된 것으로 나타났습니다 (Zhang 및 Ming, 미공개 데이터).Sb09, Sb08, Sb05 및 Sb06에 해당하는 프로브는 수수와 S. spontaneum 사이의 염색체 간 사건을 조사하는 데 추가로 사용될 수 있습니다. S. spontaneum은 광범위한 배수성 수준 (2n = 40–128)을 가지고 있습니다 (Irvine, 1999). 이 BAC 프로브는 S. spontaneum의 염색체 간 재 배열을 다양한 배수 수준으로 확인할 수 있습니다.
수수 (x = 10)와 S. spontaneum (x = 8), 수수 염색체는 S. spontaneum과 일대일 대응이 아니었다. 앞서 언급 한 유전자 매핑 연구에 따르면 Sb08은 S. spontaneum의 Sb02 및 Sb09 세그먼트와 병합 된 두 개의 세그먼트로 나뉩니다. Sb05는 Sb06의 세그먼트와 Sb07 S. spontaneum의 세그먼트와 병합 된 두 개의 세그먼트로 나뉩니다. 따라서 Sb08 및 Sb05에 해당하는 프로브는 단일 S. spontaneum 염색체에 특이 적이 지 않은 반면, 다른 8 개의 수수 염색체에 해당하는 다른 프로브는 S. spontaneum에 대한 염색체 특이 적 세포 유전 학적 BAC-FISH 프로브로 사용될 수 있습니다. 따라서 테스트 된 BAC 프로브는 8 개의 수수 염색체를 기반으로했으며 (표 2) BAC-FISH를 사용하여 염색체 식별에 충분했습니다. S. spontaneum 염색체 (염색체 3)에서 위성 염색체가 발견되었습니다 (Ha et al., 1999). 이전에 동시 FISH는 45S rDNA의 신호가 꽃밥 배양 유래 S. spontaneum 클론 (AP85-361) (Ha et al., 1999)에서 3 번 염색체 내 위성 염색체의 2 차 수축에 위치한다는 사실을 밝혀 냈습니다. 이 연구에서 Sb03 및 45s rDNA에 해당하는 BAC-2는 S. spontaneum 염색체의 동일한 위치에 매핑되어 이전 결과를 더욱 뒷받침합니다 (Ha et al., 1999). 우리의 결과는 또한 이전 연구에서 명명 된 S. spontaneum의 염색체 3이 Sb03과 동종이라는 직접적인 증거를 제공했습니다.
결론
이 연구에서 우리는 S의 염색체 특이 적 BAC를 개발했습니다. . spontaneum은 BAC-FISH 세포 유전 학적 마커를 사용하여 염색체 식별을위한 간단하고 재현 가능한 방법을 개발하기위한 단계로 사탕 수수의 물리적지도를 구축하기 위해 수수 게놈을 기반으로 염색체 특정 BAC를 분리하는 가능성을 확인합니다. 우리는 또한 수수와 S. spontaneum 사이의 염색체 간 재배 열에 대한 최초의 세포 유전 학적 증거를 제공합니다. 사탕 수수 BAC-FISH 기술 시스템의 확립은 핵형 분석, 유전자 위치 파악 및 물리적지도 구축을 포함하여 사탕 수수 분자 세포 유전학 연구를위한 새로운 기회와 수단을 제공합니다. 이러한 결과는 전체 게놈 시퀀싱에 필요한 S. spontaneum 게놈 조립에 필수적입니다.
저자 기고
GD, JS 및 JZ가 연구를 구상하고 실험을 설계했습니다. GD, JS, QZ, JW, QY, RM, KW, JZ가 실험을 수행하고 데이터를 분석했습니다. GD와 JZ가 원고를 썼습니다. 모든 저자가 최종 논문을 읽고 승인했습니다.
기금
이 프로젝트는 863 프로그램 (2013AA102604), NSFC (31201260), Program for New Century Excellent Talents in Fujian Province University 및 Fujian Agriculture and Forestry University의 자금 지원.
이해 상충 성명서
저자는 연구가 상업적 또는 재정적 관계없이 수행되었다고 선언합니다. 잠재적 인 이해 상충으로 해석 될 수 있습니다.
감사합니다
BAC 시퀀스를 조립 해 주신 Xintan Zhang 박사와 영어를 편집 한 Irene Lavagi 박사에게 감사드립니다.
보충 자료
Dhont, A. (2005). FISH 및 GISH를 사용하여 배수체의 게놈 구조 풀기; 사탕 수수와 바나나의 예. Cytogenet. Genome Res. 109, 27–33. doi : 10.1159 / 000082378
PubMed 요약 | CrossRef 전체 텍스트 | Google 학술 검색
Piperidis, G., D’ Hont, A. 및 Hogarth, D. M. (2001). “Genomic in situ (hybridisation) (GISH)에 의한 다양한 Saccharum interspecific hybrids의 염색체 조성 분석”, Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD : ASSCT), 565–566)
Google 학술 검색
