Introducción
La caña de azúcar (Saccharum spp., Poaceae) es un cultivo anual o perenne que se cultiva en regiones tropicales y subtropicales de todo el mundo. La caña de azúcar es un cultivo importante para la producción de sacarosa y representa el 70% de la producción mundial de azúcar. Además, como planta C4, la caña de azúcar puede convertir eficientemente la energía solar en energía química y, por lo tanto, es un cultivo de biocombustible ideal para la producción de etanol (Lam et al., 2009; Santchurn et al., 2014). El género Saccharum tiene seis especies: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense y S. edule, entre las cuales, S. spontaneum se considera la especie silvestre con un conjunto cromosómico básico de x = 8. S. spontaneum es también una de las dos principales especies de Saccharum que contribuyen al desarrollo de cultivares modernos de caña de azúcar (Dhont et al., 1998; Ha et al., 1999). S. spontaneum tiene una fuerte adaptabilidad ambiental y contiene importantes rasgos genéticos para la resistencia a enfermedades y sequías, contribuyendo así a la tolerancia al estrés de los híbridos de cultivares modernos (Grivet et al., 2004). S. spontaneum tiene la distribución geográfica más amplia y sus niveles de ploidía oscilan entre 2n = 5x = 40 y 2n = 16x = 128 (Panje y Babu, 1960).
Los cromosomas de la caña de azúcar tienen morfologías similares y una pequeña tamaño de 1 a 6 μm en la etapa de metafase condensada (Ha et al., 1999; Dhont, 2005); por lo tanto, es muy difícil discernir entre los diferentes cromosomas basándose en métodos citogenéticos tradicionales. Además, como autopoliploide, sus alteraciones estructurales cromosómicas causadas por poliploidización, duplicación, deleción y recombinación son muy comunes (Piperidis et al., 2010). En S. spontaneum, se han desarrollado mapas de ligamiento genético (Silva et al., 1995), pero el desarrollo de un mapa genético citológico va a la zaga de otras especies de gramíneas. Se pueden usar herramientas citológicas confiables para identificar cromosomas individuales para la investigación del genoma y la utilización de recursos de germoplasma, específicamente con el trasfondo del genoma complejo de los cultivares modernos de caña de azúcar. Los clones de cromosomas artificiales bacterianos específicos de cromosomas (BAC) son un recurso invaluable para las investigaciones del genoma de la caña de azúcar y tendrán muchas aplicaciones en el mapeo físico, la identificación de cromosomas y la reproducción asistida por marcadores de Saccharum spp., Y en la asistencia en proyectos de secuenciación y ensamblaje del genoma de la caña de azúcar. .
La hibridación fluorescente in situ (FISH) es una herramienta poderosa para la citología molecular (Jiang y Gill, 2006). En Saccharum spp., Se utilizaron 45S rDNA y 5S rDNA como sondas para detectar los cromosomas básicos de S. spontaneum, S. robustum y S. officinarum, para identificar los diferentes cromosomas básicos, x = 8 en S. spontaneum, yx = 10 en S. robustum y S. officinarum (Dhont et al., 1998; Ha et al., 1999). Según FISH, se encontró que los cultivares modernos de caña de azúcar contienen entre el 70% y el 80% de los cromosomas derivados de S. officinarum y entre el 10% y el 23% de los cromosomas de S. spontaneum, mientras que entre el 5% y el 17% parece ser el producto de la recombinación entre S. spontaneum y S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). Además, se detectaron eventos de eliminación, recombinación y translocación de cromosomas en algunos progenitores derivados de la hibridación entre Saccharum y Erianthus arundinaceus usando FISH (Dhont et al., 1995; Huang et al., 2015).
Sorghum tiene un pequeño genoma diploide (730 Mbp) con una baja frecuencia de eventos de recombinación cromosómica. El sorgo comparte una alta sintencia con el genoma de la caña de azúcar, por lo que es una planta de referencia ideal para análisis comparativos con el genoma de la caña de azúcar (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ). En este estudio, basado en las secuencias disponibles para recursos BAC de alta cobertura, se desarrollaron sondas BAC confiables para la identificación de cromosomas FISH y la construcción de mapas citogenéticos. Los objetivos de este estudio fueron: (1) desarrollar un conjunto de sondas BAC-FISH cromosómicas específicas en el cromosoma de S. spontaneum y (2) explorar el reordenamiento cromosómico entre S. spontaneum y sorgo.
Materiales y métodos
Materiales
Saccharum spontaneum SES208 (2n = 8x = 64) se utilizó para los análisis citológicos en este estudio. Las plantas SES208 se cultivaron en el campo en el campus de la Universidad Agrícola y Forestal de Fujian (Fuzhou, China) en febrero de 2015 y se mantuvieron en condiciones normales de crecimiento de la caña de azúcar.
Se construyó una biblioteca BAC a partir del genoma haploide de S. spontaneum AP85-441 (4x = 32), que se derivó de S. spontaneum SES208 (2n = 8x = 64) a través de un cultivo in vitro de anteras. El tamaño del genoma de AP85-441 es de aproximadamente 3,2 Gbp. La biblioteca constaba de 38.400 clones, con un tamaño medio de inserción de 100 kb, que cubría 6 veces todo el genoma.Esta biblioteca BAC se ha utilizado para identificar los transportadores de sacarosa y las familias de genes de fructoquinasa en S. spontaneum (Zhang et al., 2016; Chen et al., 2017).
Screening the BAC Library
Se combinaron 35156 clones de BAC de las bibliotecas AP85-441 en 701 bibliotecas. Cada biblioteca contiene un promedio de 50 clones BAC. Las bibliotecas de ADN se prepararon con el kit PhasePrepTM BAC DNA (Sigma, Estados Unidos) siguiendo los protocolos del fabricante. Las bibliotecas de ADN de BAC se secuenciaron utilizando la plataforma Illumina HiSeq 2500 con el modelo PE250. Se secuenciaron un total de 686 bibliotecas (18 bibliotecas fallaron en la secuenciación) y se generaron 267,5 Gb de datos limpios después del recorte utilizando Trimmomatic versión 0.36 (Bankevich et al., 2012). Los datos de cada grupo de BAC se recopilaron utilizando SPAdes versión 3.09 con el parámetro predeterminado. Se ensamblaron un total de 2611145 contigs con contig N50 de 7.38 kbp (Zhang y Ming, datos no publicados).
Las secuencias de la biblioteca AP85-441 BAC se buscaron con BLAST contra el genoma del sorgo con un valor E de 1e -4. Las secuencias que tenían un solo golpe BLAST en el genoma del sorgo se seleccionaron como sondas FISH candidatas. Para identificar clones de BAC de bajo número de copias de la biblioteca BAC, se diseñaron cebadores específicos de secuencia utilizando el software Primer 5.0 para permitir que los grupos de BAC se criben mediante PCR específicamente. Se seleccionaron longitudes de cebador de 18 a 25 pb, valores de Tm de 55 a 65 ° C y composiciones de nucleótidos de 40 a 60% de citosina y guanina. Se utilizó un método de PCR de dos pasos para seleccionar los grupos de dimensiones 3D de la biblioteca BAC (Asakawa et al., 1997; Crooijmans et al., 2000) con algunas modificaciones. Cada clon de una placa de 384 pocillos se cultivó en 80 μl de caldo de lisogenia + 34 mg / ml de cloranfenicol durante la noche a 37 ° C en una placa de cultivo de 384 pocillos. En total, se cultivaron ocho placas de 384 pocillos con un total de 3072 clones BAC. Para cada placa de 384 pocillos, primero construimos conjuntos de 16 filas (fila A a P) y 24 conjuntos de columnas (columna 1–24) para cada placa de 384 pocillos mezclando volúmenes iguales de cultivo de cada clon individual, y luego mezclamos los Grupos de 16 filas y grupo de 24 columnas junto con volúmenes iguales de cada grupo de filas y columnas como un grupo de placas o superpool. En este estudio, nos centramos en el cribado de ocho placas de 384 pocillos. Por tanto, se prepararon ocho superpools para las ocho placas por separado. Se identificó la placa positiva de 384 pocillos de las ocho placas y posteriormente se examinó la fila positiva entre las 16 mezclas. Finalmente, las columnas de PCR positivas se seleccionaron entre 24 mezclas de columnas. Se realizaron veinticuatro reacciones de PCR para identificar clones positivos de las ocho placas de 384 pocillos.
Las amplificaciones de PCR se realizaron como se describió anteriormente (Bouzidi et al., 2006). Cada reacción incluyó una premezcla de rTaq 7,5 μl, 0,6 μl de cada cebador (10 μM), 1,0 μl de plantilla líquida de bacterias, hasta un volumen final de 20 μl. Después de la desnaturalización inicial a 95 ° C durante 5 min, se realizaron 35 ciclos de 95 ° C durante 30 s, 55 ° C durante 30 sy 72 ° C durante 1 min. Los productos de la PCR se analizaron en un gel de agarosa al 1,5%.
Preparación del cromosoma en metafase
La preparación del cromosoma se realizó como se describió anteriormente (Lou et al., 2010) con modificaciones menores. En resumen, se cultivaron plantas de S. spontaneum en condiciones de invernadero para la recolección de las puntas de las raíces. Las puntas de las raíces extirpadas de aproximadamente 1 a 2 cm se trataron con 0,002 mol / l de 8-hidroxiquinolina a temperatura ambiente durante 2 a 4 h, se enjuagaron con agua durante 15 min y se fijaron en etanol: ácido acético (3: 1) durante al menos 24 ha temperatura ambiente. Luego, las puntas de las raíces se digirieron en una solución de enzima (4% de celulosa R-10 y 2% de pectoliasa en tampón citrato 0,1 M) a 37 ° C durante 1 h, se lavaron con agua desionizada con hielo durante 30 min y finalmente se incubaron en etanol: ácido acético (3: 1) durante aproximadamente 30 min. Los portaobjetos se prepararon de acuerdo con el método de «secado a la llama» (Iovene et al., 2008).
Purificación de ADN de BAC y etiquetado de la sonda
El ADN de BAC se extrajo con PhasePrepTM BAC DNA Kit de acuerdo con el manual del fabricante. El ADN de BAC purificado se marcó mediante una reacción de traducción de muescas estándar, que incluía ADNasa I diluida, tampón de traducción de muescas 10 ×, ADN polimerasa I, dNTP, dUTP marcados con biotina / digoxigenina y ADN de BAC. se incubó a 15 ° C durante 1,5 h.Los productos cortados se examinaron luego en un gel de agarosa al 1,5% para detectar la presencia de un frotis entre 300 y 500 pb.Las sondas obtenidas se almacenaron a -20 ° C hasta su uso.
Hibridación y detección in situ
Mezcla e hibridación de la sonda: Primero, la mezcla de la sonda (50% de formamida desionizada, 2 × SSC, 80 ng de ADN marcado con digoxigenina / biotina, 10% de sulfato de dextrano , > 1 μg C ° t-100) se colocó en un bloque caliente a 90 ° C durante 5 min, luego se transfirió inmediatamente en hielo hasta que esté listo para sondear en busca de hibridación en. Los portaobjetos secados a la llama previamente preparados se trataron con formamida desionizada al 70%, desnaturalizada en un bloque de calor a 80 ° C durante 1.5 min, inmediatamente sumergido secuencialmente en etanol al 70% helado, etanol al 90% y luego etanol al 100% cada uno durante 5 min, y luego secado al aire en el banco. Finalmente, se agregaron sondas desnaturalizadas a cada portaobjetos y se cubrieron con cubreobjetos de 24 x 32 mm. Los portaobjetos se colocaron en una cámara húmeda a 37 ° C durante la noche.
Detección de sonda: se retiraron los cubreobjetos y se lavaron los portaobjetos a temperatura ambiente en 2 × SSC durante 5 min, a 42 ° C en 2 × SSC durante 10 min y a temperatura ambiente en 1x PBS durante 5 min en esta secuencia. Las señales de sonda marcadas con biotina se detectaron con 2 mg / ml de estreptavidina Alexa Fluor 488 y las señales de sonda marcadas con digoxigenina se detectaron con 2% de anti-digoxigenina-rodamina de oveja. El cóctel de anticuerpos (100 μl de tampón TNB, 1 μl de estreptavidina Alexa Fluor 488, 1 μl de rodamina anti-dig-sheep) se añadió a los portaobjetos, que se cubrieron con cubreobjetos de 24 × 32 mm, incubados durante 1 ha 37 ° C en en una cámara húmeda y se lavó a temperatura ambiente en 1 × PBS tres veces durante 5 min cada una. Se eliminó el exceso de líquido y se añadió 4 ‘, 6-diamidino-2-fenilindol (DAPI) (Sigma, St. Louis, MO, Estados Unidos) en la solución antidesvanecimiento Vectashield (Vector, Burlingame, CA, Estados Unidos) para contratinir la cromosomas. Las imágenes se capturaron con un microscopio de epifluorescencia Olympus BX63. Las imágenes de la señal FISH se analizaron utilizando el software CellSens Dimension.
Resultados
Detección de clones de BAC de copia baja
Para analizar los clones de BAC que potencialmente tienen un número de copias bajo secuencias en el genoma de S. spontaneum, BLAST buscó en las bibliotecas de BAC AP85-441 secuenciadas contra el genoma del sorgo. Se cribaron un total de 2000 secuencias BAC correspondientes a 10 cromosomas de sorgo con buena colinealidad y que muestran especificidad de secuencia. Las secuencias de 2000 AP85-441 BAC fueron enmascaradas por REPEATMASKER contra la base de datos de ADN de sorgo de alta repetición y la base de datos de gramíneas TIGR para filtrar secuencias repetidas. Por último, se seleccionaron 114 BAC distribuidos en 10 cromosomas de sorgo para el análisis FISH (Tabla complementaria 1) con 7–16 BAC en cada uno de los 10 cromosomas del sorgo (Figura 1). Para seleccionar clones BAC positivos, se diseñaron 114 cebadores específicos basados en las secuencias de clones BAC (Tabla complementaria 2) y se seleccionaron 49 clones BAC positivos de las bibliotecas BAC (Tabla complementaria 3).
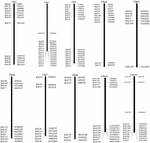
FIGURA 1. Las distribuciones de 114 clones BAC de AP85-441 en los cromosomas del sorgo. BAC1-114 representa el número BAC, los datos de la derecha son la ubicación física de BAC en la región correspondiente de los cromosomas del sorgo. Unit = bp.
Intensidad y distribución de la señal BAC-FISH
Muestras de ADN aisladas de las 49 positivas Los clones de BAC se marcaron con biotina o digoxigenina. Las sondas de ADNr 5S y ADNr 45S se utilizaron como control y se hibridaron con cromosomas en metafase somática de S. spontaneum siguiendo el procedimiento FISH. Debido a la correlación entre la fuerza de la señal y la variación del contenido de secuencia repetida de las secuencias BAC, usamos C ° t-100 como competidor (Tabla 1). Se seleccionaron clones de BAC que mostraban señales de hibridación fuertes y constantes para un análisis FISH adicional.

TABLA 1. Clasificación de 27 clones positivos con ADN bloqueante.
Se puede observar un total de 64 cromosomas distintos en la metafase del S. spontaneum. Cada BAC-FISH se realizó en cuatro experimentos independientes (o portaobjetos). Se analizaron al menos 10 extensiones de cromosomas somáticos en metafase en cada portaobjetos. Los resultados mostraron que las 27 sondas específicas (Tabla complementaria 4) se pueden clasificar en cinco grupos según la señal de hibridación (Tabla 1). En el Grupo I (incluidos BAC-18, BAC-20, BAC-29 y BAC-84), las sondas mostraron ocho sitios distintivos sin supresión competitiva in situ (CISS) utilizando C ° t-100 (Figura 2A), lo que sugiere que se repitieron pocos existían secuencias en estas secuencias BAC. En el Grupo II (incluidos BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 y BAC-74), C ° t -100 se utilizó como competidor para la hibridación de la sonda BAC, y se mostraron de seis a siete sitios bastante distintos en los cariotipos de este grupo (Figura 2B). El resultado de la hibridación fue similar al ADNr de 45S con siete sitios de señal, lo que puede ser causado por la variación de la estructura cromosómica en el cromosoma homólogo. En el Grupo III (incluidos BAC-26, BAC-66, BAC-76 y BAC-78), se utilizó ADN de C ° t-100 como competidor y se pudieron observar ocho sitios distintos en el cariotipo, lo que sugiere este grupo de BAC los clones tienen secuencias de ADN repetitivas (Figura 2C). En el Grupo IV (incluido BAC-24), se observaron más de ocho sitios de señal en el cariotipo y aparecieron en las regiones pericentromérica y telomérica (Figura 2D), lo que sugiere que estas sondas BAC tenían secuencias repetitivas en S. spontaneum.En el grupo V (incluidos BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 y BAC-81), las sondas mostraron señales dispersas en todos los cromosomas con regiones limitadas de cromosomas individuales que muestran señales específicas (Figura 2E). El resultado de la hibridación indicó que este grupo de clones BAC contenía grandes cantidades de ADN repetitivo en S. spontaneum, lo que puede ser causado por una rápida divergencia evolutiva de las regiones repetitivas después de la división del sorgo y S. spontaneum. Por lo tanto, de las 49 sondas BAC, un conjunto de 19 sondas BAC (Tabla complementaria 3) de los Grupos I, II y III mostraron señales específicas en los cromosomas de S. spontaneum correspondientes a nueve de los cromosomas del sorgo además de Sb05. Las señales de las otras 22 sondas se dispersaron en todos los cromosomas. Seleccionamos un conjunto de 19 sondas BAC como señales específicas del cromosoma de S. spontaneum (Figura 3). De las 19 sondas BAC, una sonda se ubicó en Sb02, Sb04 y Sb09; dos sondas en Sb07, Sb08 y Sb10; tres sondas en Sb01 y Sb06; y cuatro sondas en Sb03. Este conjunto de sondas proporcionó una herramienta útil para el análisis comparativo de S. spontaneum y sorgo (Tabla 2).
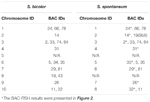
TABLA 2. Distribución de las sondas BAC-FISH en los cromosomas de S. bicolor y S. spontaneum.
Construcción de una biblioteca BAC específica para los cromosomas de S. spontaneum
Sb01 y Sb02 son los dos cromosomas de sorgo más grandes. Para detectar la sintencia del genoma entre el sorgo y S. spontaneum, se seleccionaron 10 clones BAC específicos de cromosomas de los Grupos I, II y III. De estas 10 sondas BAC, tres sondas (ID del clon BAC: 24, 66, 78) eran homólogas a Sb01 y siete sondas (ID del clon BAC: 14, 18, 20, 69, 71, 73, 77) correspondían a Sb02. Utilizamos la detección de doble color de FISH para pares BAC marcados de forma complementaria y las sondas para mapear la relación citogenética de la cadena para los clones BAC. Al hacerlo, construimos el mapa citogenético de S. spontaneum basado en las 10 sondas BAC.
Para tres sondas correspondientes a Sb01, el análisis FISH de S. spontaneum (SES208) mostró que BAC-24 y BAC -78 sondas se localizaron principalmente en la región distal del cromosoma 1 de S. spontaneum, y se mapeó BAC-66 muy cerca de la región centromérica (Figura 4). BAC-24 y BAC-66 ubicados en el mismo cromosoma (cromosoma 1 de S. spontaneum); simultáneamente, mientras que la sonda 66 y la sonda 78 también se encuentran en el mismo cromosoma, lo que demuestra que las tres sondas de BAC se distribuyen en los mismos cromosomas en S. spontaneum, lo que sugiere sintetización entre S. spontaneum y sorgo para el cromosoma 1. Los resultados de FISH revelaron que las siete sondas de BAC alineado con Sb02; solo las sondas 69, 71, 73 y 77 de los siete BAC generaron señales intensas en un cromosoma de S. spontaneum (SsChr2) (Figura 5); mientras que las sondas 14, 18 y 20 fueron indetectables en el cromosoma 2 de S. spontaneum pero se observaron en otros cromosomas de S. spontaneum. Estos resultados demostraron que los reordenamientos cromosómicos ocurrieron en el Sb02 correspondiente entre S. spontaneum y sorgo.
Además, la secuencia de BAC-2 compartió una alta similitud (97%) con un fragmento en el cromosoma 3 de sorgo. El análisis FISH de doble color mostró que tanto el ADNr BAC-2 como el 45s se mapearon en la misma ubicación del cromosoma de S. spontaneum (Figura 6).
Reordenamiento cromosómico en Sb02 entre sorgo y S. spontaneum
Para investigar más a fondo el reordenamiento cromosómico en Sb02 entre sorgo y S. spontaneum, cromosoma -se utilizaron sondas específicas correspondientes a Sb02 (BAC-14, BAC-18 y BAC-20) y correspondientes a Sb08 (BAC-19 y BAC-43) para el análisis FISH. Los resultados mostraron que BAC-14 y BAC-19 se mapearon en diferentes brazos de los mismos cromosomas de S. spontaneum, lo que indica que se produjeron reordenamientos cromosómicos entre los cromosomas 2 y 8 en S. spontaneum y sorgo (Figura 7).
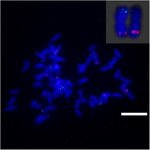
FIGURA 7. El reordenamiento de los cromosomas en la caña de azúcar y el sorgo según lo revelado por FISH. Sondas BAC-14 y BAC-19 marcadas con biotina (verde) y digoxigenina (rojo). Barras de escala = 10 μm.
Discusión
Saccharum es un género complejo caracterizado por altos niveles de poliploidía, con cromosomas pequeños (Ha et al., 1999; Dhont, 2005). Distinguir los cromosomas individuales de Saccharum en función de su morfología es un gran desafío. Aprovechando las secuencias que están muy cubiertas por la biblioteca BAC de S. spontaneum, pudimos cribar los posibles clones de BAC de bajo número de copias en S. spontaneum utilizando el genoma del sorgo como referencia. Como especie con alto contenido de poliploide, S. spontaneum puede contener más reordenamientos del genoma que los diploides.Las diferencias de los genomas entre S. spontaneum y sorgo pueden ser la causa de la inexactitud en las predicciones del bajo número de copias de secuencias BAC en el genoma de S. spontaneum. En este estudio, hemos obtenido inicialmente 114 secuencias BAC potenciales con copia única basadas en el genoma del sorgo. Sin embargo, solo 49 pudieron identificarse en las bibliotecas BAC parciales, y se encontró que 27 eran sondas BAC-FISH específicas de cromosomas. Hay al menos dos razones para explicar estos resultados. En primer lugar, las 114 secuencias de BAC eran fragmentos parciales de la inserción de BAC debido a las limitaciones del ensamblaje de secuencias de NGS y pueden contener secuencias repetidas en las otras regiones que no tienen información de secuencia. Estas secuencias repetidas potencialmente descubiertas podrían inducir a error a la detección de secuencias con pocas copias a través de la comparación de secuencias. En segundo lugar, la poliploidización reciente en S. spontaneum puede causar la variación de secuencias repetidas entre S. spontaneum y sorgo y, por lo tanto, dar como resultado la señal FISH inespecífica en S. spontaneum. En un estudio anterior, la secuenciación de 20 BAC en híbridos de caña de azúcar generó secuencias contig de 1,45 Mb, las secuencias alineadas con el genoma del sorgo abarcaron 0,99 Mb, lo que representó aproximadamente la mitad de las secuencias BAC de la caña de azúcar (Wang et al., 2010). Obviamente, existían deleciones / inserciones entre sorgo y Saccharum, y la utilización del genoma del sorgo como referencia no puede reemplazar las características únicas del genoma de Saccharum y, por lo tanto, puede causar resultados de FISH impredecibles.
Algunos portaobjetos de FISH tuvieron fuertes resultados. ruido de fondo, que puede deberse a pequeñas regiones de secuencias repetidas (como secuencias SSR) en el clon BAC, que en consecuencia producen interferencias durante la hibridación. El genoma del sorgo tiene un contenido repetido de aproximadamente el 61% (Paterson et al., 2009), mientras que aproximadamente la mitad del genoma está compuesto por secuencias repetidas en híbridos de Saccarrum (De et al., 2014). En estos genomas ricos en repeticiones, es difícil desarrollar sondas FISH específicas de señal, que distinguen citogenéticamente cromosomas con morfologías similares. CISS con ADN bloqueante C ° t-100 puede excluir eficazmente secuencias repetidas. En este estudio, se utilizó ADN de bloqueo C ° t-100 para el análisis FISH de las sondas BAC en los Grupos II y III (Figuras 2D-F), que se verificó que eran las sondas BAC cromosómicas específicas, mientras que las sondas FISH de BAC en los Grupos IV y V produjo una fuerte interferencia de fondo (Figuras 2D-F). Es obvio que existen variaciones de secuencias repetidas entre las sondas BAC examinadas. Por lo tanto, la optimización de los experimentos sería necesaria para el análisis BAC-FISH en Saccharum. Se mostraron de seis a siete sitios en el Grupo II, lo que podría ser causado por las variaciones de la estructura cromosómica homóloga, como la deleción del fragmento en uno o dos cromosomas homólogos. En el trigo hexaploide, BAC 676D4 se hibridó más fuertemente con los cromosomas del genoma A que con los cromosomas del genoma B y D (Zhang et al., 2004). Estas señales indetectables de una o dos en los cromosomas homólogos también podrían ser causadas por problemas técnicos de BAC-FISH para tantos cromosomas con tamaño pequeño.
En este estudio, identificamos 27 BAC- específicos de cromosomas Sondas FISH que corresponden a 9 de los 10 cromosomas del sorgo (todos excepto Sb05). En un estudio del mapa genético derivado de un cruce entre S. officinarum y el cultivar de caña de azúcar, Sb05 se fusionó con Sb06 en la caña de azúcar (Aitken et al., 2014). En nuestro estudio, un mapa genético basado en la población F1 de S. spontaneum reveló que Sb05 se dividió en dos segmentos, y los dos segmentos se fusionaron con Sb06 y Sb07, respectivamente (Zhang y Ming, datos no publicados). Recientemente, el mapa genético de un miembro de Andropogoneae, Miscanthus sinensis, demostró que Sb05 tiene mala colinealidad con el grupo de ligamiento correspondiente en M. sinensis (Ma et al., 2012). De manera similar, los dos cromosomas ancestrales del maíz ortólogos de Sb05 retienen el menor número de ortólogos sinténicos de los genes del sorgo (Schubert y Lysak, 2011). Por lo tanto, la ausencia del BAC-FISH correspondiente a Sb05 puede indicar la fusión cromosómica en S. spontaneum.
El sorgo y S. spontaneum divergieron hace 12 millones de años (MYA), el número de cromosomas básicos se redujo de x = 10 ax = 8. En Aitken et al. (2014), el HG2 (grupo homólogo 2) de la caña de azúcar alineado con Sb05 y Sb06, y el HG8 (grupo homólogo 8) con Sb02 y Sb08, proporcionando evidencia del evento básico de reducción de cromosomas (Aitken et al., 2014). En este estudio, observamos que los cromosomas de sorgo Sb02 y Sb08 tenían reordenamiento intercromosómico en S. spontaneum como lo demuestra la evidencia de que BAC-14 se alineó con Sb02 y BAC-19 se alineó con Sb08 (Figura 7). Nuestro estudio proporcionó la primera evidencia física y citogenética para el reordenamiento entre cromosomas del sorgo en S. spontaneum. Los datos no publicados de nuestro grupo también revelaron que Sb08 se divide en dos segmentos, y se fusionaron con Sb02 y Sb09 en S. spontaneum, respectivamente (Zhang y Ming, datos no publicados).Las sondas correspondientes a Sb09, Sb08, Sb05 y Sb06 podrían usarse más para investigar los eventos intercromosómicos entre sorgo y S. spontaneum. S. spontaneum tiene una amplia gama de niveles de ploidía (2n = 40-128) (Irvine, 1999). Estas sondas de BAC podrían usarse para confirmar el reordenamiento intercromosómico de S. spontaneum con los diferentes niveles de poliploidía.
Debido al diferente número de cromosomas básicos entre sorgo (x = 10) y S. spontaneum (x = 8), los cromosomas del sorgo no eran una correspondencia biunívoca con S. spontaneum. Como se mencionó anteriormente en nuestro estudio de mapeo genético, Sb08 se dividió en dos segmentos que se fusionaron con segmentos de Sb02 y segmentos de Sb09 en S. spontaneum; Sb05 se dividió en dos segmentos que se fusionaron con segmentos de Sb06 y segmentos de Sb07 S. spontaneum. Por tanto, las sondas correspondientes a Sb08 y Sb05 no eran específicas de cromosomas individuales de S. spontaneum, mientras que las otras sondas correspondientes a los otros ocho cromosomas de sorgo se pueden utilizar como sondas BAC-FISH citogenéticas específicas de cromosomas para S. spontaneum. Por lo tanto, las sondas BAC probadas se basaron en los ocho cromosomas de sorgo (Tabla 2) y fueron suficientes para la identificación de cromosomas usando BAC-FISH. Se encontró un cromosoma satélite en un cromosoma de S. spontaneum (cromosoma 3) (Ha et al., 1999). Previamente, la FISH simultánea reveló que las señales del rDNA 45S estaban ubicadas en las constricciones secundarias de los cromosomas satélite dentro del cromosoma 3 del clon de S. spontaneum derivado del cultivo de anteras (AP85-361) (Ha et al., 1999). En este estudio, BAC-2 que corresponde a Sb03 y 45s rDNA fueron mapeados en la misma ubicación del cromosoma de S. spontaneum, apoyando así aún más hallazgos previos (Ha et al., 1999). Nuestros resultados también proporcionaron evidencia directa de que el cromosoma 3 de S. spontaneum nombrado en el estudio anterior es homólogo a Sb03.
Conclusión
En este estudio, desarrollamos BAC específicos de cromosomas de S . spontaneum como un paso hacia el desarrollo de un método simple y reproducible para la identificación de cromosomas usando marcadores citogenéticos BAC-FISH, confirmando la viabilidad de aislar BACs específicos de cromosomas basados en el genoma del sorgo para construir un mapa físico de la caña de azúcar. También proporcionamos la primera evidencia citogenética de reordenamiento intercromosómico entre sorgo y S. spontaneum. El establecimiento del sistema de tecnología de la caña de azúcar BAC-FISH ofrece nuevas oportunidades y los medios para la investigación de la citogenética molecular de la caña de azúcar, incluido el análisis del cariotipo, la localización de genes y la construcción de mapas físicos. Estos resultados son esenciales para el ensamblaje del genoma de S. spontaneum requerido para la secuenciación del genoma completo.
Contribuciones de los autores
GD, JS y JZ concibieron el estudio y diseñaron los experimentos. GD, JS, QZ, JW, QY, RM, KW y JZ llevaron a cabo los experimentos y analizaron los datos. GD y JZ escribieron el manuscrito. Todos los autores leyeron y aprobaron el documento final.
Financiamiento
Este proyecto fue apoyado por subvenciones del programa 863 (2013AA102604), NSFC (31201260), Programa para Talentos Excelentes del Nuevo Siglo en Universidad de la provincia de Fujian, y la financiación de la Universidad de Agricultura y Silvicultura de Fujian.
Declaración de conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de relaciones comerciales o financieras que podría interpretarse como un posible conflicto de intereses.
Agradecimientos
Agradecemos al Dr. Xintan Zhang por ensamblar las secuencias BAC y a la Dra. Irene Lavagi por editar el inglés.
Material complementario
Dhont, A. (2005). Desentrañar la estructura del genoma de poliploides usando FISH y GISH; ejemplos de caña de azúcar y plátano. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
Resumen de PubMed | CrossRef Texto completo | Google Académico
Piperidis, G., D’Hont, A. y Hogarth, D. M. (2001). «Análisis de la composición cromosómica de varios híbridos interespecíficos de Saccharum por genómica in situ (hibridación) (GISH)», en Actas de la Sociedad Internacional de Tecnólogos de la Caña de Azúcar, XXIV Congreso (Brisbane, QLD: ASSCT), 565–566.
Google Académico
