Inleiding
Suikerriet (Saccharum spp., Poaceae) is een eenjarig of meerjarig gewas dat wordt geteeld in tropische en subtropische gebieden over de hele wereld. Suikerriet is een belangrijk gewas voor de productie van sucrose en is goed voor 70% van de wereldproductie van suiker. Bovendien kan suikerriet als C4-plant efficiënt zonne-energie omzetten in chemische energie en is het daarom een ideaal biobrandstofgewas voor de productie van ethanol (Lam et al., 2009; Santchurn et al., 2014). Het geslacht Saccharum heeft zes soorten: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense en S. edule, waaronder S. spontaneum wordt beschouwd als de wilde soort met een basale chromosoomset van x = 8. S. spontaneum is ook een van de twee belangrijkste Saccharum-soorten die bijdragen aan de ontwikkeling van de moderne suikerrietcultivars (Dhont et al., 1998; Ha et al., 1999). S. spontaneum heeft een sterk aanpassingsvermogen aan de omgeving en bevat belangrijke genetische eigenschappen voor ziekte- en droogteresistentie, en draagt zo bij aan de stresstolerantie van moderne cultivarhybriden (Grivet et al., 2004). S. spontaneum heeft de breedste geografische spreiding en de ploïdie-niveaus variëren van 2n = 5x = 40 tot 2n = 16x = 128 (Panje en Babu, 1960).
Suikerrietchromosomen hebben vergelijkbare morfologieën en hebben een kleine grootte van 1-6 μm in het gecondenseerde metafase-stadium (Ha et al., 1999; Dhont, 2005); Daarom is het erg moeilijk om onderscheid te maken tussen de verschillende chromosomen op basis van traditionele cytogenetische methoden. Bovendien zijn, als autopolyploïde, zijn chromosomale structurele veranderingen veroorzaakt door polyploïdisatie, duplicatie, deletie en recombinatie zeer gebruikelijk (Piperidis et al., 2010). In S. spontaneum zijn genetische koppelingskaarten ontwikkeld (Silva et al., 1995), maar de ontwikkeling van een cytologische genetische kaart bleef achter bij andere grassoorten. Betrouwbare cytologische hulpmiddelen om individuele chromosomen te identificeren, kunnen worden gebruikt voor effectief genoomonderzoek en het gebruik van kiemplasma-bronnen, met name met de complexe genoomachtergrond van moderne suikerrietcultivars. Chromosoom-specifieke bacteriële kunstmatige chromosoom (BAC) klonen zijn van onschatbare waarde voor onderzoek naar het suikerrietgenoom, en zullen veel toepassingen hebben bij het fysiek in kaart brengen, chromosoomidentificatie en marker-geassisteerde veredeling van de Saccharum spp., En bij het assisteren van sequentiebepaling en assemblageprojecten van suikerriet .
Fluorescentie in situ hybridisatie (FISH) is een krachtig hulpmiddel voor moleculaire cytologie (Jiang en Gill, 2006). In Saccharum spp. Werden 45S-rDNA en 5S-rDNA gebruikt als sondes om de basischromosomen van S. spontaneum, S. robustum en S. officinarum te detecteren, om de verschillende basischromosomen te identificeren, x = 8 in S. spontaneum en x = 10 in S. robustum en S. officinarum (Dhont et al., 1998; Ha et al., 1999). Op basis van FISH bleken moderne suikerrietcultivars 70-80% van de chromosomen afkomstig van S. officinarum en 10-23% van de chromosomen van S. spontaneum te bevatten, terwijl 5-17% het product lijkt te zijn van de recombinatie tussen S. spontaneum en S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). Bovendien werden chromosoomeliminatie, recombinatie en translocatiegebeurtenissen gedetecteerd in sommige voorlopers die waren afgeleid van de hybridisatie tussen Saccharum en Erianthus arundinaceus met behulp van FISH (Dhont et al., 1995; Huang et al., 2015).
Sorghum heeft een klein diploïde genoom (730 Mbp) met een lage frequentie van chromosoomrecombinatiegebeurtenissen. Sorghum deelt een hoge syntenie met het suikerrietgenoom, waardoor het een ideale referentieplant is voor vergelijkende analyses met het suikerrietgenoom (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ). In deze studie werden, gebaseerd op de beschikbare sequenties voor BAC-bronnen met een hoge dekking, betrouwbare BAC-probes ontwikkeld voor FISH-chromosoomidentificatie en cytogenetische kaartconstructie. De doelstellingen van deze studie waren: (1) het ontwikkelen van een reeks chromosoomspecifieke BAC-FISH-sondes op S. spontaneum-chromosoom en (2) het onderzoeken van de chromosoomherschikking tussen S. spontaneum en sorghum.
Materialen and Methods
Materialen
Saccharum spontaneum SES208 (2n = 8x = 64) werd gebruikt voor cytologische analyses in deze studie. De SES208-planten werden in februari 2015 in het veld gekweekt op de campus van de Fujian Agricultural and Forestry University (Fuzhou, China) en onder normale omstandigheden voor suikerrietgroei gehouden.
Er werd een BAC-bibliotheek geconstrueerd van het haploïde genoom van S. spontaneum AP85-441 (4x = 32), die was afgeleid van S. spontaneum SES208 (2n = 8x = 64) via helmknop in vitro cultuur. De genoomgrootte van AP85-441 is ongeveer 3,2 Gbp. De bibliotheek bestond uit 38.400 klonen, met een gemiddelde insertgrootte van 100 kb, die 6 × van het hele genoom beslaan.Deze BAC-bibliotheek is gebruikt voor het identificeren van de sucrosetransporters en fructokinase-genfamilies in S. spontaneum (Zhang et al., 2016; Chen et al., 2017).
Screening van de BAC-bibliotheek
35.156 BAC-klonen uit de AP85-441-bibliotheken werden samengevoegd in 701-bibliotheken. Elke bibliotheek bevat gemiddeld 50 BAC-klonen. De DNA-bibliotheken werden bereid met de PhasePrepTM BAC DNA Kit (Sigma, Verenigde Staten) volgens de protocollen van de fabrikant. Van BAC-DNA-bibliotheken werd de sequentie bepaald met behulp van het Illumina HiSeq 2500-platform met PE250-model. Een totaal van 686 bibliotheken (18 bibliotheken konden de sequentie niet bepalen) werden gesequenced en 267,5 Gb aan opgeschoonde gegevens werd gegenereerd na trimmen met Trimmomatic versie 0.36 (Bankevich et al., 2012). De gegevens van elke BAC-pool zijn verzameld met SPAdes-versie 3.09 met standaardparameter. In totaal werden 2.611.145 contigs geassembleerd met contig N50 van 7,38 kbp (Zhang en Ming, ongepubliceerde gegevens).
De AP85-441 BAC-bibliotheeksequenties werden BLAST doorzocht tegen het sorghumgenoom met een E-waarde van 1e -4. De sequenties met enkele BLAST-hits in het sorghumgenoom werden geselecteerd als kandidaat-FISH-sondes. Om BAC-klonen met een laag aantal kopieën uit de BAC-bibliotheek te identificeren, werden sequentiespecifieke primers ontworpen met behulp van Primer 5.0-software om BAC-pools specifiek door PCR te laten screenen. Primerlengtes van 18-25 bp, Tm-waarden van 55-65 ° C en nucleotidesamenstellingen van 40-60% cytosine en guanine werden geselecteerd. Een tweestaps PCR-methode werd gebruikt om de 3D-dimensiepools van de BAC-bibliotheek te screenen (Asakawa et al., 1997; Crooijmans et al., 2000) met enkele aanpassingen. Elke kloon van een plaat met 384 putjes werd gedurende de nacht bij 37 ° C in een kweekplaat met 384 putjes gekweekt in 80 μl Lysogeny-bouillon + 34 mg / ml chlooramfenicol. In totaal werden acht platen met 384 putjes met in totaal 3072 BAC-klonen gekweekt. Voor elke 384-wells plaat hebben we eerst 16 rijen pools (rij A tot P) en 24 kolompools (kolom 1-24) geconstrueerd voor elke 384-wells plaat door gelijke volumes kweek van elke individuele kloon te mengen en vervolgens de Pool met 16 rijen en pool met 24 kolommen samen met gelijke volumes van elke pool met rijen en kolommen als één platenpool of superpool. In deze studie hebben we ons gericht op het screenen van acht platen met 384 putjes. Zo werden er acht superpools voorbereid voor de acht platen afzonderlijk. De positieve plaat met 384 putjes van de acht platen werd geïdentificeerd en vervolgens werd de positieve rij tussen de 16 mengsels gescreend. Ten slotte werden de PCR-positieve kolommen geselecteerd uit 24 kolommengsels. Vierentwintig PCR-reacties werden uitgevoerd om positieve klonen van de acht platen met 384 putjes te identificeren.
PCR-amplificaties werden uitgevoerd zoals eerder beschreven (Bouzidi et al., 2006). Elke reactie omvatte rTaq-premix 7,5 μl, 0,6 μl van elke primer (10 μM), 1,0 μl bacterievloeistofsjabloon, tot een eindvolume van 20 μl. Na een eerste denaturatie bij 95 ° C gedurende 5 minuten, werden 35 cycli van 95 ° C gedurende 30 seconden, 55 ° C gedurende 30 seconden en 72 ° C gedurende 1 minuut uitgevoerd. PCR-producten werden geanalyseerd op een 1,5% agarosegel.
Metafase-chromosoombereiding
Chromosoombereiding werd uitgevoerd zoals eerder beschreven (Lou et al., 2010) met kleine aanpassingen. In het kort werden S. spontaneum-planten gekweekt in de kasomstandigheden voor het oogsten van wortelpunten. Uitgesneden wortelpunten van ongeveer 1-2 cm werden behandeld met 0,002 mol / l 8-hydroxychinoline bij kamertemperatuur gedurende 2-4 uur, gespoeld in water gedurende 15 minuten en gefixeerd in ethanol: azijnzuur (3: 1) gedurende ten minste 24 uur bij kamertemperatuur. De wortelpunten werden vervolgens gedigereerd in een enzymoplossing (4% cellulose R-10 en 2% pectolyase in 0,1 M citraatbuffer) bij 37 ° C gedurende 1 uur, gewassen met ijs gedeïoniseerd water gedurende 30 minuten en tenslotte geïncubeerd in ethanol: azijnzuur (3: 1) gedurende ongeveer 30 minuten. Objectglaasjes werden bereid volgens de “vlamgedroogde” methode (Iovene et al., 2008).
BAC DNA-zuivering en probe-etikettering
Het BAC-DNA werd geëxtraheerd met PhasePrepTM BAC DNA-kit volgens de handleiding van de fabrikant. Gezuiverd BAC-DNA werd gelabeld door middel van standaard nick-translatiereactie, inclusief verdunde DNase I, 10 × nick-translatiebuffer, DNA-polymerase I, dNTP’s, met biotine / digoxigenine gelabelde dUTP’s en BAC-DNA. Het mengsel werd geïncubeerd bij 15 ° C gedurende 1,5 uur. De gesneden producten werden vervolgens onderzocht op een 1,5% agarosegel op de aanwezigheid van een uitstrijkje tussen 300 en 500 bp. De verkregen sondes werden bewaard bij -20 ° C tot ze werden gebruikt.
In situ hybridisatie en detectie
Probe mix en hybridisatie: Ten eerste, het probemengsel (50% gedeïoniseerd formamide, 2 × SSC, 80 ng digoxygenine / biotine-gelabeld DNA, 10% dextraansulfaat , > 1 μg C ° t-100) werd gedurende 5 minuten in een warm blok van 90 ° C geplaatst en vervolgens onmiddellijk op ijs overgebracht totdat het klaar was om te testen op hybridisatie Aan. De eerder bereide gevlamd-gedroogde objectglaasjes werden behandeld met 70% gedeïoniseerd formamide, gedenatureerd op een warmteblok bij 80 ° C gedurende 1.5 minuten, onmiddellijk achtereenvolgens ondergedompeld in ijskoude 70% ethanol, 90% ethanol en vervolgens 100% ethanol elk gedurende 5 minuten, en vervolgens aan de lucht gedroogd op de bank. Ten slotte werden gedenatureerde sondes aan elk objectglaasje toegevoegd en bedekt met dekglaasjes van 24 x 32 mm. Objectglaasjes werden ’s nachts in een vochtige kamer bij 37 ° C geplaatst.
Sondedetectie: de dekglaasjes werden verwijderd en de objectglaasjes werden 5 minuten bij kamertemperatuur in 2 × SSC gewassen, bij 42 ° C in 2 × SSC gedurende 10 minuten en bij kamertemperatuur in 1 × PBS gedurende 5 minuten in deze volgorde. Met biotine gemerkte probesignalen werden gedetecteerd met 2 mg / ml Alexa Fluor 488 streptavidine en met digoxygenine gemerkte probesignalen werden gedetecteerd met 2% anti-digoxigenine-rhodamine van schapen. De antilichaamcocktail (100 μl TNB-buffer, 1 μl Alexa Fluor 488 streptavidine, 1 μl rhodamine anti-dig-schaap) werd toegevoegd aan de objectglaasjes, die waren bedekt met dekglaasjes van 24 x 32 mm, 1 uur geïncubeerd bij 37 ° C in een vochtige kamer, en gewassen bij kamertemperatuur in 1 × PBS driemaal gedurende 5 minuten elk. Overtollige vloeistof werd verwijderd en 4 ‘, 6-diamidino-2-fenylindool (DAPI) (Sigma, St.Louis, MO, Verenigde Staten) in de antifade-oplossing Vectashield (Vector, Burlingame, CA, Verenigde Staten) werd toegevoegd om de chromosomen. Beelden werden gemaakt met een Olympus BX63 epifluorescentiemicroscoop. FISH-signaalbeelden werden geanalyseerd met behulp van de CellSens Dimension-software.
Resultaten
Screening van BAC-klonen met lage kopieën
Om de BAC-klonen te screenen die mogelijk een laag aantal kopieën hebben sequenties in het S. spontaneum-genoom, zochten we BLAST de gesequentieerde AP85-441 BAC-bibliotheken tegen het sorghumgenoom. Een totaal van 2000 BAC-sequenties die overeenkomen met 10 sorghumchromosomen met goede collineariteit en die sequentiespecificiteit vertonen, werden gescreend. De 2000 AP85-441 BAC-sequenties werden gemaskeerd door REPEATMASKER tegen de high-repeat sorghum-DNA-database en TIGR gramineae-database om herhaalsequenties te filteren. Ten slotte werden 114 BAC’s verdeeld over 10 sorghumchromosomen geselecteerd voor FISH-analyse (aanvullende tabel 1) met 7-16 BAC’s op elk van de 10 sorghum-chromosomen (Figuur 1). Om positieve BAC-klonen te screenen, werden 114 specifieke primers ontworpen op basis van de BAC-kloonsequenties (aanvullende tabel 2) en 49 positieve BAC-klonen werden gescreend uit de BAC-bibliotheken (aanvullende tabel 3).
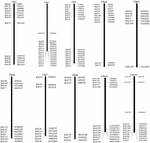
FIGUUR 1. De verdelingen van 114 BAC-klonen van AP85-441 in de chromosomen van sorghum. BAC1-114 vertegenwoordigt het BAC-nummer, de gegevens aan de rechterkant zijn de fysieke locatie van BAC in het corresponderende gebied van de sorghumchromosomen. Unit = bp.
BAC-FISH Signaalsterkte en -verdeling
DNA-monsters geïsoleerd uit de 49 positieve BAC-klonen werden gemerkt met biotine of digoxigenine. 5S-rDNA- en 45S-rDNA-probes werden gebruikt als controle en werden gehybridiseerd met S. spontaneum somatische metafasechromosomen volgens de FISH-procedure. Vanwege de correlatie tussen signaalsterkte en de variatie van herhaalde sequentie-inhoud van de BAC-sequenties, hebben we C ° t-100 als concurrent gebruikt (tabel 1). BAC-klonen die sterke en stabiele hybridisatiesignalen vertoonden, werden geselecteerd voor verdere FISH-analyse.

TABEL 1. De classificatie van 27 positieve klonen met blokkerend DNA.
In totaal kunnen 64 verschillende chromosomen worden waargenomen in de metafase van de S. spontaneum. Elke BAC-FISH werd uitgevoerd in vier onafhankelijke experimenten (of objectglaasjes). In elk glaasje werden ten minste 10 spreads van somatische metafasechromosomen geanalyseerd. De resultaten toonden aan dat de 27 specifieke probes (aanvullende tabel 4) kunnen worden ingedeeld in vijf groepen op basis van het signaal van hybridisatie (tabel 1). In groep I (inclusief BAC-18, BAC-20, BAC-29 en BAC-84) vertoonden sondes acht onderscheidende sites zonder competitieve in situ onderdrukking (CISS) met behulp van C ° t-100 (Figuur 2A), wat suggereert dat er weinig sequenties bestonden in deze BAC-sequenties. In groep II (inclusief BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 en BAC-74), C ° t -100 werd gebruikt als concurrent voor BAC-probe-hybridisatie en zes tot zeven vrij verschillende sites werden weergegeven in karyotypen van deze groep (Figuur 2B). Het hybridisatieresultaat was vergelijkbaar met 45S-rDNA met zeven signaalplaatsen, wat kan worden veroorzaakt door chromosoomstructuurvariatie in het homologe chromosoom. In groep III (inclusief BAC-26, BAC-66, BAC-76 en BAC-78) werd C ° t-100 DNA gebruikt als concurrent en konden acht verschillende plaatsen worden waargenomen in het karyotype, wat suggereert dat deze groep van BAC klonen hebben repetitieve DNA-sequenties (Figuur 2C). In groep IV (inclusief BAC-24) werden meer dan acht signaalplaatsen waargenomen in het karyotype en verschenen in de pericentromere en telomere gebieden (figuur 2D), wat suggereert dat deze BAC-probes repetitieve sequenties hadden in S. spontaneum.In groep V (inclusief BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 en BAC-81) vertoonden de sondes verspreide signalen in alle chromosomen met beperkte regio’s van individuele chromosomen die specifieke signalen weergeven (Figuur 2E). Het hybridisatieresultaat gaf aan dat deze groep BAC-klonen grote hoeveelheden repetitief DNA in S. spontaneum bevatte, wat kan worden veroorzaakt door een snelle evolutionaire divergentie voor de repetitieve regio’s na de splitsing van sorghum en S. spontaneum. Daarom vertoonde van de 49 BAC-sondes een set van 19 BAC-sondes (aanvullende tabel 3) uit groepen I, II en III specifieke signalen op S. spontaneum-chromosomen die overeenkomen met negen van de sorghum-chromosomen naast Sb05. De signalen van de andere 22 sondes waren verspreid over alle chromosomen. We selecteerden een set van 19 BAC-sondes als S. spontaneum chromosoomspecifieke signalen (Figuur 3). Van de 19 BAC-sondes bevond één sonde zich op Sb02, Sb04 en Sb09; twee sondes op Sb07, Sb08 en Sb10; drie sondes op Sb01 en Sb06; en vier sondes op Sb03. Deze probeset was een handig hulpmiddel voor vergelijkende analyse van S. spontaneum en sorghum (tabel 2).
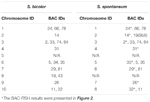
TABEL 2. De verdeling van BAC-FISH-sondes in chromosomen van S. bicolor en S. spontaneum.
Constructie van een BAC-bibliotheek specifiek voor S. spontaneum-chromosomen
Sb01 en Sb02 zijn de twee grootste sorghum-chromosomen. Om de genoomsyntenie tussen sorghum en S. spontaneum te detecteren, werden 10 chromosoomspecifieke BAC-klonen geselecteerd uit groepen I, II en III. Van deze 10 BAC-probes waren drie probes (BAC-kloon ID: 24, 66, 78) homoloog aan Sb01 en zeven probes (BAC-kloon ID: 14, 18, 20, 69, 71, 73, 77) kwamen overeen met Sb02. We gebruikten tweekleurige detectie van FISH voor complementair gelabelde BAC-paren en de probes voor het in kaart brengen van de cytogenetische ketenrelatie voor de BAC-klonen. Door dit te doen, hebben we de cytogenetische kaart van S. spontaneum geconstrueerd op basis van de 10 BAC-probes.
Voor drie probes die overeenkomen met Sb01, toonde FISH-analyse van S. spontaneum (SES208) aan dat BAC-24 en BAC -78 sondes bevonden zich voornamelijk op het distale gebied van S. spontaneum chromosoom 1, en BAC-66 werd in kaart gebracht in de nabijheid van het centromere gebied (Figuur 4). BAC-24 en BAC-66 gelokaliseerd op hetzelfde chromosoom (S. spontaneum chromosoom 1); tegelijkertijd terwijl sonde 66 en sonde 78 zich ook op hetzelfde chromosoom bevonden, wat aantoont dat de drie BAC-sondes verdeeld zijn over dezelfde chromosomen in S. spontaneum, wat duidt op syntenie tussen S. spontaneum en sorghum voor chromosoom 1. FISH-resultaten onthulden dat de zeven BAC-sondes afgestemd op Sb02; alleen sondes 69, 71, 73 en 77 van de zeven BAC’s genereerden intense signalen op één S. spontaneum-chromosoom (SsChr2) (Figuur 5); terwijl sondes 14, 18 en 20 niet detecteerbaar waren op S. spontaneum chromosoom 2 maar werden waargenomen op andere S. spontaneum chromosomen. Deze resultaten toonden aan dat de chromosoomherschikkingen plaatsvonden in de overeenkomstige Sb02 tussen S. spontaneum en sorghum.
Bovendien deelde de sequentie van BAC-2 een hoge gelijkenis (97%) met een fragment op chromosoom 3 van sorghum. Dubbelkleurige FISH-analyse toonde aan dat zowel BAC-2 als 45s rDNA in kaart waren gebracht op dezelfde locatie van het S. spontaneum-chromosoom (Figuur 6).
Chromosoomherschikking op Sb02 tussen Sorghum en S. spontaneum
Om de chromosoomherschikking op Sb02 tussen sorghum en S. spontaneum verder te onderzoeken, chromosoom -specifieke sondes die overeenkomen met Sb02 (BAC-14, BAC-18 en BAC-20) en die overeenkomen met Sb08 (BAC-19 en BAC-43), werden gebruikt voor FISH-analyse. De resultaten toonden aan dat BAC-14 en BAC-19 werden toegewezen aan verschillende armen van dezelfde S. spontaneum-chromosomen, wat aangeeft dat chromosoomherschikkingen plaatsvonden tussen chromosomen 2 en 8 in S. spontaneum en sorghum (Figuur 7).
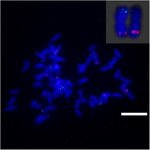
FIGUUR 7. De herschikking van chromosoom in suikerriet en sorghum zoals onthuld door FISH. BAC-14 en BAC-19 probes gelabeld met biotine (groen) en digoxigenine (rood). Schaalbalken = 10 μm.
Discussie
Saccharum is een complex geslacht dat wordt gekenmerkt door hoge polyploïdie-niveaus, met kleine chromosomen (Ha et al., 1999; Dhont, 2005). Het onderscheiden van individuele Saccharum-chromosomen op basis van hun morfologie is een grote uitdaging. Door gebruik te maken van sequenties die in hoge mate worden gedekt door de BAC-bibliotheek van S. spontaneum, waren we in staat om te screenen op de potentiële BAC-klonen met een laag aantal kopieën in S. spontaneum met behulp van het sorghumgenoom als referentie. Omdat S. spontaneum een hoge polyploïde soort is, kan het meer genoomherschikkingen bevatten dan diploïden.De verschillen in de genomen tussen S. spontaneum en sorghum kunnen de oorzaak zijn van onnauwkeurigheid in de voorspellingen van het lage aantal kopieën van BAC-sequenties in het S. spontaneum-genoom. In deze studie hebben we in eerste instantie 114 potentiële BAC-sequenties verkregen met enkele kopie op basis van het sorghumgenoom. Er konden echter slechts 49 worden geïdentificeerd in de gedeeltelijke BAC-bibliotheken en 27 bleken chromosoomspecifieke BAC-FISH-sondes te zijn. Er zijn minstens twee redenen om deze resultaten te verklaren. Ten eerste waren de 114 BAC-sequenties gedeeltelijke fragmenten van de BAC-insertie vanwege de beperkingen van NGS-sequentiesamenstelling, en kunnen herhaalsequenties bevatten in de andere regio’s die geen sequentie-informatie hebben. Deze potentieel ongedekte herhalingsreeksen zouden de detectie van sequenties met weinig kopieën kunnen misleiden door sequentievergelijking. Ten tweede kan de recente polyploïdisatie in S. spontaneum de variatie van herhalingssequenties tussen S. spontaneum en sorghum veroorzaken, en dus resulteren in het niet-specifieke FISH-signaal in S. spontaneum. In een eerdere studie genereerde het sequencen van 20 BAC’s in suikerriethybriden 1,45 Mb aaneengesloten sequenties, waarbij de sequenties overeenkwamen met het sorghumgenoom 0,99 Mb besloeg, wat ongeveer de helft van de suikerriet-BAC-sequenties vertegenwoordigde (Wang et al., 2010). Het is duidelijk dat er deleties / inserties bestonden tussen sorghum en Saccharum, en het gebruik van het sorghumgenoom als referentie kan de unieke kenmerken van het Saccharum-genoom niet vervangen en kan dus onvoorspelbare FISH-resultaten veroorzaken.
Sommige FISH-dia’s hadden sterke achtergrondruis, die het gevolg kan zijn van kleine gebieden van herhaalde sequenties (zoals SSR-sequenties) in de BAC-kloon, die bijgevolg interferentie produceren tijdens hybridisatie. Het sorghumgenoom heeft een herhalingsgehalte van ongeveer 61% (Paterson et al., 2009), terwijl ongeveer de helft van het genoom bestaat uit herhalingssequenties in Saccarrum-hybriden (De et al., 2014). In deze herhaalrijke genomen is het moeilijk om signaalspecifieke FISH-probes te ontwikkelen, die chromosomen cytogenetisch onderscheiden met vergelijkbare morfologieën. CISS met het blokkeren van DNA C ° t-100 kan herhaalde sequenties efficiënt uitsluiten. In deze studie werd het blokkeren van DNA C ° t-100 gebruikt voor FISH-analyse van de BAC-sondes in groepen II en III (figuren 2D-F), waarvan werd geverifieerd dat ze de chromosoomspecifieke BAC-sondes waren, terwijl de FISH van BAC-sondes veroorzaakte in groepen IV en V sterke achtergrondinterferentie (figuren 2D-F). Het is duidelijk dat er variaties zijn van herhalingssequenties onder de onderzochte BAC-probes. Daarom zou optimalisatie van experimenten nodig zijn voor BAC-FISH-analyse in Saccharum. Zes tot zeven plaatsen werden weergegeven in Groep II, wat veroorzaakt zou kunnen worden door de homologe chromosoomstructuurvariaties, zoals de fragmentschrapping in een of twee homologe chromosomen. In hexaploïde tarwe hybridiseerde BAC 676D4 sterker met de A-genoomchromosomen dan met de B- en D-genoomchromosomen (Zhang et al., 2004). Deze niet-detecteerbare één of twee signalen in de homologe chromosomen kunnen ook worden veroorzaakt door de technische problemen van BAC-FISH voor zoveel chromosomen met een kleine omvang.
In deze studie identificeerden we 27 chromosoomspecifieke BAC- FISH-sondes die overeenkomen met 9 van de 10 sorghumchromosomen (alle behalve Sb05). In een studie van de genetische kaart afgeleid van een kruising tussen S. officinarum en suikerriet cultivar, werd Sb05 samengevoegd met Sb06 in suikerriet (Aitken et al., 2014). In onze studie onthulde een genetische kaart op basis van de F1-populatie van S. spontaneum dat Sb05 in twee segmenten was verdeeld en dat de twee segmenten werden samengevoegd met respectievelijk Sb06 en Sb07 (Zhang en Ming, niet-gepubliceerde gegevens). Onlangs toonde de genetische kaart van een lid van de Andropogoneae, Miscanthus sinensis, aan dat Sb05 een slechte collineariteit heeft met de overeenkomstige koppelingsgroep in M. sinensis (Ma et al., 2012). Evenzo behouden de twee voorouderlijke maïschromosomen ortholoog aan Sb05 het kleinste aantal syntenische orthologen voor sorghumgenen (Schubert en Lysak, 2011). Daarom kan de afwezigheid van de BAC-FISH die overeenkomt met Sb05 wijzen op chromosoomfusie in S. spontaneum.
Sorghum en S. spontaneum divergeerden 12 miljoen jaar geleden (MYA), het basischromosoomgetal werd verlaagd van x = 10 tot x = 8. In Aitken et al. (2014), de HG2 (homologe groep 2) van suikerriet uitgelijnd met Sb05 en Sb06, en HG8 (homologe groep 8) met Sb02 en Sb08, die bewijs leveren voor de fundamentele chromosoomreductie (Aitken et al., 2014). In deze studie hebben we waargenomen dat de sorghumchromosomen Sb02 en Sb08 interchromosomale herschikking hadden in S. spontaneum, zoals aangetoond door het bewijs dat BAC-14 uitgelijnd was met Sb02 en BAC-19 uitgelijnd met Sb08 (Figuur 7). Onze studie leverde het eerste fysieke en cytogenetische bewijs voor de interchromosoomherschikking van sorghum in S. spontaneum. Niet-gepubliceerde gegevens van onze groep onthulden ook dat Sb08 is verdeeld in twee segmenten en samengevoegd zijn met respectievelijk Sb02 en Sb09 in S. spontaneum (Zhang en Ming, niet-gepubliceerde gegevens).De sondes die overeenkomen met Sb09, Sb08, Sb05 en Sb06 zouden verder kunnen worden gebruikt voor het onderzoeken van de interchromosomale gebeurtenissen tussen sorghum en S. spontaneum. S. spontaneum heeft een breed scala aan ploïdie-niveaus (2n = 40-128) (Irvine, 1999). Deze BAC-sondes zouden kunnen worden gebruikt om de interchromosomale herschikking van S. spontaneum met de verschillende polyploïdie-niveaus te bevestigen.
Vanwege het verschillende basis chromosoomgetal tussen sorghum (x = 10) en S. spontaneum (x = 8), waren de sorghum-chromosomen geen een-op-een correspondentie met S. spontaneum. Zoals onze genetische mapping studie hierboven vermeldde, werd Sb08 verdeeld in twee segmenten die samenvloeiden met segmenten van Sb02 en segmenten van Sb09 in S. spontaneum; Sb05 werd opgedeeld in twee segmenten die samengevoegd werden met segmenten van Sb06 en segmenten van Sb07 S. spontaneum. Daarom waren de sondes die overeenkomen met Sb08 en Sb05 niet specifiek voor afzonderlijke S. spontaneum-chromosomen, terwijl de andere sondes die overeenkomen met de andere acht sorghumchromosomen kunnen worden gebruikt als chromosoomspecifieke cytogenetische BAC-FISH-sondes voor S. spontaneum. De geteste BAC-sondes waren dus gebaseerd op de acht sorghumchromosomen (tabel 2) en waren voldoende voor chromosoomidentificatie met behulp van BAC-FISH. Een satellietchromosoom werd gevonden in één S. spontaneum-chromosoom (chromosoom 3) (Ha et al., 1999). Eerder onthulde gelijktijdige FISH dat de signalen van 45S-rDNA zich bevonden op de secundaire vernauwingen van de satellietchromosomen binnen het chromosoom 3 van de in helmknopcultuur afgeleide S. spontaneum-kloon (AP85-361) (Ha et al., 1999). In deze studie werd BAC-2 dat overeenkomt met Sb03 en 45s-rDNA in kaart gebracht op dezelfde locatie van het S. spontaneum-chromosoom, waardoor eerdere bevindingen verder worden ondersteund (Ha et al., 1999). Onze resultaten leverden ook direct bewijs dat chromosoom 3 van S. spontaneum genoemd in de vorige studie homoloog is aan Sb03.
Conclusie
In deze studie ontwikkelden we chromosoomspecifieke BAC’s van S spontaneum als een stap in de richting van de ontwikkeling van een eenvoudige en reproduceerbare methode voor chromosoomidentificatie met behulp van BAC-FISH cytogenetische markers, waarmee de haalbaarheid wordt bevestigd van het isoleren van chromosoomspecifieke BAC’s op basis van het sorghumgenoom om een fysieke kaart van suikerriet te construeren. We leveren ook het eerste cytogenetische bewijs van interchromosomale herschikking tussen sorghum en S. spontaneum. De oprichting van het suikerriet BAC-FISH-technologiesysteem biedt nieuwe kansen en middelen voor moleculair cytogenetisch onderzoek in suikerriet, inclusief karyotype-analyse, genlokalisatie en fysieke kaartconstructie. Deze resultaten zijn essentieel voor de assemblage van het S. spontaneum-genoom dat nodig is voor sequentiebepaling van het hele genoom.
Bijdragen van de auteur
GD, JS en JZ hebben de studie bedacht en de experimenten ontworpen. GD, JS, QZ, JW, QY, RM, KW en JZ voerden de experimenten uit en analyseerden de gegevens. GD en JZ schreven het manuscript. Alle auteurs hebben het definitieve document gelezen en goedgekeurd.
Financiering
Dit project werd ondersteund door subsidies van het 863-programma (2013AA102604), NSFC (31201260), Program for New Century Excellent Talents in Fujian Province University, en de financiering van de Fujian Agriculture and Forestry University.
Verklaring van belangenconflicten
De auteurs verklaren dat het onderzoek is uitgevoerd zonder enige commerciële of financiële relatie dat kan worden opgevat als een mogelijk belangenconflict.
Dankbetuigingen
We danken Dr. Xintan Zhang voor het samenstellen van de BAC-reeksen en Dr. Irene Lavagi voor het redigeren van het Engels.
Aanvullend materiaal
Dhont, A. (2005). Ontrafelen van de genoomstructuur van polyploïden met behulp van FISH en GISH; voorbeelden van suikerriet en banaan. Cytogenet. Genome Res. 109, 27-33. doi: 10.1159 / 000082378
PubMed Abstract | CrossRef Volledige tekst | Google Scholar
Piperidis, G., D’Hont, A., en Hogarth, D. M. (2001). “Chromosoomsamenstellingsanalyse van verschillende Saccharum interspecifieke hybriden door genomic in situ (hybridisatie) (GISH),” in Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD: ASSCT), 565-566.
Google Scholar
