Bevezetés
A cukornád (Saccharum spp., Poaceae) egyéves vagy évelő növény, amelyet világszerte trópusi és szubtrópusi régiókban termesztenek. A cukornád fontos növény a szacharóztermelésben, és a világ cukortermelésének 70% -át adja. Ezenkívül C4 üzemként a cukornád hatékonyan képes a napenergiát kémiai energiává alakítani, ezért ideális bioüzemanyag-termény az etanolgyártáshoz (Lam et al., 2009; Santchurn et al., 2014). A Saccharum nemzetségnek hat faja van: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense és S. edule, amelyek közül a S. spontaneum vadfajnak tekinthető, amelynek alapvető kromoszómahalmaza x = 8. A S. spontaneum a két fő Saccharum faj egyike, amely hozzájárul a modern cukornádfajták fejlődéséhez (Dhont et al., 1998; Ha et al., 1999). A S. spontaneum erős környezeti alkalmazkodóképességgel rendelkezik, és fontos genetikai tulajdonságokat tartalmaz a betegségekkel és az aszályokkal szembeni ellenállóképesség szempontjából, hozzájárulva ezzel a modern fajtahibridek stressztűrő képességéhez (Grivet et al., 2004). A S. spontaneum földrajzi eloszlása a legszélesebb, ploiditási szintje 2n = 5x = 40 és 2n = 16x = 128 között mozog (Panje és Babu, 1960).
A cukornád kromoszómáinak morfológiája hasonló és kicsi 1–6 μm-es méret a kondenzált metafázis szakaszában (Ha et al., 1999; Dhont, 2005); ezért nagyon nehéz megkülönböztetni a különböző kromoszómákat a hagyományos citogenetikai módszerek alapján. Sőt, mint autopolyploid, kromoszómális szerkezeti változásai, amelyeket a poliploidizáció, a duplikáció, a deléció és a rekombináció okoz, nagyon gyakoriak (Piperidis et al., 2010). Az S. spontaneumban genetikai kötöttségi térképeket dolgoztak ki (Silva et al., 1995), de egy citológiai genetikai térkép kidolgozása elmaradt a többi füvfajtól. Az egyes kromoszómák azonosítására megbízható citológiai eszközök használhatók a hatékony genomkutatáshoz és a csíraplasma-erőforrás-felhasználáshoz, különös tekintettel a modern cukornádfajták összetett genom-hátterére. A kromoszómaspecifikus bakteriális mesterséges kromoszóma (BAC) klónok felbecsülhetetlen erőforrást jelentenek a cukornád genomkutatásaiban, és számos alkalmazásuk lesz a Saccharum spp. Fizikai feltérképezésében, kromoszóma-azonosításában és markerekkel segített tenyésztésében, valamint a cukornád genomszekvenálásának és összeszerelésének projektjeiben. .
A fluoreszcencia in situ hibridizáció (FISH) a molekuláris citológia hatékony eszköze (Jiang és Gill, 2006). Saccharum spp.-ben 45S rDNS-t és 5S rDNS-t alkalmaztunk próbaként a S. spontaneum, S. robustum és S. officinarum alapvető kromoszómáinak kimutatására, a különböző bázisos kromoszómák azonosítására, x = 8 az S. spontaneumban és x = 10 in S. robustum és S. officinarum (Dhont et al., 1998; Ha et al., 1999). A FISH alapján megállapították, hogy a modern cukornádfajták a S. officinarumból származó kromoszómák 70–80% -át, a S. spontaneum kromoszómáinak 10–23% -át tartalmazzák, míg 5–17% a közötti rekombináció terméke. S. spontaneum és S. officinarum (Dhont és mtsai, 1996; Piperidis és mtsai, 2001; Cuadrado és mtsai, 2004). Ezenkívül a Saccharum és az Erianthus arundinaceus közötti hibridizációból származó FISH segítségével kromoszóma eliminációs, rekombinációs és transzlokációs eseményeket detektáltak (Dhont et al., 1995; Huang et al., 2015).
Cirok kicsi diploid genomja van (730 Mbp), a kromoszóma rekombinációs eseményei alacsony gyakorisággal bírnak. A cirok magas szineténiát mutat a cukornád genomjával, így ideális referencia növény a cukornád genommal való összehasonlító elemzésekhez (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ). Ebben a tanulmányban a magas lefedettségű BAC erőforrásokhoz rendelkezésre álló szekvenciák alapján megbízható BAC próbákat fejlesztettek ki a FISH kromoszóma azonosítására és a citogenetikai térkép felépítésére. A vizsgálat céljai a következők voltak: (1) kromoszómaspecifikus BAC-FISH próbák kidolgozása S. spontaneum kromoszómán és (2) a S. spontaneum és a cirok közötti kromoszóma-átrendeződés feltárása.
Anyagok és módszerek
Anyagok
Saccharum spontaneum SES208-at (2n = 8x = 64) alkalmaztunk citológiai elemzésekhez ebben a vizsgálatban. A SES208 növényeket a Fujian Mezőgazdasági és Erdészeti Egyetem (Fuzhou, Kína) campusán termesztették 2015 februárjában, és rendszeres cukornád-növekedési körülmények között tartották fenn.
A haploid genomból BAC könyvtárat készítettek S. spontaneum AP85-441 (4x = 32), amelyet S. spontaneum SES208-ból (2n = 8x = 64) származtattunk in vitro in vitro tenyészeten keresztül. Az AP85-441 genommérete körülbelül 3,2 Gbp. A könyvtár 38 400 klónból állt, átlagos inzertmérete 100 kb, amely a teljes genom 6x-át fedte.Ezt a BAC könyvtárat használták a szacharóz transzporterek és a fruktokináz gén családok azonosítására az S. spontaneumban (Zhang és mtsai., 2016; Chen és mtsai., 2017).
A BAC könyvtár szűrése
Az AP85-441 könyvtárakból 35 156 BAC klónt 701 könyvtárba egyesítettünk. Minden könyvtár átlagosan 50 BAC klónt tartalmaz. A DNS-könyvtárakat a PhasePrepTM BAC DNS Kit segítségével (Sigma, Egyesült Államok) készítettük a gyártó protokolljainak betartásával. A BAC DNS könyvtárakat az Illumina HiSeq 2500 platform és PE250 modell segítségével szekvenáltuk. Összesen 686 könyvtárat (18 könyvtárat nem sikerült szekvenálni) szekvenáltunk, és 267,5 Gb megtisztított adatot állítottunk elő a Trimmomatic 0,36-os verziójával végzett vágás után (Bankevich et al., 2012). Az egyes BAC-állományokból származó adatokat a SPAdes 3.09-es verzióval állítottuk össze alapértelmezett paraméterrel. Összesen 2.611.145 kontigot állítottunk össze 7,38 kbp N50 kontigggel (Zhang és Ming, publikálatlan adatok).
Az AP85-441 BAC könyvtár szekvenciákat BLAST-on kerestük az 1e E értékű cirok genomjával szemben. -4. Azokat a szekvenciákat, amelyeknek egyetlen BLAST találata volt a cirok genomjában, FISH jelölt próbának választottuk. Az alacsony kópiaszámú BAC klónok azonosítására a BAC könyvtárból szekvencia-specifikus primereket terveztek a Primer 5.0 szoftver segítségével annak lehetővé tétele érdekében, hogy a BAC készleteket speciálisan PCR-rel szkríneljék. 18-25 bp alapozóhosszakat, 55-65 ° C Tm értékeket, valamint 40-60% citozint és guanint tartalmazó nukleotid kompozíciókat választottunk. Kétlépcsős PCR-módszert alkalmaztunk a BAC könyvtár 3D dimenziós készletének átvilágítására (Asakawa et al., 1997; Crooijmans et al., 2000), némi módosítással. A 384-lyukú lemez minden egyes klónját tenyésztettük 80 μl Lysogeny-táptalajban + 34 mg / ml kloramfenikolban egy éjszakán át 37 ° C-on, 384-lyukú tenyésztőlemezen. Összesen nyolc 384 lyukú lemezt tenyésztettünk összesen 3072 BAC-klónnal. Minden 384-lyukú lemezhez először 16 soros medencét (A – P sor) és 24 oszlop-medencét (1–24. Oszlop) állítottunk fel minden egyes 384-lyukú lemezhez úgy, hogy az egyes klónok azonos mennyiségű tenyészetét összekevertük, majd összekevertük a 16 soros és 24 oszlopos medence, mindegyik sorból és oszlop medencéből egyenlő térfogattal, mint egy lemezes pool vagy superpool. Ebben a tanulmányban nyolc 384 lyukú lemez szűrésére összpontosítottunk. Így nyolc szuperpoolot készítettek külön a nyolc lemezhez. A nyolc lemez pozitív 384 lyukú lemezét azonosítottuk, majd a 16 keverék pozitív sorát átvilágítottuk. Végül a PCR pozitív oszlopokat 24 oszlopkeverék közül választottuk ki. Huszonnégy PCR-reakciót hajtottunk végre a nyolc 384-lyukú lemez pozitív klónjainak azonosítására.
A PCR-amplifikációkat az előzőekben leírtak szerint hajtottuk végre (Bouzidi et al., 2006). Mindegyik reakció 7,5 μl rTaq premixet, minden egyes primer 0,6 μl-jét (10 μM), 1,0 μl baktérium-folyadék templátot tartalmazott, 20 μl végtérfogatig. A kezdeti denaturálást 95 ° C-on 5 percig, 35 ciklust 95 ° C-on 30 másodpercig, 55 ° C-on 30 másodpercig és 72 ° C-on 1 percig. A PCR-termékeket 1,5% -os agarózgélen analizáltuk.
Metafázisos kromoszóma-előkészítés
A kromoszóma-előkészítést a korábban leírtak szerint végeztük (Lou et al., 2010), kisebb módosításokkal. Röviden, az S. spontaneum növényeket üvegházi körülmények között termesztették a gyökérhegyek betakarításához. Az 1-2 cm körüli kivágott gyökércsúcsokat 0,002 mol / l 8-hidroxi-kinolinnal kezeltük szobahőmérsékleten 2–4 órán át, vízben 15 percig öblítettük, és legalább 24 órán át etanol: ecetsavban (3: 1) rögzítettük. h szobahőmérsékleten. A gyökércsúcsokat ezután enzimoldatban (4% R-10 cellulóz és 2% pektoláz 0,1 M citrátpufferben) emésztettük 37 ° C-on 1 órán át, jeges ionmentes vízzel mostuk 30 percig, végül etanolban inkubáltuk: ecetsavat (3: 1) kb. 30 percig. A tárgylemezeket “lángon szárított” módszerrel állítottuk elő (Iovene és mtsai., 2008).
BAC DNS tisztítása és szonda jelölése
A BAC DNS-t PhasePrepTM BAC-mal extraháltuk DNS-készlet a gyártó kézikönyve szerint. A tisztított BAC DNS-t standard nick transzlációs reakcióval jelöltük, beleértve hígított DNáz I-t, 10 × nick transzlációs puffert, DNS Polymerase I-t, dNTP-ket, biotinnal / digoxigeninnel jelölt dUTP-ket és BAC DNS-t. 15 ° C-on 1,5 órán át inkubáltuk. A kivágott termékeket 1,5% -os agarózgélen megvizsgáltuk 300 és 500 bázispár közötti kenet jelenlétére. A kapott próbákat felhasználásig -20 ° C-on tároltuk. >
Helyi hibridizáció és detektálás
Szonda keverék és hibridizáció: Először a próbaelegyet (50% ioncserélt formamid, 2 × SSC, 80 ng digoxigeninnel / biotinnal jelölt DNS, 10% dextrán-szulfát , > 1 μg C ° t-100) egy 90 ° C-os forró blokkba helyeztük 5 percre, majd azonnal jégre helyeztük, amíg készen álltunk a hibridizálásra tovább. Az előzőleg előkészített, lángon szárított tárgylemezeket 70% -os ionmentes formamiddal kezeltük, amelyet 1 órán át 80 ° C-on hőblokkon denaturáltunk.5 percig, azonnal egymás után jéghideg 70% -os etanolban, 90% -os etanolban, majd egyenként 100% -os etanolban 5 percig, majd levegőn szárítottuk a padon. Végül minden tárgylemezhez denaturált próbákat adtunk, és 24 × 32 mm-es fedőlemezekkel borítottuk be. A tárgylemezeket egy éjszakán át nedves kamrába helyeztük 37 ° C-on.
Szonda detektálása: a fedőlemezeket eltávolítottuk, és a tárgylemezeket szobahőmérsékleten 2x SSC-ben 5 percig, 42 ° C-on 2x-en mostuk. SSC 10 percig és szobahőmérsékleten 1x PBS-ben 5 percig ebben a sorrendben. Biotinnal jelzett szonda jeleket 2 mg / ml Alexa Fluor 488 sztreptavidinnel detektáltunk, digoxigeninnel jelzett szonda jeleket juhok 2% anti-digoxigenin-rodaminjával. Az antitest koktélt (100 μl TNB puffer, 1 μl Alexa Fluor 488 sztreptavidin, 1 μl rodamin anti-dig-juh) adtunk a tárgylemezekhez, amelyeket 24 × 32 mm-es fedőlemezekkel borítottak, 1 órán át 37 ° C-on inkubáltunk nedves kamrát, majd szobahőmérsékleten 1x PBS-ben háromszor 5 percig mossuk. A felesleges folyadékot eltávolítottuk, és a Vectashield (Vector, Burlingame, CA, Egyesült Államok) elhalványult oldatába 4′-6-diamidino-2-fenil-indolt (DAPI) (Sigma, St. Louis, MO, Egyesült Államok) adtunk hozzá. kromoszómák. A képeket Olympus BX63 epifluoreszcens mikroszkóppal rögzítettük. A FISH szignálképeket a CellSens Dimension szoftver segítségével elemeztük.
Eredmények
Alacsony példányszámú BAC klónok szűrése
Azok a BAC klónok kiszűrése, amelyek potenciálisan alacsony példányszámmal rendelkeznek szekvenciákat a S. spontaneum genomban, a BLAST a szekvenált AP85-441 BAC könyvtárakat kerestük a cirok genomjával szemben. Összesen 2000 BAC-szekvenciát szkríneltünk, amelyek megfelelő kollinearitású 10 cirok-kromoszómának felelnek meg. A 2000-es AP85-441 BAC szekvenciákat a REPEATMASKER maszkolta a nagy ismétlődésű cirok DNS adatbázisával és a TIGR gramineae adatbázissal az ismétlődő szekvenciák szűrésére. Végül 114 cirokot osztottunk ki 10 cirok-kromoszómán a FISH-analízishez (1. kiegészítő táblázat), a 10 cirok-kromoszóma mindegyikén 7–16 BAC-t (1. ábra). A pozitív BAC klónok szűrésére 114 specifikus primert terveztek a BAC klón szekvenciák alapján (2. kiegészítő táblázat), és 49 pozitív BAC klónt szkríneltek a BAC könyvtárakból (3. kiegészítő táblázat).
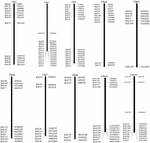
1. ÁBRA 114 APAC-441 BAC-klón eloszlása a cirok kromoszómáiban. A BAC1-114 a BAC számot jelöli, a jobb oldali adatok a BAC fizikai elhelyezkedését jelentik a cirok kromoszómák megfelelő régiójában. Unit = bp.
BAC-FISH jelerősség és eloszlás
A 49 pozitív mintából izolált DNS-minták A BAC klónokat biotinnal vagy digoxigeninnel jelöltük. 5S rDNS és 45S rDNS próbákat használtunk kontrollként, és hibridizáltuk S. spontaneum szomatikus metafázis kromoszómákkal a FISH eljárást követve. A jelerősség és a BAC szekvenciák ismételt szekvencia tartalmának változása közötti összefüggés miatt C ° t-100-at használtunk versenyzőként (1. táblázat). Az erős és stabil hibridizációs jeleket mutató BAC klónokat választottuk ki a további FISH elemzéshez.

1. TÁBLÁZAT 27 pozitív klón osztályozása blokkoló DNS-sel.
Összesen 64 különböző kromoszóma figyelhető meg a S. spontaneum metafázisában. Mindegyik BAC-FISH-t négy független kísérletben (vagy tárgylemezen) végeztük. Mindegyik tárgylemezen legalább 10 szomatikus metafázisos kromoszóma terjedését elemeztük. Az eredmények azt mutatták, hogy a 27 specifikus próba (4. kiegészítő táblázat) öt csoportba sorolható a hibridizációs jel alapján (1. táblázat). Az I. csoportban (beleértve a BAC-18, BAC-20, BAC-29 és BAC-84) a próbák nyolc megkülönböztető helyet mutattak ki versenyképes in situ szuppresszió (CISS) nélkül C ° t-100 alkalmazásával (2A. Ábra), ami azt sugallja, hogy kevés ismételt szekvenciák léteztek ezekben a BAC szekvenciákban. A II. Csoportban (beleértve a BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 és BAC-74), C ° t -100-at alkalmaztunk versenytársként a BAC próba hibridizációjához, és hat-hét egészen különböző helyet mutattunk ki ennek a csoportnak a kariotípusaiban (2B. Ábra). A hibridizációs eredmény hasonló volt a hét szignálhellyel rendelkező 45S rDNS-hez, amelyet a homológ kromoszóma kromoszómaszerkezetének változása okozhat. A III. Csoportban (beleértve a BAC-26, BAC-66, BAC-76 és BAC-78) a C ° t-100 DNS-t használták versenyzőként, és nyolc különálló helyet lehetett megfigyelni a kariotípusban, ami arra utal, hogy ez a BAC csoport a klónok ismétlődő DNS-szekvenciákkal rendelkeznek (2C. ábra). A IV. Csoportban (beleértve a BAC-24-et is) több mint nyolc szignálhelyet figyeltek meg a kariotípusban, és a pericentromerikus és telomerikus régiókban jelentek meg (2D. Ábra), ami arra utal, hogy ezeknek a BAC próbáknak ismétlődő szekvenciájuk volt a S. spontaneumban.Az V. csoportban (beleértve a BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 és BAC-81) a próbák szétszórt jeleket mutattak a korlátozott régiójú kromoszómákban specifikus jeleket megjelenítő kromoszómákból (2E. ábra). A hibridizációs eredmény azt mutatta, hogy a BAC-klónok ezen csoportja nagy mennyiségű ismétlődő DNS-t tartalmazott a S. spontaneumban, amit a cirok és az S. spontaneum hasadása után az ismétlődő régiók gyors evolúciós divergenciája okozhat. Ezért a 49 BAC próba közül az I, II és III csoport 19 BAC szondája (3. táblázat) mutatott specifikus jeleket az S. spontaneum kromoszómákon, amelyek az Sb05 mellett kilenc cirokromoszómának felelnek meg. A másik 22 szonda jelei szétszóródtak az összes kromoszómában. 19 BAC próbát választottunk ki S. spontaneum kromoszómaspecifikus szignálként (3. ábra). A 19 BAC szonda közül egy szonda az Sb02, Sb04 és Sb09 helyeken található; két szonda az Sb07, Sb08 és Sb10 készülékeken; három szonda az Sb01 és Sb06-on; és négy szonda az Sb03-on. Ez a szondakészlet hasznos eszközt nyújtott a S. spontaneum és a cirok összehasonlító elemzéséhez (2. táblázat).
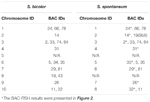
2. TÁBLÁZAT A BAC-FISH próbák eloszlása az S. bicolor és az S. spontaneum kromoszómáiban.
S. spontaneum kromoszómákra specifikus BAC könyvtár létrehozása
Az Sb01 és az Sb02 a két legnagyobb cirok kromoszóma. A cirok és az S. spontaneum közötti genom szintenziójának kimutatásához 10 kromoszómaspecifikus BAC klónt választottunk ki az I., II. És III. Csoportból. Ebből a 10 BAC próbából három szonda (BAC klón azonosító: 24, 66, 78) homológ volt az Sb01-gyel, hét próba (BAC klón azonosító: 14, 18, 20, 69, 71, 73, 77) pedig az Sb02-nek felelt meg. A FISH kétszínű detektálását alkalmaztuk a komplementeren jelölt BAC-párokhoz, a próbákat pedig a BAC-klónok lánc-citogenetikai kapcsolatának feltérképezéséhez. Ezzel elkészítettük az S. spontaneum citogenetikus térképét a 10 BAC próba alapján.
Három Sb01-nek megfelelő próbánál az S. spontaneum FISH-analízise (SES208) kimutatta, hogy a BAC-24 és a BAC -78 próba főleg az S. spontaneum 1. kromoszóma disztális régiójában található, és a BAC-66-ot a centromer régió közvetlen közelében térképeztük fel (4. ábra). A BAC-24 és a BAC-66 ugyanazon kromoszómán található (S. spontaneum 1. kromoszóma); ugyanakkor a 66-os és a 78-as szonda szintén ugyanazon a kromoszómán helyezkedik el, bizonyítva, hogy a három BAC-próba ugyanazokra a kromoszómákra oszlik a S. spontaneum-ban, ami arra utal, hogy az 1. kromoszóma esetében a S. spontaneum és a cirok közötti szintenzia alakul ki. A FISH eredmények azt mutatták, hogy a hét BAC-próba az Sb02-hez igazítva; a hét BAC-ból csak a 69., 71., 73. és 77. szonda generált intenzív jeleket egy S. spontaneum kromoszómán (SsChr2) (5. ábra); míg a 14., 18. és 20. próba nem volt kimutatható a S. spontaneum 2. kromoszómáján, de más S. spontaneum kromoszómákon észlelték. Ezek az eredmények azt mutatták, hogy a S. spontaneum és a cirok között a megfelelő Sb02-ben kromoszóma-átrendeződések következtek be.
Ezenkívül a BAC-2 szekvenciája nagy hasonlóságot mutat (97%) a 3. kromoszóma fragmentumához cirok. Kettős színű FISH elemzés azt mutatta, hogy mind a BAC-2, mind a 45s rDNS-t az S. spontaneum kromoszóma azonos helyére térképezték fel (6. ábra).
Kromoszóma-átrendeződés az Sb02-en a cirok és a S. spontaneum között
Az Sb02-en található kromoszóma-átrendeződés további vizsgálata a cirok és a S. spontaneum között, a kromoszóma az Sb02-nek (BAC-14, BAC-18 és BAC-20) és az Sb08-nak (BAC-19 és BAC-43) megfelelő specifikus próbákat használtunk a FISH elemzéshez. Az eredmények azt mutatták, hogy a BAC-14-et és a BAC-19-et azonos S. spontaneum kromoszómák különböző karjaihoz térképezték fel, jelezve, hogy a S. spontaneumban és a cirokban a 2. és 8. kromoszóma között kromoszóma-átrendeződések történtek (7. ábra).
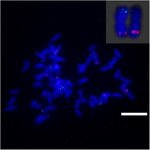
7. ÁBRA A kromoszóma átrendeződése cukornádban és cirokban, amint azt a FISH feltárta. Biotinnal (zöld) és digoxigeninnel (piros) jelölt BAC-14 és BAC-19 próbák. Méretarány = 10 μm.
Megbeszélés
A Saccharum egy összetett nemzetség, amelyet magas poliploidiumszint jellemez, kis kromoszómákkal (Ha et al., 1999; Dhont, 2005). Az egyes Saccharum kromoszómák morfológiájuk alapján történő megkülönböztetése nagyon kihívást jelent. Kihasználva azokat a szekvenciákat, amelyeket a S. spontaneum BAC könyvtárának nagy kiterjedése fed le, képesek voltunk a S. spontaneum potenciálisan alacsony kópiaszámú BAC klónjait kiszűrni a cirok genomjának felhasználásával. Magas poliploid fajként a S. spontaneum több genom-átrendeződést tartalmazhat, mint a diploidok.A S. spontaneum és a cirok közötti genomok különbségei okozhatják a pontatlanságot az alacsony kópiaszámú BAC szekvenciák előrejelzésében a S. spontaneum genomban. Ebben a tanulmányban kezdetben 114 potenciális BAC szekvenciát nyertünk egy példányban a cirok genomja alapján. A részleges BAC könyvtárakban azonban csak 49-et sikerült azonosítani, és 27-ről kiderült, hogy kromoszómaspecifikus BAC-FISH próbák. Legalább két oka van ezen eredmények magyarázatának. Először is, a 114 BAC szekvencia a BAC inszerció részleges fragmensei voltak az NGS szekvencia összeállításának korlátai miatt, és tartalmazhatnak olyan ismétlődő szekvenciákat a többi régióban, amelyek nem rendelkeznek szekvencia információval. Ezek a potenciálisan fedetlen ismétlődő szekvenciák félrevezethetik az alacsony példányszámú szekvenciák detektálását szekvencia-összehasonlítással. Másodszor, a közelmúltban a S. spontaneumban végzett poliploidizáció az S. spontaneum és a cirok közötti ismétlődő szekvenciák variációját okozhatja, és így a S. spontaneum nemspecifikus FISH jelét eredményezheti. Egy korábbi tanulmányban 20 BAC szekvenálása cukornád hibridekben 1,45 Mb folytonossági szekvenciákat generált, a cirok genomjához igazodó szekvenciák 0,99 Mb terjedtek ki, ami a cukornád BAC szekvenciák körülbelül felét tette ki (Wang et al., 2010). Nyilvánvalóan törlések / beillesztések voltak a cirok és a Saccharum között, és a cirok genomjának referenciaként történő felhasználása nem pótolhatja a Saccharum genom egyedi jellemzőit, és így előre nem látható FISH eredményeket okozhat.
Egyes FISH diák erősek voltak háttérzaj, amelynek oka lehet a BAC klónban található ismétlődő szekvenciák (például SSR szekvenciák) kis régiói, amelyek következésképpen interferenciát okoznak a hibridizáció során. A cirokgenom megismételt tartalma körülbelül 61% (Paterson et al., 2009), míg a genom körülbelül fele a Saccarrum hibridek ismétlődő szekvenciáiból áll (De et al., 2014). Ezekben az ismétlésben gazdag genomokban nehéz olyan jelspecifikus FISH-próbákat kifejleszteni, amelyek hasonló morfológiával citogenetikusan különböztetik meg a kromoszómákat. A CISS blokkoló DNS-sel C ° t-100 hatékonyan kizárhatja az ismétlődő szekvenciákat. Ebben a tanulmányban a blokkoló DNS C ° t-100 értéket alkalmaztuk a BAC próbák FISH elemzéséhez a II. És III. Csoportban (2D – F. Ábra), amelyekről kiderült, hogy kromoszómaspecifikus BAC próbák, míg a BAC próbák FISH a IV. és az V. csoportban erős háttérinterferenciát okozott (2D – F. ábra). Nyilvánvaló, hogy a vizsgált BAC próbák között vannak ismétlődő szekvenciák variációi. Ezért a kísérletek optimalizálására lenne szükség Saccharumban a BAC-FISH elemzéshez. Hat-hét helyet mutattak ki a II. Csoportban, amelyet a homológ kromoszóma-szerkezet variációi okozhattak, például a fragmens deléciója egy vagy két homológ kromoszómában. A hexaploid búzában a BAC 676D4 erősebben hibridizálódott az A-genom kromoszómáihoz, mint a B- és D-genom kromoszómáihoz (Zhang et al., 2004). Ezeket a homológ kromoszómákban észlelhető egy-két szignál kimutatását a BAC-FISH technikai problémái is okozhatják ennyi kisméretű kromoszómánál.
Ebben a tanulmányban 27 kromoszómaspecifikus BAC- FISH szondák, amelyek a 10 cirok kromoszómából 9-nek felelnek meg (az Sb05 kivételével mindegyik). Az S. officinarum és a cukornádfajta keresztezéséből származó genetikai térkép vizsgálatában az Sb05-öt cukornádban egyesítették az Sb06-tal (Aitken et al., 2014). Vizsgálatunk során az S. spontaneum F1 populációján alapuló genetikai térkép feltárta, hogy az Sb05 két szegmensre volt felosztva, és a két szegmenst egyesítették az Sb06-tal, illetve az Sb07-gyel (Zhang és Ming, publikálatlan adatok). Nemrégiben az Andropogoneae egyik tagjának, a Miscanthus sinensis genetikai térképe kimutatta, hogy az Sb05 kollinearitása gyenge a M. sinensis megfelelő kötési csoportjával (Ma és mtsai, 2012). Hasonlóképpen, az Sb05-re ortológ két ősi kukorica kromoszóma megtartja a legkevesebb szintenikus ortológot a cirok génjeihez képest (Schubert és Lysak, 2011). Ezért az Sb05-nek megfelelő BAC-FISH hiánya kromoszóma-fúzióra utalhat az S. spontaneumban.
A cirok és a S. spontaneum 12 millió évvel ezelőtt szétvált (MYA), az alap kromoszóma száma x-ről csökkent = 10-től x = 8-ig. Aitken és mtsai. (2014), a cukornád HG2-je (2. homológ csoport) az Sb05 és Sb06-hoz, a HG8 (8. homológ csoport) pedig az Sb02 és Sb08-hoz igazodik, bizonyítékot szolgáltatva az alapvető kromoszóma-redukciós eseményre (Aitken et al., 2014). Ebben a tanulmányban azt figyeltük meg, hogy az Sb02 és Sb08 cirok kromoszómák interkromoszomális átrendeződéssel rendelkeztek a S. spontaneumban, amint azt bizonyítják, hogy a BAC-14 illeszkedik az Sb02-hez, és a BAC-19 illeszkedik az Sb08-hoz (7. ábra). Vizsgálatunk az első fizikai és citogenetikai bizonyítékot szolgáltatta a cirok kromoszómák közötti átrendeződésére a S. spontaneumban. Csoportunk nem publikált adataiból az is kiderült, hogy az Sb08 két szegmensre oszlik, és az S. spontaneumban Sb02-gyel és Sb09-gyel egyesült (Zhang és Ming, publikálatlan adatok).Az Sb09, Sb08, Sb05 és Sb06 szondák további felhasználhatók a cirok és az S. spontaneum közötti kromoszomális események vizsgálatára. A S. spontaneum ploiditási szintje széles skálán mozog (2n = 40–128) (Irvine, 1999). Ezekkel a BAC próbákkal igazolható volt a S. spontaneum kromoszómák közötti átrendeződése a különböző poliploidia szintekkel.
A cirok (x = 10) és az S. spontaneum (x = (8), a cirok kromoszómák nem voltak egy az egyben levelezéssel az S. spontaneummal. Amint azt korábban említettük genetikai térképészeti tanulmányunkban, az Sb08 két szegmensre oszlott, amelyek összeolvadtak az S. spontaneum Sb02 és Sb09 szegmenseivel; Az Sb05-et két szegmensre osztottuk, amelyek összeolvadtak az Sb06 és az Sb07 S. spontaneum szegmensekkel. Ezért az Sb08 és Sb05-nek megfelelő próbák nem voltak specifikusak az egyes S. spontaneum kromoszómákra, míg a többi nyolc cirok-kromoszómának megfelelő többi próba használható fel kromoszómaspecifikus citogenetikai BAC-FISH próbaként az S. spontaneum esetében. Így a vizsgált BAC próbák a nyolc cirok kromoszómán alapultak (2. táblázat) elegendőek voltak a kromoszóma azonosításához BAC-FISH alkalmazásával. Szatellit kromoszómát találtak az egyik S. spontaneum kromoszómában (3. kromoszóma) (Ha et al., 1999). Korábban a szimultán FISH feltárta, hogy a 45S rDNS jelei a 3-as kromoszómán lévő műholdas kromoszómák szekunder szűkületein helyezkedtek el a portok tenyészetből származó S. spontaneum klónból (AP85-361) (Ha és mtsai, 1999). Ebben a tanulmányban az Sb03 és 45s rDNS-nek megfelelő BAC-2-t a S. spontaneum kromoszóma azonos helyére térképeztük fel, ezáltal tovább alátámasztva a korábbi eredményeket (Ha et al., 1999). Eredményeink közvetlen bizonyítékot szolgáltattak arra is, hogy az előző vizsgálatban megnevezett S. spontaneum 3. kromoszóma homológ az Sb03-mal.
Következtetés
Ebben a vizsgálatban az S kromoszómaspecifikus BAC-jait fejlesztettük ki. a spontaneum a kromoszóma-azonosítás egyszerű és reprodukálható módszerének kifejlesztése felé, a BAC-FISH citogenetikai markerek alkalmazásával, megerősítve a cirokgenomon alapuló kromoszómaspecifikus BAC-ok izolálásának megvalósíthatóságát a cukornád fizikai térképének elkészítéséhez. Bemutatjuk az első citogenetikai bizonyítékot a cirok és az S. spontaneum közötti kromoszómák közötti átrendeződésre is. A cukornád BAC-FISH technológiai rendszerének létrehozása új lehetőségeket és eszközöket kínál a cukornád molekuláris citogenetikai kutatásaihoz, beleértve a kariotípuselemzést, a gének lokalizációját és a fizikai térképszerkezetet. Ezek az eredmények elengedhetetlenek az egész genom szekvenálásához szükséges S. spontaneum genom összeállításához.
Szerzői hozzájárulás
GD, JS és JZ megfogalmazta a vizsgálatot és megtervezte a kísérleteket. GD, JS, QZ, JW, QY, RM, KW és JZ elvégezték a kísérleteket és elemezték az adatokat. GD és JZ írta a kéziratot. Valamennyi szerző elolvasta és jóváhagyta a záró papírt.
Finanszírozás
Ezt a projektet a 863 program (2013AA102604), az NSFC (31201260), az Új Század Kiváló Tehetségek Programja támogatta. A Fujian tartományi egyetem és a Fujian Mezőgazdasági és Erdészeti Egyetem támogatása.
Összeférhetetlenségi nyilatkozat
A szerzők kijelentik, hogy a kutatást kereskedelmi vagy pénzügyi kapcsolatok hiányában végezték. ez potenciális összeférhetetlenségként értelmezhető.
Köszönetnyilvánítás
Köszönjük Dr. Xintan Zhangnak, hogy összeállította a BAC szekvenciákat, és Dr. Irene Lavaginak az angol szerkesztésért.
Kiegészítő anyag
Dhont, A. (2005). A poliploidok genomszerkezetének feltárása FISH és GISH alkalmazásával; példák cukornádra és banánra. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
PubMed Abstract | CrossRef teljes szöveg | Google Tudós
Piperidis, G., D’Hont, A. és Hogarth, D. M. (2001). “Különböző Saccharum interspecifikus hibridek kromoszómakompozíciójának elemzése genomiális in situ (hibridizáció) (GISH) alapján”, Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD: ASSCT), 565–566.
Google Tudós
