はじめに
Sugarcane(Saccharum spp。、イネ科)は、世界中の熱帯および亜熱帯地域で栽培される一年生または永年性作物です。サトウキビはショ糖生産にとって重要な作物であり、世界の砂糖生産の70%を占めています。さらに、C4植物として、サトウキビは太陽エネルギーを化学エネルギーに効率的に変換できるため、エタノール生産に理想的なバイオ燃料作物です(Lam et al。、2009; Santchurn et al。、2014)。サトウキビ属には、S。spontaneum、S。robustum、S。officinarum、S。barberi、S。sinense、およびS. eduleの6種があり、そのうちS.spontaneumは基本染色体セットがxの野生種と見なされます。 = 8. S. spontaneumは、現代のサトウキビ栽培品種の開発に貢献している2つの主要なSaccharum種の1つでもあります(Dhont et al。、1998; Ha et al。、1999)。 S. spontaneumは強い環境適応性を持ち、病気や干ばつ耐性のための重要な遺伝的特徴を含んでいるため、現代の栽培品種ハイブリッドのストレス耐性に貢献しています(Grivet et al。、2004)。 S. spontaneumは地理的分布が最も広く、倍数性レベルは2n = 5x = 40から2n = 16x = 128の範囲です(Panje and Babu、1960)。
サトウキビ染色体は類似した形態を持ち、小さい凝縮した中期段階での1〜6μmのサイズ(Ha et al。、1999; Dhont、2005);したがって、従来の細胞遺伝学的手法に基づいて異なる染色体を識別することは非常に困難です。さらに、自己倍数体として、倍数体化、重複、欠失、および組換えによって引き起こされるその染色体構造の変化は非常に一般的です(Piperidis et al。、2010)。 S. spontaneumでは、遺伝連鎖地図が開発されたが(Silva et al。、1995)、細胞学的遺伝子地図の開発は他の草種に遅れをとった。個々の染色体を特定するための信頼性の高い細胞学的ツールは、特に現代のサトウキビ栽培品種の複雑なゲノム背景を使用して、効果的なゲノム研究と遺伝資源の利用に使用できます。染色体特異的細菌人工染色体(BAC)クローンは、サトウキビゲノム研究にとって非常に貴重なリソースであり、物理マッピング、染色体同定、Saccharum spp。のマーカー支援育種、およびサトウキビゲノム配列決定とアセンブリプロジェクトの支援に多くの用途があります。 。
蛍光insituハイブリダイゼーション(FISH)は、分子細胞学のための強力なツールです(Jiang and Gill、2006)。サトウキビ属では、45SrDNAと5SrDNAをプローブとして使用して、S。spontaneum、S。robustum、およびS. officinarumの基本染色体を検出し、さまざまな基本染色体を識別しました。S。spontaneumではx = 8、x = S.robustumおよびS.officinarumでは10(Dhont et al。、1998; Ha et al。、1999)。 FISHに基づくと、現代のサトウキビ栽培品種には、S。officinarumに由来する染色体の70〜80%、およびS. spontaneumに由来する染色体の10〜23%が含まれていることがわかりましたが、5〜17%はS.spontaneumおよびS.officinarum(Dhont et al。、1996; Piperidis et al。、2001; Cuadrado et al。、2004)。さらに、FISHを使用したSaccharumとErianthus arundinaceusのハイブリダイゼーションに由来するいくつかの前駆細胞で、染色体の除去、組換え、および転座のイベントが検出されました(Dhont et al。、1995; Huang et al。、2015)。
Sorghum染色体組換えイベントの頻度が低い小さな二倍体ゲノム(730 Mbp)を持っています。ソルガムはサトウキビゲノムと高いシンテニーを共有しているため、サトウキビゲノムとの比較分析に理想的な参照植物となっています(Ming et al。、1998; Nazeema et al。、2007; Wang et al。、2010; Aitken et al。、2014 )。この研究では、高カバレッジBACリソースの利用可能なシーケンスに基づいて、信頼性の高いBACプローブがFISH染色体の識別と細胞遺伝学的マップの構築のために開発されました。この研究の目的は、(1)S。spontaneum染色体上に染色体特異的BAC-FISHプローブのセットを開発し、(2)S.spontaneumとソルガムの間の染色体再配列を調査することでした。
材料および方法
材料
この研究では、細胞学的分析にSaccharum spontaneum SES208(2n = 8x = 64)を使用しました。 SES208植物は、2015年2月に福建農林大学(福州、中国)のキャンパスの野外で栽培され、通常のサトウキビ栽培条件下で維持されました。
BACライブラリーは一倍体ゲノムから構築されました。 S. spontaneum SES208(2n = 8x = 64)から葯invitro培養を介して誘導されたS.spontaneum AP85-441(4x = 32)の。 AP85-441のゲノムサイズは約3.2Gbpです。ライブラリーは38,400クローンで構成され、平均インサートサイズは100 kbで、ゲノム全体の6倍をカバーしています。このBACライブラリーは、S。spontaneumのスクローストランスポーターとフルクトキナーゼ遺伝子ファミリーを特定するために使用されています(Zhang et al。、2016; Chen et al。、2017)。
BACライブラリーのスクリーニング
AP85-441ライブラリからの35,156個のBACクローンが701個のライブラリにプールされました。各ライブラリには、平均50個のBACクローンが含まれています。 DNAライブラリーは、PhasePrepTM BAC DNAキット(Sigma、米国)を使用して、製造元のプロトコルに従って調製しました。 BAC DNAライブラリは、PE250モデルを備えたイルミナHiSeq2500プラットフォームを使用してシーケンスされました。合計686のライブラリ(18のライブラリがシーケンスに失敗しました)がシーケンスされ、Trimmomaticバージョン0.36(Bankevich et al。、2012)を使用してトリミングした後、267.5Gbのクリーンなデータが生成されました。各BACプールからのデータは、デフォルトのパラメーターでSPAdesバージョン3.09を使用してアセンブルされました。合計2,611,145個のコンティグが7.38kbpのコンティグN50でアセンブルされました(Zhang and Ming、未発表データ)。
AP85-441 BACライブラリー配列は、E値1eのソルガムゲノムに対してBLAST検索されました。 -4。ソルガムゲノムに単一のBLASTヒットがあった配列をFISHプローブ候補として選択しました。 BACライブラリーから低コピー数のBACクローンを特定するために、Primer 5.0ソフトウェアを使用して配列特異的プライマーを設計し、BACプールをPCRで特異的にスクリーニングできるようにしました。 18〜25 bpのプライマー長、55〜65°CのTm値、および40〜60%のシトシンとグアニンのヌクレオチド組成が選択されました。 2段階のPCR法を使用して、BACライブラリの3D次元プールをスクリーニングしました(Asakawa et al。、1997; Crooijmans et al。、2000)。 384ウェルプレートの各クローンを、80μlの溶原性ブロス+ 34 mg / mlクロラムフェニコールで、384ウェル培養プレートで37℃で一晩培養しました。合計で、合計3072個のBACクローンを含む8枚の384ウェルプレートを培養しました。 384ウェルプレートごとに、最初に各クローンの等量の培養物を混合することにより、384ウェルプレートごとに16行プール(行AからP)と24列プール(列1〜24)を構築し、次に16行プールと24列プール、および各行と列プールからの等しいボリュームを1つのプレートプールまたはスーパープールとして。この研究では、8枚の384ウェルプレートのスクリーニングに焦点を当てました。したがって、8つのスーパープールが8つのプレート用に別々に準備されました。 8枚のプレートからの陽性の384ウェルプレートを特定し、続いて16種類の混合物の中の陽性の列をスクリーニングした。最後に、PCR陽性カラムを24カラム混合物から選択しました。 8つの384ウェルプレートから陽性クローンを同定するために24のPCR反応を実施しました。
PCR増幅は以前に記載されたように実施しました(Bouzidi et al。、2006)。各反応には、rTaqプレミックス7.5μl、0.6μlの各プライマー(10μM)、1.0μlの細菌液体テンプレートが含まれ、最終容量は20μlになりました。 95°Cで5分間の初期変性に続いて、95°Cで30秒間、55°Cで30秒間、72°Cで1分間の35サイクルを実行しました。 PCR産物を1.5%アガロースゲルで分析しました。
中期染色体の調製
染色体の調製は、以前に説明したように(Lou et al。、2010)、わずかな変更を加えて行いました。手短に言えば、S。spontaneum植物は、根端収穫のために温室条件で育てられた。切除した根の先端約1〜2 cmを、0.002 mol / lの8-ヒドロキシキノリンで室温で2〜4時間処理し、水で15分間すすぎ、エタノール:酢酸(3:1)で少なくとも24時間固定しました。室温でh。次に、根の先端を酵素溶液(0.1 Mクエン酸緩衝液中の4%セルロースR-10および2%ペクトリアーゼ)で37°Cで1時間消化し、氷脱イオン水で30分間洗浄し、最後にエタノール中でインキュベートしました。酢酸(3:1)で約30分間。スライドは「火炎乾燥」法(Iovene et al。、2008)を使用して調製しました。
BACDNAの精製とプローブの標識
BACDNAはPhasePrepTMBACで抽出しました。製造元のマニュアルに従ったDNAキット。精製されたBACDNAは、希釈DNase I、10×ニックトランスレーションバッファー、DNA Polymerase I、dNTP、ビオチン/ジゴキシゲニン標識dUTP、BACDNAなどの標準的なニックトランスレーション反応によって標識されました。 15°Cで1.5時間インキュベートしました。次に、切断した生成物を1.5%アガロースゲルで300〜500 bpのスミアの存在について調べました。得られたプローブは、使用するまで-20°Cで保存しました。
InSituハイブリダイゼーションと検出
プローブミックスとハイブリダイゼーション:最初に、プローブ混合物(50%脱イオンホルムアミド、2×SSC、80 ngジゴキシゲニン/ビオチン標識DNA、10%デキストラン硫酸、>1μgC°t-100)を90°Cのホットブロックに5分間入れ、ハイブリダイズをプローブする準備ができるまですぐに氷上に移しました。オン。以前に調製した火炎乾燥スライドを70%脱イオン化ホルムアミドで処理し、ヒートブロック上で80°Cで1分間変性させました。5分間、すぐに氷冷した70%エタノール、90%エタノール、100%エタノールにそれぞれ5分間連続して浸漬し、ベンチで風乾しました。最後に、変性プローブを各スライドに追加し、24×32mmのカバースリップで覆った。スライドを37℃の湿室に一晩置いた。
プローブ検出:カバーガラスを取り外し、スライドを室温で2×SSCで5分間、42℃で2×で洗浄した。このシーケンスで、SSCを10分間、室温で1×PBSで5分間。ビオチン標識プローブシグナルは2mg / ml Alexa Fluor 488ストレプトアビジンで検出され、ジゴキシゲニン標識プローブシグナルはヒツジ由来の2%抗ジゴキシゲニン-ローダミンで検出されました。抗体カクテル(100μlTNBバッファー、1μlAlexaFluor488ストレプトアビジン、1μlローダミン抗発掘羊)をスライドに加え、24×32 mmカバースリップで覆い、37°Cで1時間インキュベートしました。湿ったチャンバーに入れ、室温で1×PBSで3回、それぞれ5分間洗浄した。過剰な液体を除去し、退色防止溶液Vectashield(Vector、Burlingame、CA、United States)中の4 ‘、6-diamidino-2-phenylindole(DAPI)(Sigma、St。Louis、MO、United States)を添加して、染色体。画像はオリンパスBX63落射蛍光顕微鏡で撮影されました。 CellSensDimensionソフトウェアを使用してFISHシグナル画像を分析しました。
結果
低コピーBACクローンのスクリーニング
コピー数が少ない可能性のあるBACクローンをスクリーニングします。 S. spontaneumゲノムの配列について、シーケンスされたAP85-441BACライブラリをソルガムゲノムに対してBLAST検索しました。良好な共線性を持ち、配列特異性を示す10個のソルガム染色体に対応する合計2000個のBAC配列をスクリーニングしました。 2000 AP85-441 BAC配列は、反復配列をフィルタリングするために、高反復ソルガムDNAデータベースおよびTIGRイネ科データベースに対してREPEATMASKERによってマスクされました。最後に、10個のソルガム染色体に分布する114個のBACがFISH分析用に選択され(補足表1)、10個のソルガム染色体のそれぞれに7〜16個のBACがあります(図1)。陽性のBACクローンをスクリーニングするために、BACクローン配列に基づいて114の特異的プライマーを設計し(補足表2)、BACライブラリーから49の陽性BACクローンをスクリーニングしました(補足表3)。
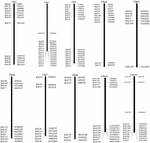
図1.ソルガムの染色体におけるAP85-441からの114個のBACクローンの分布。 BAC1-114はBAC番号を表し、右側のデータは、ソルガム染色体の対応する領域におけるBACの物理的な位置です。単位= bp。
BAC-FISH信号強度と分布
49個の陽性から分離されたDNAサンプルBACクローンはビオチンまたはジゴキシゲニンで標識されました。 5SrDNAおよび45SrDNAプローブをコントロールとして使用し、FISH手順に従ってS.spontaneum体細胞中期染色体にハイブリダイズさせました。シグナル強度とBACシーケンスの繰り返しシーケンスコンテンツの変動との相関関係のため、競合他社としてC°t-100を使用しました(表1)。強力で安定したハイブリダイゼーションシグナルを示すBACクローンを、さらなるFISH分析のために選択しました。

表1.ブロッキングDNAを持つ27個の陽性クローンの分類。
合計64個の異なる染色体を観察できます。 S.spontaneumのメタフェーズで。各BAC-FISHは、4つの独立した実験(またはスライド)で実行されました。各スライドで、体細胞中期染色体の少なくとも10の広がりを分析しました。結果は、27の特定のプローブ(補足表4)がハイブリダイゼーションのシグナル(表1)に基づいて5つのグループに分類できることを示しました。グループI(BAC-18、BAC-20、BAC-29、およびBAC-84を含む)では、プローブは、C°t-100を使用した競合insitu抑制(CISS)なしで8つの特徴的なサイトを表示し(図2A)、繰り返しがほとんどないことを示唆していますこれらのBACシーケンスにはシーケンスが存在していました。グループII(BAC-2、BAC-11、BAC-14、BAC-19、BAC-31、BAC-33、BAC-43、BAC-71、BAC-73、およびBAC-74を含む)では、C°t -100はBACプローブハイブリダイゼーションの競合物質として使用され、このグループの核型では6〜7つのまったく異なる部位が表示されました(図2B)。ハイブリダイゼーションの結果は、7つのシグナル部位を持つ45S rDNAと同様でした。これは、相同染色体の染色体構造の変化によって引き起こされる可能性があります。グループIII(BAC-26、BAC-66、BAC-76、およびBAC-78を含む)では、C°t-100 DNAが競合物質として使用され、核型で8つの異なる部位が観察され、このグループのBACが示唆されました。クローンには反復DNA配列があります(図2C)。グループIV(BAC-24を含む)では、核型で8つ以上のシグナル部位が観察され、セントロメア周辺およびテロメア領域に現れ(図2D)、これらのBACプローブがS.spontaneumで反復配列を持っていることを示唆しています。グループV(BAC-4、BAC-5、BAC-32、BAC-34、BAC-35、BAC-69、BAC-77、およびBAC-81を含む)では、プローブは限られた領域ですべての染色体に分散したシグナルを示しました特定のシグナルを表示する個々の染色体の数(図2E)。ハイブリダイゼーションの結果は、このグループのBACクローンがS. spontaneumに大量の反復DNAを含んでいることを示しました。これは、ソルガムとS.spontaneumの分割後の反復領域の急速な進化的分岐によって引き起こされる可能性があります。したがって、49個のBACプローブのうち、グループI、II、およびIIIの19個のBACプローブのセット(補足表3)は、Sb05以外の9個のソルガム染色体に対応するS.spontaneum染色体上に特定のシグナルを示しました。他の22個のプローブのシグナルはすべての染色体に分散していた。 S. spontaneum染色体特異的シグナルとして19個のBACプローブのセットを選択しました(図3)。 19個のBACプローブのうち、1個のプローブはSb02、Sb04、およびSb09にありました。 Sb07、Sb08、およびSb10の2つのプローブ。 Sb01とSb06の3つのプローブ。 Sb03の4つのプローブ。このプローブセットは、S。spontaneumとソルガムの比較分析に役立つツールを提供しました(表2)。
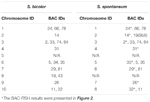
表2.S.bicolorおよびS.spontaneumの染色体におけるBAC-FISHプローブの分布。
S.spontaneum染色体に特異的なBACライブラリーの構築
Sb01とSb02は2つの最大のソルガム染色体です。ソルガムとS.spontaneumの間のゲノムシンテニーを検出するために、グループI、II、およびIIIから10個の染色体特異的BACクローンを選択しました。これらの10個のBACプローブのうち、3個のプローブ(BACクローンID:24、66、78)はSb01と相同であり、7個のプローブ(BACクローンID:14、18、20、69、71、73、77)はSb02に対応しました。相補的に標識されたBACペアのFISHのデュアルカラー検出と、BACクローンの鎖細胞遺伝学的関係をマッピングするためのプローブを利用しました。そうすることで、10個のBACプローブに基づいてS.spontaneumの細胞遺伝学的マップを作成しました。
Sb01に対応する3つのプローブについて、S。spontaneum(SES208)のFISH分析により、BAC-24とBACが示されました。 -78プローブは主にS.spontaneum染色体1の遠位領域に位置し、BAC-66はセントロメア領域のすぐ近くにマッピングされました(図4)。同じ染色体(S. spontaneum染色体1)に位置するBAC-24とBAC-66。同時に、プローブ66とプローブ78も同じ染色体上にあり、3つのBACプローブがS. spontaneumの同じ染色体に分布していることを示しており、S。spontaneumとソルガムの染色体1のシンテニーを示唆しています。FISHの結果から、7つのBACプローブがSb02に合わせて; 7つのBACのうち69、71、73、および77のプローブのみが、1つのS. spontaneum染色体(SsChr2)で強いシグナルを生成しました(図5)。一方、プローブ14、18、および20は、S。spontaneum染色体2では検出できませんでしたが、他のS.spontaneum染色体では観察されました。これらの結果は、S。spontaneumとソルガムの間の対応するSb02で染色体再配列が発生したことを示しています。
さらに、BAC-2の配列は、第3染色体上のフラグメントと高い類似性(97%)を共有していました。ソルガム。 2色FISH分析は、BAC-2と45srDNAの両方がS.spontaneum染色体の同じ位置にマッピングされたことを示しました(図6)。
ソルガムとS.spontaneumの間のSb02の染色体再配列
ソルガムとS.spontaneumの間のSb02の染色体再配列、染色体Sb02(BAC-14、BAC-18、およびBAC-20)に対応し、Sb08(BAC-19およびBAC-43)に対応する特定のプローブをFISH分析に使用しました。結果は、BAC-14とBAC-19が同じS. spontaneum染色体の異なるアームにマッピングされたことを示し、染色体再配列がS. spontaneumとソルガムの染色体2と8の間で起こったことを示しています(図7)。
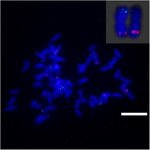
図7.FISHによって明らかにされたサトウキビとソルガムの染色体の再配列。ビオチン(緑)とジゴキシゲニン(赤)で標識されたBAC-14およびBAC-19プローブ。スケールバー=10μm。
ディスカッション
サトウキビは、倍数性レベルが高いことを特徴とする複雑な属です。小さな染色体を持つ(Ha et al。、1999; Dhont、2005)。形態に基づいて個々のサトウキビ染色体を区別することは非常に困難です。 S. spontaneumのBACライブラリーによって高度にカバーされている配列を利用して、ソルガムゲノムを参照として使用して、S。spontaneumの潜在的な低コピー数BACクローンをスクリーニングすることができました。高倍数体種として、S。spontaneumは二倍体よりも多くのゲノム再配列を含む可能性があります。S. spontaneumとソルガムのゲノムの違いは、S。spontaneumゲノムの低コピー数BAC配列の予測における不正確さの原因である可能性があります。この研究では、ソルガムゲノムに基づいたシングルコピーで114の潜在的なBAC配列を最初に取得しました。ただし、部分的なBACライブラリでは49しか識別できず、27は染色体特異的なBAC-FISHプローブであることがわかりました。これらの結果を説明する理由は少なくとも2つあります。第一に、114個のBAC配列は、NGS配列アセンブリの制限により、BAC挿入の部分的な断片であり、配列情報を持たない他の領域に反復配列が含まれている可能性があります。これらの潜在的にカバーされていない反復配列は、配列比較による低コピー配列の検出を誤解させる可能性があります。第二に、S。spontaneumの最近の倍数化は、S。spontaneumとソルガムの間の反復配列の変動を引き起こし、S。spontaneumで非特異的なFISHシグナルをもたらす可能性があります。以前の研究では、サトウキビハイブリッドで20個のBACをシーケンスすると、1.45 Mbのコンティグシーケンスが生成され、ソルガムゲノムと整列するシーケンスは0.99 Mbに及び、サトウキビのBACシーケンスの約半分を占めました(Wang et al。、2010)。明らかに、ソルガムとサッカラムの間に欠失/挿入が存在し、ソルガムゲノムを参照として利用しても、サッカラムゲノムの固有の機能を置き換えることはできないため、予期しないFISH結果が生じる可能性があります。
一部のFISHスライドは強力でしたバックグラウンドノイズ。これは、BACクローンの反復配列(SSR配列など)の小さな領域が原因である可能性があり、その結果、ハイブリダイゼーション中に干渉が発生します。ソルガムゲノムの反復含有量は約61%ですが(Paterson et al。、2009)、ゲノムの約半分はSaccarrumハイブリッドの反復配列で構成されています(De et al。、2014)。これらのリピートが豊富なゲノムでは、類似の形態で細胞遺伝学的に染色体を区別するシグナル特異的FISHプローブを開発することは困難です。 DNAC°t-100をブロックするCISSは、反復配列を効率的に排除できます。この研究では、ブロッキングDNA C°t-100をグループIIおよびIIIのBACプローブのFISH分析に使用し(図2D–F)、染色体特異的BACプローブであることが確認されました。BACプローブのFISHはグループIVおよびVでは、強いバックグラウンド干渉が発生しました(図2D–F)。調べたBACプローブ間で反復配列にばらつきがあることは明らかです。したがって、SaccharumでのBAC-FISH分析には、実験の最適化が必要です。グループIIでは6〜7個の部位が表示されましたが、これは1つまたは2つの相同染色体のフラグメント欠失などの相同染色体構造の変化によって引き起こされる可能性があります。六倍体コムギでは、BAC 676D4はBおよびDゲノム染色体よりもAゲノム染色体に強くハイブリダイズしました(Zhang et al。、2004)。相同染色体におけるこれらの1〜2個のシグナルの検出不能は、サイズが小さい非常に多くの染色体に対するBAC-FISHの技術的問題によっても引き起こされる可能性があります。
この研究では、27個の染色体特異的BAC-を特定しました。 10個のソルガム染色体のうち9個に対応するFISHプローブ(Sb05を除くすべて)。 S. officinarumとサトウキビ栽培品種の交配から得られた遺伝子地図の研究では、Sb05はサトウキビのSb06と統合されました(Aitken et al。、2014)。私たちの研究では、S。spontaneumのF1集団に基づく遺伝子地図により、Sb05が2つのセグメントに分割され、2つのセグメントがそれぞれSb06およびSb07とマージされたことが明らかになりました(ZhangおよびMing、未発表データ)。最近、AndropogoneaeのメンバーであるMiscanthus sinensisの遺伝子地図は、Sb05がM. sinensisの対応するリンケージグループとの共線性が低いことを示しました(Ma et al。、2012)。同様に、Sb05にオーソロガスである2つの祖先のトウモロコシ染色体は、ソルガム遺伝子に最も少ない数のシンテニーオルソログを保持します(Schubert and Lysak、2011)。したがって、Sb05に対応するBAC-FISHがないことは、S。spontaneumの染色体融合を示している可能性があります。
ソルガムとS.spontaneumは1200万年前に分岐し(MYA)、基本染色体数はxから減少しました。 = 10からx =8。Aitkenetal。 (2014)、サトウキビのHG2(相同グループ2)はSb05とSb06に整列し、HG8(相同グループ8)はSb02とSb08に整列し、基本的な染色体減少イベントの証拠を提供します(Aitken et al。、2014)。この研究では、BAC-14がSb02に整列し、BAC-19がSb08に整列したという証拠によって示されるように、ソルガム染色体Sb02およびSb08がS. spontaneumで染色体間再配列を示すことを観察しました(図7)。私たちの研究は、S。spontaneumにおけるソルガムの染色体間再配列の最初の物理的および細胞遺伝学的証拠を提供しました。私たちのグループからの未発表のデータは、Sb08が2つのセグメントに分割され、S。spontaneumでそれぞれSb02とSb09と統合されたことも明らかにしました(ZhangとMing、未発表のデータ)。Sb09、Sb08、Sb05、およびSb06に対応するプローブは、ソルガムとS.spontaneumの間の染色体間イベントを調査するためにさらに使用できます。 S. spontaneumには、さまざまな倍数性レベルがあります(2n = 40–128)(Irvine、1999)。これらのBACプローブは、倍数性レベルが異なるS.spontaneumの染色体間再配列を確認するために使用できます。
ソルガム(x = 10)とS. spontaneum(x = 8)、ソルガム染色体はS.spontaneumと1対1の対応ではありませんでした。前述の遺伝子マッピング研究のように、Sb08は2つのセグメントに分割され、S。spontaneumのSb02のセグメントとSb09のセグメントに統合されました。 Sb05は、Sb06のセグメントとSb07 S.spontaneumのセグメントと統合された2つのセグメントに分割されました。したがって、Sb08およびSb05に対応するプローブは単一のS. spontaneum染色体に特異的ではありませんでしたが、他の8つのソルガム染色体に対応する他のプローブはS.spontaneumの染色体特異的細胞遺伝学的BAC-FISHプローブとして使用できます。したがって、テストされたBACプローブは、8つのソルガム染色体(表2)に基づいており、BAC-FISHを使用した染色体の同定には十分でした。衛星染色体は、1つのS. spontaneum染色体(染色体3)で発見されました(Ha et al。、1999)。以前、同時FISHにより、45S rDNAのシグナルが、葯培養由来のS. spontaneumクローン(AP85-361)の染色体3内の衛星染色体の二次狭窄に位置することが明らかになりました(Ha et al。、1999)。この研究では、Sb03および45s rDNAに対応するBAC-2が、S。spontaneum染色体の同じ位置にマッピングされたため、以前の発見がさらに裏付けられました(Ha et al。、1999)。私たちの結果はまた、前の研究で命名されたS.spontaneumの染色体3がSb03と相同であるという直接的な証拠を提供しました。
結論
この研究では、Sの染色体特異的BACを開発しました.spontaneumは、BAC-FISH細胞遺伝学的マーカーを使用して染色体を同定するための簡単で再現性のある方法の開発に向けたステップとして、サトウキビの物理的マップを構築するためにソルガムゲノムに基づいて染色体特異的BACを分離する可能性を確認します。また、ソルガムとS.spontaneumの間の染色体間再配列の最初の細胞遺伝学的証拠を提供します。サトウキビBAC-FISH技術システムの確立は、核型分析、遺伝子局在化、物理的地図構築など、サトウキビ分子細胞遺伝学研究の新しい機会と手段を提供します。これらの結果は、全ゲノムシーケンスに必要なS.spontaneumゲノムの組み立てに不可欠です。
著者の貢献
GD、JS、およびJZが研究を考案し、実験を設計しました。 GD、JS、QZ、JW、QY、RM、KW、およびJZが実験を実行し、データを分析しました。 GDとJZが原稿を書きました。すべての著者が最終論文を読み、承認しました。
資金
このプロジェクトは、863プログラム(2013AA102604)、NSFC(31201260)、新世紀優秀人材プログラムからの助成金によって支援されました。福建省大学、および福建農林大学からの資金提供。
利益相反に関する声明
著者は、研究が商業的または財政的関係がない状態で行われたことを宣言します。これは潜在的な利益相反と解釈される可能性があります。
謝辞
BACシーケンスを組み立ててくれたXintanZhang博士と、英語を編集してくれたIreneLavagi博士に感謝します。
補足資料
Dhont、A。(2005)。 FISHとGISHを使用して倍数体のゲノム構造を解明する。サトウキビとバナナの例。 Cytogenet。ゲノム解像度109、27–33。 doi:10.1159 / 000082378
PubMed要約| CrossRef全文| Google Scholar
Piperidis、G.、D’Hont、A。、およびHogarth、D。M.(2001)。 「ゲノムinsitu(ハイブリダイゼーション)(GISH)によるさまざまなSaccharum種間雑種の染色体組成分析」、国際サトウキビ技術者協会の議事録、XXIV会議(ブリスベン、クイーンズランド州:ASSCT)、565–566。
Google Scholar
