Introdução
A cana-de-açúcar (Saccharum spp., Poaceae) é uma cultura anual ou perene cultivada em regiões tropicais e subtropicais em todo o mundo. A cana-de-açúcar é uma cultura importante para a produção de sacarose, responsável por 70% da produção mundial de açúcar. Além disso, por ser uma planta C4, a cana-de-açúcar pode converter energia solar em energia química de forma eficiente, sendo, portanto, uma cultura de biocombustível ideal para a produção de etanol (Lam et al., 2009; Santchurn et al., 2014). O gênero Saccharum possui seis espécies: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense e S. edule, entre as quais S. spontaneum é considerada a espécie selvagem com um conjunto cromossômico básico de x = 8. S. spontaneum também é uma das duas principais espécies de Saccharum que contribuem para o desenvolvimento de cultivares de cana-de-açúcar modernas (Dhont et al., 1998; Ha et al., 1999). S. spontaneum tem uma forte adaptabilidade ambiental e contém importantes características genéticas para resistência a doenças e à seca, contribuindo assim para a tolerância ao estresse de híbridos de cultivares modernos (Grivet et al., 2004). S. spontaneum tem a distribuição geográfica mais ampla, e seus níveis de ploidia variam de 2n = 5x = 40 a 2n = 16x = 128 (Panje e Babu, 1960).
Os cromossomos da cana-de-açúcar têm morfologias semelhantes e têm uma pequena tamanho de 1–6 μm no estágio de metáfase condensada (Ha et al., 1999; Dhont, 2005); portanto, é muito desafiador discernir entre os diferentes cromossomos com base nos métodos citogenéticos tradicionais. Além disso, por ser autopoliploide, são muito comuns suas alterações estruturais cromossômicas causadas por poliploidização, duplicação, deleção e recombinação (Piperidis et al., 2010). Em S. spontaneum, mapas de ligação genética foram desenvolvidos (Silva et al., 1995), mas o desenvolvimento de um mapa genético citológico ficou atrás de outras espécies de gramíneas. Ferramentas citológicas confiáveis para identificar cromossomos individuais podem ser usadas para pesquisa de genoma eficaz e utilização de recursos de germoplasma, especificamente com o histórico de genoma complexo de cultivares de cana-de-açúcar modernas. Os clones do cromossomo bacteriano artificial específico do cromossomo (BAC) são um recurso inestimável para pesquisas do genoma da cana-de-açúcar e terão muitas aplicações no mapeamento físico, identificação de cromossomos e criação assistida por marcador de Saccharum spp. E no auxílio a projetos de sequenciamento e montagem do genoma da cana-de-açúcar .
A hibridização fluorescente in situ (FISH) é uma ferramenta poderosa para a citologia molecular (Jiang e Gill, 2006). Em Saccharum spp., 45S rDNA e 5S rDNA foram usados como sondas para detectar os cromossomos básicos de S. spontaneum, S. robustum e S. officinarum, para identificar os diferentes cromossomos básicos, x = 8 em S. spontaneum, e x = 10 em S. robustum e S. officinarum (Dhont et al., 1998; Ha et al., 1999). Com base no FISH, os cultivares modernos de cana-de-açúcar foram encontrados para conter 70-80% dos cromossomos derivados de S. officinarum e 10-23% dos cromossomos de S. spontaneum, enquanto 5-17% parece ser o produto da recombinação entre S. spontaneum e S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). Além disso, eventos de eliminação, recombinação e translocação de cromossomos foram detectados em alguns progenitores derivados da hibridização entre Saccharum e Erianthus arundinaceus usando FISH (Dhont et al., 1995; Huang et al., 2015).
Sorgo tem um pequeno genoma diplóide (730 Mbp) com baixa frequência de eventos de recombinação de cromossomos. O sorgo compartilha alta sintenia com o genoma da cana-de-açúcar, tornando-se uma planta de referência ideal para análises comparativas com o genoma da cana-de-açúcar (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ) Neste estudo, com base nas sequências disponíveis para recursos BAC de alta cobertura, sondas BAC confiáveis foram desenvolvidas para identificação de cromossomos FISH e construção de mapa citogenético. Os objetivos deste estudo foram: (1) desenvolver um conjunto de sondas BAC-FISH específicas para cromossomos no cromossomo S. spontaneum e (2) explorar o rearranjo cromossômico entre S. spontaneum e sorgo.
Materiais e métodos
Materiais
Saccharum spontaneum SES208 (2n = 8x = 64) foi usado para análises citológicas neste estudo. As plantas SES208 foram cultivadas no campo no campus da Fujian Agricultural and Forestry University (Fuzhou, China) em fevereiro de 2015 e mantidas em condições normais de cultivo de cana-de-açúcar.
Uma biblioteca BAC foi construída a partir do genoma haplóide de S. spontaneum AP85-441 (4x = 32), que foi derivado de S. spontaneum SES208 (2n = 8x = 64) por meio de cultura in vitro de anteras. O tamanho do genoma de AP85-441 é de cerca de 3,2 Gbp. A biblioteca consistia em 38.400 clones, com um tamanho médio de inserção de 100 kb, cobrindo 6 × de todo o genoma.Esta biblioteca BAC foi usada para identificar os transportadores de sacarose e famílias de genes de frutocinase em S. spontaneum (Zhang et al., 2016; Chen et al., 2017).
Triagem da biblioteca BAC
35.156 clones BAC das bibliotecas AP85-441 foram agrupados em 701 bibliotecas. Cada biblioteca contém uma média de 50 clones BAC. As bibliotecas de DNA foram preparadas com o PhasePrepTM BAC DNA Kit (Sigma, Estados Unidos) seguindo os protocolos do fabricante. As bibliotecas de DNA BAC foram sequenciadas usando a plataforma Illumina HiSeq 2500 com o modelo PE250. Um total de 686 bibliotecas (18 bibliotecas falharam no sequenciamento) foram sequenciadas e 267,5 Gb de dados limpos foram gerados após o corte usando o Trimmomatic versão 0,36 (Bankevich et al., 2012). Os dados de cada pool BAC foram montados usando SPAdes versão 3.09 com parâmetro padrão. Um total de 2.611.145 contigs foram montados com contig N50 de 7,38 kbp (Zhang e Ming, dados não publicados).
As sequências da biblioteca AP85-441 BAC foram pesquisadas no BLAST contra o genoma do sorgo com um valor E de 1e -4. As sequências que tiveram acertos BLAST únicos no genoma do sorgo foram selecionadas como sondas FISH candidatas. Para identificar clones BAC de baixo número de cópias da biblioteca BAC, iniciadores específicos de sequência foram projetados usando o software Primer 5.0 para permitir que os pools de BAC sejam rastreados por PCR especificamente. Comprimentos de primer de 18-25 bp, valores de Tm de 55-65 ° C e composições de nucleotídeos de 40-60% de citosina e guanina foram selecionados. Um método de PCR de duas etapas foi usado para rastrear os pools de dimensão 3D da biblioteca BAC (Asakawa et al., 1997; Crooijmans et al., 2000) com algumas modificações. Cada clone de uma placa de 384 poços foi cultivado em 80 μl de caldo Lysogeny + 34 mg / ml de cloranfenicol durante a noite a 37 ° C em uma placa de cultura de 384 poços. No total, oito placas de 384 poços com um total de 3.072 clones BAC foram cultivadas. Para cada placa de 384 poços, primeiro construímos 16 pools de linha (linha A a P) e 24 pools de coluna (coluna 1-24) para cada placa de 384 poços, misturando volumes iguais de cultura de cada clone individual e, em seguida, misturamos os 16 conjuntos de linhas e 24 conjuntos de colunas junto com volumes iguais de cada conjunto de linhas e colunas como um conjunto de placas ou superconjunto. Neste estudo, focamos na triagem de oito placas de 384 poços. Assim, foram preparadas oito superpools para as oito placas separadamente. A placa de 384 poços positiva das oito placas foi identificada e, subsequentemente, a linha positiva entre as 16 misturas foi rastreada. Finalmente, as colunas positivas para PCR foram selecionadas entre 24 misturas de colunas. Vinte e quatro reações de PCR foram realizadas para identificar clones positivos de oito placas de 384 poços.
As amplificações de PCR foram realizadas como descrito anteriormente (Bouzidi et al., 2006). Cada reação incluiu rTaq pré-mistura 7,5 μl, 0,6 μl de cada iniciador (10 μM), 1,0 μl de molde líquido de bactérias, para um volume final de 20 μl. Após desnaturação inicial a 95 ° C por 5 min, foram realizados 35 ciclos de 95 ° C por 30 s, 55 ° C por 30 s e 72 ° C por 1 min. Os produtos de PCR foram analisados em um gel de agarose a 1,5%.
Preparação do cromossomo da metáfase
A preparação do cromossomo foi realizada conforme descrito anteriormente (Lou et al., 2010) com pequenas modificações. Em resumo, as plantas de S. spontaneum foram cultivadas em casa de vegetação para a colheita das pontas das raízes. As pontas das raízes excisadas com cerca de 1–2 cm foram tratadas com 0,002 mol / l 8-hidroxiquinolina em temperatura ambiente por 2–4 h, enxaguadas em água por 15 min e fixadas em etanol: ácido acético (3: 1) por pelo menos 24 h à temperatura ambiente. As pontas das raízes foram então digeridas em uma solução de enzima (4% de celulose R-10 e 2% de pectoliase em tampão de citrato 0,1 M) a 37 ° C por 1 h, lavadas com água desionizada com gelo por 30 min e finalmente incubadas em etanol: ácido acético (3: 1) durante cerca de 30 min. As lâminas foram preparadas de acordo com o método “seco à chama” (Iovene et al., 2008).
Purificação do DNA BAC e rotulagem da sonda
O DNA BAC foi extraído com PhasePrepTM BAC DNA Kit de acordo com o manual do fabricante. O DNA BAC purificado foi marcado pela reação de tradução de nick padrão, incluindo DNase I diluída, tampão de tradução de 10 × nick, DNA polimerase I, dNTPs, dUTPs marcados com biotina / digoxigenina e DNA BAC. foi incubado a 15 ° C por 1,5 h. Os produtos cortados foram então examinados em um gel de agarose a 1,5% quanto à presença de um esfregaço entre 300 e 500 bp. As sondas obtidas foram armazenadas a -20 ° C até serem utilizadas.
Hibridização e detecção in situ
Mistura de sondas e hibridização: Primeiro, a mistura de sondas (50% de formamida desionizada, 2 × SSC, 80 ng de DNA marcado com digoxigenina / biotina, 10% de sulfato de dextrana , > 1 μg C ° t-100) foi colocado em um bloco quente de 90 ° C por 5 min, em seguida, imediatamente transferido em gelo até que esteja pronto para sondar a hibridização sobre. As lâminas secas com chama previamente preparadas foram tratadas com formamida desionizada 70%, desnaturada em um bloco de aquecimento a 80 ° C por 1.5 min, imediatamente imerso sequencialmente em etanol 70% gelado, etanol 90% e, em seguida, etanol 100% cada por 5 min, e depois seco ao ar na bancada. Finalmente, sondas desnaturadas foram adicionadas a cada lâmina e cobertas com lamínulas de 24 × 32 mm. As lâminas foram colocadas em uma câmara úmida a 37 ° C durante a noite.
Detecção da sonda: as lamínulas foram removidas e as lâminas foram lavadas em temperatura ambiente em 2 × SSC por 5 min, a 42 ° C em 2 × SSC por 10 min e à temperatura ambiente em 1 × PBS por 5 min nesta sequência. Os sinais da sonda marcada com biotina foram detectados com 2 mg / ml de estreptavidina Alexa Fluor 488 e os sinais da sonda marcada com digoxigenina foram detectados com 2% de antidigoxigenina-rodamina de ovelhas. O coquetel de anticorpos (100 μl de tampão TNB, 1 μl de Alexa Fluor 488 estreptavidina, 1 μl de rodamina anti-dig-sheep) foi adicionado às lâminas, que foram cobertas com lamínulas de 24 × 32 mm, incubadas por 1 h a 37 ° C uma câmara úmida e lavada em temperatura ambiente em 1 × PBS três vezes por 5 min cada. O excesso de líquido foi removido e 4 ‘, 6-diamidino-2-fenilindol (DAPI) (Sigma, St. Louis, MO, Estados Unidos) na solução anti-desbotamento Vectashield (Vector, Burlingame, CA, Estados Unidos) foi adicionado para contrastar o cromossomos. As imagens foram capturadas com um microscópio de epifluorescência Olympus BX63. As imagens do sinal FISH foram analisadas usando o software CellSens Dimension.
Resultados
Triagem de clones BAC de baixa cópia
Para triar os clones BAC que potencialmente têm baixo número de cópias sequências no genoma de S. spontaneum, nós BLAST pesquisamos as bibliotecas AP85-441 BAC sequenciadas contra o genoma do sorgo. Um total de 2.000 sequências BAC correspondentes a 10 cromossomos de sorgo com boa colinearidade e mostrando especificidade de sequência foram rastreadas. As sequências BAC de 2000 AP85-441 foram mascaradas pelo REPEATMASKER contra o banco de dados de DNA de sorgo de alta repetição e o banco de dados de gramíneas TIGR para filtrar sequências de repetição. Finalmente, 114 BACs distribuídos em 10 cromossomos de sorgo foram selecionados para análise FISH (Tabela Suplementar 1) com 7–16 BACs em cada um dos 10 cromossomos de sorgo (Figura 1). Para rastrear clones BAC positivos, 114 iniciadores específicos foram projetados com base nas sequências do clone BAC (Tabela Suplementar 2) e 49 clones BAC positivos foram rastreados a partir das bibliotecas BAC (Tabela Suplementar 3).
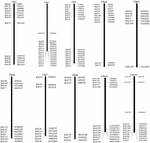
FIGURA 1. As distribuições de 114 clones BAC de AP85-441 nos cromossomos de sorgo. BAC1-114 representa o número BAC, os dados à direita são a localização física do BAC na região correspondente dos cromossomos do sorgo. Unit = bp.
Força e distribuição do sinal BAC-FISH
Amostras de DNA isoladas das 49 amostras positivas Os clones BAC foram marcados com biotina ou digoxigenina. As sondas 5S rDNA e 45S rDNA foram utilizadas como controle e foram hibridizadas com cromossomos metafásicos somáticos de S. spontaneum seguindo o procedimento FISH. Devido à correlação entre a força do sinal e a variação do conteúdo da sequência repetida das sequências BAC, usamos C ° t-100 como competidor (Tabela 1). Clones BAC exibindo sinais de hibridização fortes e estáveis foram selecionados para análise FISH adicional.

TABELA 1. A classificação de 27 clones positivos com bloqueio de DNA.
Um total de 64 cromossomos distintos pode ser observado na metáfase do S. spontaneum. Cada BAC-FISH foi realizado em quatro experimentos independentes (ou lâminas). Pelo menos 10 propagações de cromossomos metafásicos somáticos foram analisados em cada lâmina. Os resultados mostraram que as 27 sondas específicas (Tabela Complementar 4) podem ser classificadas em cinco grupos com base no sinal de hibridização (Tabela 1). No Grupo I (incluindo BAC-18, BAC-20, BAC-29 e BAC-84), as sondas exibiram oito locais distintos sem supressão competitiva in situ (CISS) usando C ° t-100 (Figura 2A), sugerindo poucas repetições sequências existiam nestas sequências BAC. No Grupo II (incluindo BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 e BAC-74), C ° t -100 foi usado como competidor para a hibridização da sonda BAC, e seis a sete locais bastante distintos foram exibidos em cariótipos deste grupo (Figura 2B). O resultado da hibridização foi semelhante ao rDNA 45S com sete sítios de sinal, que pode ser causado pela variação da estrutura cromossômica no cromossomo homólogo. No Grupo III (incluindo BAC-26, BAC-66, BAC-76 e BAC-78), DNA C ° t-100 foi usado como competidor e oito sítios distintos puderam ser observados no cariótipo, sugerindo este grupo de BAC os clones têm sequências de DNA repetitivas (Figura 2C). No Grupo IV (incluindo BAC-24), mais de oito locais de sinal foram observados no cariótipo e apareceram nas regiões pericentromérica e telomérica (Figura 2D), sugerindo que essas sondas BAC tinham sequências repetitivas em S. spontaneum.No grupo V (incluindo BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 e BAC-81), as sondas mostraram sinais dispersos em todos os cromossomos com regiões limitadas de cromossomos individuais exibindo sinais específicos (Figura 2E). O resultado da hibridização indicou que este grupo de clones BAC continha grandes quantidades de DNA repetitivo em S. spontaneum, o que pode ser causado por uma divergência evolutiva rápida para as regiões repetitivas após a divisão do sorgo e S. spontaneum. Portanto, das 49 sondas BAC, um conjunto de 19 sondas BAC (Tabela Suplementar 3) dos Grupos I, II e III mostraram sinais específicos nos cromossomos de S. spontaneum correspondendo a nove dos cromossomos de sorgo ao lado de Sb05. Os sinais das outras 22 sondas foram dispersos em todos os cromossomos. Selecionamos um conjunto de 19 sondas BAC como sinais específicos do cromossomo S. spontaneum (Figura 3). Das 19 sondas BAC, uma delas estava localizada em Sb02, Sb04 e Sb09; duas sondas em Sb07, Sb08 e Sb10; três sondas em Sb01 e Sb06; e quatro sondas em Sb03. Este conjunto de sondas forneceu uma ferramenta útil para a análise comparativa de S. spontaneum e sorgo (Tabela 2).
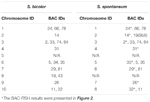
TABELA 2. A distribuição das sondas BAC-FISH nos cromossomos de S. bicolor e S. spontaneum.
Construção de uma biblioteca BAC específica para cromossomos de S. spontaneum
Sb01 e Sb02 são os dois maiores cromossomos de sorgo. Para detectar a sintenia do genoma entre sorgo e S. spontaneum, 10 clones BAC específicos para cromossomos foram selecionados dos Grupos I, II e III. Destas 10 sondas BAC, três sondas (clone BAC ID: 24, 66, 78) eram homólogas a Sb01 e sete sondas (clone BAC ID: 14, 18, 20, 69, 71, 73, 77) correspondiam a Sb02. Utilizamos a detecção de duas cores de FISH para pares BAC marcados de forma complementar e as sondas para mapear a relação citogenética da cadeia para os clones BAC. Fazendo isso, construímos o mapa citogenético de S. spontaneum com base nas 10 sondas BAC.
Para três sondas correspondentes a Sb01, a análise de FISH de S. spontaneum (SES208) mostrou que BAC-24 e BAC -78 sondas foram localizadas principalmente na região distal do cromossomo 1 de S. spontaneum, e BAC-66 foi mapeado nas proximidades da região centromérica (Figura 4). BAC-24 e BAC-66 localizados no mesmo cromossomo (cromossomo 1 de S. spontaneum); simultaneamente, enquanto a sonda 66 e a sonda 78 também estão localizadas no mesmo cromossomo, demonstrando que as três sondas BAC distribuídas para os mesmos cromossomos em S. spontaneum, sugerindo assim sintenia entre S. spontaneum e sorgo para o cromossomo 1. Os resultados FISH revelaram que as sete sondas BAC alinhado com Sb02; apenas as sondas 69, 71, 73 e 77 dos sete BACs geraram sinais intensos em um cromossomo S. spontaneum (SsChr2) (Figura 5); enquanto as sondas 14, 18 e 20 eram indetectáveis no cromossomo 2 de S. spontaneum, mas foram observadas em outros cromossomos de S. spontaneum. Estes resultados demonstraram que os rearranjos cromossômicos ocorreram no Sb02 correspondente entre S. spontaneum e sorgo.
Além disso, a sequência de BAC-2 compartilhou alta similaridade (97%) com um fragmento no cromossomo 3 de sorgo. A análise de FISH de cor dupla mostrou que ambos BAC-2 e rDNA 45s foram mapeados para a mesma localização do cromossomo S. spontaneum (Figura 6).
Reorganização de cromossomos em Sb02 entre Sorgo e S. spontaneum
Para investigar o rearranjo cromossômico em Sb02 entre sorgo e S. spontaneum, cromossomo sondas específicas correspondentes a Sb02 (BAC-14, BAC-18 e BAC-20) e correspondentes a Sb08 (BAC-19 e BAC-43) foram utilizadas para a análise de FISH. Os resultados mostraram que BAC-14 e BAC-19 foram mapeados para braços diferentes dos mesmos cromossomos de S. spontaneum, indicando que rearranjos cromossômicos ocorreram entre os cromossomos 2 e 8 em S. spontaneum e sorgo (Figura 7).
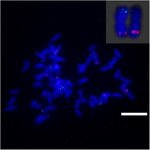
FIGURA 7. O rearranjo do cromossomo na cana-de-açúcar e sorgo revelado por FISH. Sondas BAC-14 e BAC-19 marcadas com biotina (verde) e digoxigenina (vermelho). Barras de escala = 10 μm.
Discussão
Saccharum é um gênero complexo caracterizado por altos níveis de poliploidia, com pequenos cromossomos (Ha et al., 1999; Dhont, 2005). Distinguir cromossomos Saccharum individuais com base em sua morfologia é muito desafiador. Aproveitando as sequências que são altamente cobertas pela biblioteca BAC de S. spontaneum, fomos capazes de rastrear o potencial de clones BAC de baixo número de cópias em S. spontaneum usando o genoma do sorgo como referência. Por ser uma espécie altamente poliplóide, S. spontaneum pode conter mais rearranjos de genoma do que diplóides.As diferenças dos genomas entre S. spontaneum e sorgo podem ser a causa da inexatidão nas previsões de sequências BAC de baixo número de cópias no genoma de S. spontaneum. Neste estudo, inicialmente obtivemos 114 sequências BAC potenciais com cópia única com base no genoma do sorgo. No entanto, apenas 49 puderam ser identificados nas bibliotecas BAC parciais e 27 foram consideradas sondas BAC-FISH específicas para cromossomos. Existem pelo menos duas razões para explicar esses resultados. Em primeiro lugar, as 114 sequências BAC eram fragmentos parciais da inserção BAC devido às limitações da montagem da sequência NGS e podem conter sequências repetidas nas outras regiões que não têm informações de sequência. Essas sequências de repetição potencialmente descobertas podem enganar a detecção de sequência de baixa cópia por meio da comparação de sequência. Em segundo lugar, a recente poliploidização em S. spontaneum pode causar a variação de sequências repetidas entre S. spontaneum e sorgo e, assim, resultar no sinal de FISH inespecífico em S. spontaneum. Em um estudo anterior, o sequenciamento de 20 BACs em híbridos de cana-de-açúcar gerou sequências contig de 1,45 Mb, as sequências alinhadas com o genoma do sorgo mediram 0,99 Mb, o que representou cerca de metade das sequências BAC da cana-de-açúcar (Wang et al., 2010). Obviamente, as deleções / inserções existiam entre o sorgo e o Saccharum, e a utilização do genoma do sorgo como referência não pode substituir as características únicas do genoma do Saccharum e, portanto, pode causar resultados de FISH imprevisíveis.
Alguns slides de FISH foram fortes ruído de fundo, que pode ser devido a pequenas regiões de sequências repetidas (como sequências SSR) no clone BAC, que consequentemente produzem interferência durante a hibridização. O genoma do sorgo tem um conteúdo de repetição de aproximadamente 61% (Paterson et al., 2009), enquanto cerca de metade do genoma é composto por sequências repetidas em híbridos Saccarrum (De et al., 2014). Nesses genomas ricos em repetições, é difícil desenvolver sondas FISH específicas para sinais, que distinguem cromossomos citogeneticamente com morfologias semelhantes. CISS com bloqueio de DNA C ° t-100 pode impedir eficientemente a repetição de sequências. Neste estudo, o bloqueio de DNA C ° t-100 foi usado para a análise de FISH das sondas BAC nos Grupos II e III (Figuras 2D-F), que foram verificadas como sondas BAC específicas do cromossomo, enquanto o FISH das sondas BAC nos Grupos IV e V produziu forte interferência de fundo (Figuras 2D-F). É óbvio que existem variações de sequências de repetição entre as sondas BAC examinadas. Portanto, a otimização dos experimentos seria necessária para a análise BAC-FISH em Saccharum. Seis a sete locais foram exibidos no Grupo II, o que pode ser causado por variações na estrutura do cromossomo homólogo, como a deleção do fragmento em um ou dois cromossomos homólogos. No trigo hexaplóide, o BAC 676D4 hibridizou mais fortemente com os cromossomos do genoma A do que com os cromossomos dos genomas B e D (Zhang et al., 2004). Estes indetectáveis de um a dois sinais nos cromossomos homólogos também podem ser causados por problemas técnicos de BAC-FISH para tantos cromossomos de tamanho pequeno.
Neste estudo, identificamos 27 BAC- específicos de cromossomos Sondas FISH que correspondem a 9 dos 10 cromossomos do sorgo (todos exceto Sb05). Em um estudo de mapa genético derivado de um cruzamento entre S. officinarum e cultivar de cana-de-açúcar, Sb05 foi fundido com Sb06 na cana-de-açúcar (Aitken et al., 2014). Em nosso estudo, um mapa genético baseado na população F1 de S. spontaneum revelou que Sb05 foi dividido em dois segmentos, e os dois segmentos foram fundidos com Sb06 e Sb07, respectivamente (Zhang e Ming, dados não publicados). Recentemente, o mapa genético de um membro da família Andropogoneae, Miscanthus sinensis, demonstrou que Sb05 tem baixa colinearidade com o grupo de ligação correspondente em M. sinensis (Ma et al., 2012). Da mesma forma, os dois cromossomos ancestrais de milho ortólogos para Sb05 retêm o menor número de ortólogos sintênicos para genes de sorgo (Schubert e Lysak, 2011). Portanto, a ausência do BAC-FISH correspondente a Sb05 pode indicar fusão cromossômica em S. spontaneum.
Sorghum e S. spontaneum divergiram 12 milhões de anos atrás (MYA), o número básico de cromossomos foi reduzido de x = 10 a x = 8. Em Aitken et al. (2014), o HG2 (grupo homólogo 2) de cana-de-açúcar alinhado a Sb05 e Sb06, e HG8 (grupo homólogo 8) a Sb02 e Sb08, fornecendo evidências para o evento de redução cromossômica básica (Aitken et al., 2014). Neste estudo, observamos que os cromossomos de sorgo Sb02 e Sb08 apresentavam rearranjo intercromossômico em S. spontaneum, conforme demonstrado pela evidência de que BAC-14 alinhado com Sb02 e BAC-19 alinhado com Sb08 (Figura 7). Nosso estudo forneceu a primeira evidência física e citogenética para o rearranjo intercromossômico do sorgo em S. spontaneum. Dados não publicados de nosso grupo também revelaram que Sb08 é dividido em dois segmentos e foram mesclados com Sb02 e Sb09 em S. spontaneum, respectivamente (Zhang e Ming, dados não publicados).As sondas correspondentes a Sb09, Sb08, Sb05 e Sb06 poderiam ser posteriormente utilizadas para investigar os eventos intercomossômicos entre sorgo e S. spontaneum. S. spontaneum tem uma ampla gama de níveis de ploidia (2n = 40–128) (Irvine, 1999). Essas sondas BAC podem ser usadas para confirmar o rearranjo intercromossômico de S. spontaneum com os diferentes níveis de poliploidia.
Devido ao número de cromossomos básicos diferentes entre sorgo (x = 10) e S. spontaneum (x = 8), os cromossomos do sorgo não eram uma correspondência de um para um com S. spontaneum. Conforme nosso estudo de mapeamento genético mencionado aqui antes, Sb08 foi dividido em dois segmentos que se fundiram com segmentos de Sb02 e segmentos de Sb09 em S. spontaneum; Sb05 foi dividido em dois segmentos que se fundiram com segmentos de Sb06 e segmentos de Sb07 S. spontaneum. Portanto, as sondas correspondentes a Sb08 e Sb05 não eram específicas para cromossomos únicos de S. spontaneum, enquanto as outras sondas correspondentes aos outros oito cromossomos de sorgo podem ser usadas como sondas citogenéticas BAC-FISH cromossômicas específicas para S. spontaneum. Assim, as sondas BAC testadas foram baseadas nos oito cromossomos de sorgo (Tabela 2) foram suficientes para a identificação de cromossomos usando BAC-FISH. Um cromossomo satélite foi encontrado em um cromossomo S. spontaneum (cromossomo 3) (Ha et al., 1999). Anteriormente, FISH simultâneo revelou que os sinais de 45S rDNA estavam localizados nas constrições secundárias dos cromossomos satélite dentro do cromossomo 3 do clone de S. spontaneum derivado de cultura de anteras (AP85-361) (Ha et al., 1999). Neste estudo, o BAC-2 que corresponde a Sb03 e 45s rDNA foram mapeados para a mesma localização do cromossomo S. spontaneum, apoiando, assim, descobertas anteriores (Ha et al., 1999). Nossos resultados também forneceram evidências diretas de que o cromossomo 3 de S. spontaneum nomeado no estudo anterior é homólogo ao Sb03.
Conclusão
Neste estudo, desenvolvemos BACs específicos para cromossomos de S . spontaneum como um passo em direção ao desenvolvimento de um método simples e reprodutível para identificação de cromossomos usando marcadores citogenéticos BAC-FISH, confirmando a viabilidade de isolar BACs cromossômicos específicos com base no genoma do sorgo para construir um mapa físico da cana-de-açúcar. Nós também fornecemos a primeira evidência citogenética de rearranjo intercromossômico entre sorgo e S. spontaneum. O estabelecimento do sistema de tecnologia BAC-FISH da cana-de-açúcar oferece novas oportunidades e os meios para a pesquisa citogenética molecular da cana-de-açúcar, incluindo análise de cariótipo, localização de genes e construção de mapas físicos. Esses resultados são essenciais para a montagem do genoma de S. spontaneum necessário para o sequenciamento do genoma inteiro.
Contribuições dos autores
GD, JS e JZ conceberam o estudo e projetaram os experimentos. GD, JS, QZ, JW, QY, RM, KW e JZ realizaram os experimentos e analisaram os dados. GD e JZ escreveram o manuscrito. Todos os autores leram e aprovaram o artigo final.
Financiamento
Este projeto foi apoiado por doações do programa 863 (2013AA102604), NSFC (31201260), Programa para talentos excelentes do novo século em Fujian Province University, e o financiamento da Fujian Agriculture and Forestry University.
Declaração de Conflito de Interesses
Os autores declaram que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pode ser interpretado como um potencial conflito de interesses.
Agradecimentos
Agradecemos ao Dr. Xintan Zhang por montar as sequências BAC e à Dra. Irene Lavagi pela edição do inglês.
Material Suplementar
Dhont, A. (2005). Desvendando a estrutura do genoma de poliplóides usando FISH e GISH; exemplos de cana-de-açúcar e banana. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
Resumo PubMed | CrossRef Full Text | Google Scholar
Piperidis, G., D’Hont, A. e Hogarth, D. M. (2001). “Análise da composição cromossômica de vários híbridos interespecíficos de Saccharum por genômica in situ (hibridização) (GISH)”, em Proceedings of the International Society of Sugar Cane Technologists, XXIV Congresso (Brisbane, QLD: ASSCT), 565-566.
Google Scholar
