Introduzione
La canna da zucchero (Saccharum spp., Poaceae) è una coltura annuale o perenne coltivata nelle regioni tropicali e subtropicali di tutto il mondo. La canna da zucchero è una coltura importante per la produzione di saccarosio e rappresenta il 70% della produzione mondiale di zucchero. Inoltre, come pianta C4, la canna da zucchero può convertire in modo efficiente l’energia solare in energia chimica, ed è quindi una coltura ideale per biocarburanti per la produzione di etanolo (Lam et al., 2009; Santchurn et al., 2014). Il genere Saccharum comprende sei specie: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense e S. edule, tra le quali S. spontaneum è considerata la specie selvatica con un insieme cromosomico di base x = 8. S. spontaneum è anche una delle due principali specie di Saccharum che contribuiscono allo sviluppo delle moderne cultivar di canna da zucchero (Dhont et al., 1998; Ha et al., 1999). S. spontaneum ha una forte adattabilità ambientale e contiene importanti tratti genetici per la resistenza alle malattie e alla siccità, contribuendo così alla tolleranza allo stress dei moderni ibridi di cultivar (Grivet et al., 2004). S. spontaneum ha la più ampia distribuzione geografica e i suoi livelli di ploidia vanno da 2n = 5x = 40 a 2n = 16x = 128 (Panje e Babu, 1960).
I cromosomi della canna da zucchero hanno morfologie simili e hanno un piccolo dimensione di 1–6 μm allo stadio di metafase condensata (Ha et al., 1999; Dhont, 2005); quindi, è molto difficile discernere tra i diversi cromosomi sulla base di metodi citogenetici tradizionali. Inoltre, come autopoliploide, le sue alterazioni strutturali cromosomiche causate da poliploidizzazione, duplicazione, delezione e ricombinazione sono molto comuni (Piperidis et al., 2010). In S. spontaneum, sono state sviluppate mappe di collegamento genetico (Silva et al., 1995), ma lo sviluppo di una mappa genetica citologica è rimasta indietro rispetto ad altre specie di erba. Strumenti citologici affidabili per identificare i singoli cromosomi possono essere utilizzati per un’efficace ricerca sul genoma e l’utilizzo delle risorse del germoplasma, in particolare con il complesso background genomico delle moderne cultivar di canna da zucchero. I cloni di cromosomi artificiali batterici specifici per cromosomi (BAC) sono una risorsa inestimabile per le ricerche sul genoma della canna da zucchero e avranno molte applicazioni nella mappatura fisica, nell’identificazione dei cromosomi e nell’allevamento assistito da marcatori delle Saccharum spp. .
L’ibridazione in situ a fluorescenza (FISH) è un potente strumento per la citologia molecolare (Jiang e Gill, 2006). In Saccharum spp., 45S rDNA e 5S rDNA sono stati utilizzati come sonde per rilevare i cromosomi di base di S. spontaneum, S. robustum e S. officinarum, per identificare i diversi cromosomi di base, x = 8 in S. spontaneum e x = 10 in S. robustum e S. officinarum (Dhont et al., 1998; Ha et al., 1999). Sulla base della FISH, è stato scoperto che le cultivar moderne della canna da zucchero contengono il 70-80% dei cromosomi derivati da S. officinarum e il 10-23% dei cromosomi da S. spontaneum, mentre il 5-17% sembra essere il prodotto della ricombinazione tra S. spontaneum e S. officinarum (Dhont et al., 1996; Piperidis et al., 2001; Cuadrado et al., 2004). Inoltre, sono stati rilevati eventi di eliminazione, ricombinazione e traslocazione cromosomica in alcuni progenitori derivati dall’ibridazione tra Saccharum ed Erianthus arundinaceus utilizzando FISH (Dhont et al., 1995; Huang et al., 2015).
Sorgo ha un piccolo genoma diploide (730 Mbp) con una bassa frequenza di eventi di ricombinazione cromosomica. Il sorgo condivide un’elevata sintonia con il genoma della canna da zucchero, rendendolo una pianta di riferimento ideale per analisi comparative con il genoma della canna da zucchero (Ming et al., 1998; Nazeema et al., 2007; Wang et al., 2010; Aitken et al., 2014 ). In questo studio, sulla base delle sequenze disponibili per le risorse BAC ad alta copertura, sono state sviluppate sonde BAC affidabili per l’identificazione del cromosoma FISH e la costruzione di mappe citogenetiche. Gli obiettivi di questo studio erano: (1) sviluppare una serie di sonde BAC-FISH specifiche per cromosomi sul cromosoma S. spontaneum e (2) esplorare il riarrangiamento cromosomico tra S. spontaneum e sorgo.
Materiali e metodi
Materiali
Saccharum spontaneum SES208 (2n = 8x = 64) è stato utilizzato per le analisi citologiche in questo studio. Le piante SES208 sono state coltivate sul campo nel campus della Fujian Agricultural and Forestry University (Fuzhou, Cina) nel febbraio 2015 e mantenute in condizioni di crescita regolare della canna da zucchero.
È stata costruita una libreria BAC dal genoma aploide di S. spontaneum AP85-441 (4x = 32), che è stato derivato da S. spontaneum SES208 (2n = 8x = 64) tramite coltura in vitro di antera. La dimensione del genoma dell’AP85-441 è di circa 3,2 Gbp. La libreria consisteva di 38.400 cloni, con una dimensione media dell’inserto di 100 kb, che coprivano 6 × dell’intero genoma.Questa libreria BAC è stata utilizzata per identificare i trasportatori di saccarosio e le famiglie di geni della fruttochinasi in S. spontaneum (Zhang et al., 2016; Chen et al., 2017).
Screening the BAC Library
35.156 cloni BAC dalle librerie AP85-441 sono stati raggruppati in 701 librerie. Ogni libreria contiene una media di 50 cloni BAC. Le librerie di DNA sono state preparate con il kit PhasePrepTM BAC DNA (Sigma, Stati Uniti) seguendo i protocolli del produttore. Le librerie di DNA BAC sono state sequenziate utilizzando la piattaforma HiSeq 2500 Illumina con il modello PE250. Un totale di 686 librerie (18 librerie non riuscite per il sequenziamento) sono state sequenziate e 267,5 Gb di dati puliti sono stati generati dopo il taglio utilizzando Trimmomatic versione 0.36 (Bankevich et al., 2012). I dati di ogni pool BAC sono stati assemblati utilizzando SPAdes versione 3.09 con parametro predefinito. Un totale di 2.611.145 contig sono stati assemblati con contig N50 di 7,38 kbp (Zhang e Ming, dati non pubblicati).
Le sequenze della libreria BAC AP85-441 sono state ricercate BLAST contro il genoma del sorgo con un valore E di 1e -4. Le sequenze che hanno avuto singoli colpi BLAST nel genoma del sorgo sono state selezionate come sonde FISH candidate. Per identificare i cloni BAC con un numero basso di copie dalla libreria BAC, sono stati progettati primer specifici per sequenza utilizzando il software Primer 5.0 per consentire lo screening specifico dei pool BAC mediante PCR. Sono state selezionate lunghezze di primer di 18–25 bp, valori di Tm di 55–65 ° C e composizioni nucleotidiche di citosina e guanina al 40–60%. Un metodo PCR in due fasi è stato utilizzato per schermare i pool di dimensioni 3D della libreria BAC (Asakawa et al., 1997; Crooijmans et al., 2000) con alcune modifiche. Ciascun clone da una piastra da 384 pozzetti è stato coltivato in 80 μl di brodo Lysogeny + 34 mg / ml di cloramfenicolo per una notte a 37 ° C in una piastra di coltura da 384 pozzetti. In totale, sono state coltivate otto piastre da 384 pozzetti con un totale di 3072 cloni BAC. Per ogni piastra da 384 pozzetti, abbiamo prima costruito 16 pool di righe (riga da A a P) e 24 pool di colonne (colonna 1–24) per ciascuna piastra da 384 pozzetti mescolando volumi uguali di coltura di ogni singolo clone, quindi miscelando il 16 pool di righe e 24 pool di colonne insieme con volumi uguali da ogni pool di righe e colonne come un pool di piastre o superpool. In questo studio, ci siamo concentrati sullo screening di otto piastre da 384 pozzetti. Pertanto, otto superpiscine sono state preparate separatamente per le otto piastre. La piastra positiva a 384 pozzetti dalle otto piastre è stata identificata e successivamente è stata selezionata la riga positiva tra le 16 miscele. Infine, le colonne positive alla PCR sono state selezionate tra 24 miscele di colonne. Sono state eseguite 24 reazioni PCR per identificare cloni positivi dalle otto piastre da 384 pozzetti.
Le amplificazioni PCR sono state eseguite come descritto in precedenza (Bouzidi et al., 2006). Ogni reazione includeva rTaq premix 7,5 μl, 0,6 μl di ciascun primer (10 μM), 1,0 μl di stampo liquido per batteri, fino a un volume finale di 20 μl. Dopo la denaturazione iniziale a 95 ° C per 5 minuti, sono stati eseguiti 35 cicli a 95 ° C per 30 secondi, 55 ° C per 30 secondi e 72 ° C per 1 minuto. I prodotti della PCR sono stati analizzati su un gel di agarosio all’1,5%.
Preparazione dei cromosomi in metafase
La preparazione dei cromosomi è stata eseguita come descritto in precedenza (Lou et al., 2010) con piccole modifiche. In breve, le piante di S. spontaneum sono state coltivate in condizioni di serra per la raccolta delle punte delle radici. Le punte delle radici asportate di circa 1-2 cm sono state trattate con 0,002 mol / l di 8-idrossichinolina a temperatura ambiente per 2-4 ore, sciacquate in acqua per 15 minuti e fissate in etanolo: acido acetico (3: 1) per almeno 24 ore h a temperatura ambiente. Le punte delle radici sono state quindi digerite in una soluzione enzimatica (4% di cellulosa R-10 e 2% di pectolasi in tampone citrato 0,1 M) a 37 ° C per 1 h, lavate con acqua deionizzata con ghiaccio per 30 min e infine incubate in etanolo: acido acetico (3: 1) per circa 30 min. I vetrini sono stati preparati utilizzando il metodo “essiccato alla fiamma” (Iovene et al., 2008).
Purificazione del DNA BAC ed etichettatura della sonda
Il DNA BAC è stato estratto con PhasePrepTM BAC Kit DNA secondo il manuale del produttore. Il DNA BAC purificato è stato etichettato mediante reazione di traduzione nick standard, tra cui DNasi I diluito, tampone di traduzione nick 10 ×, DNA polimerasi I, dNTP, dUTP marcati con biotina / digossigenina e DNA BAC. La miscela è stato incubato a 15 ° C per 1,5 h. I prodotti tagliati sono stati quindi esaminati su un gel di agarosio all’1,5% per la presenza di uno striscio tra 300 e 500 bp. Le sonde ottenute sono state conservate a -20 ° C fino al momento dell’uso.
Ibridazione e rilevamento in situ
Miscela di sonde e ibridazione: in primo luogo, la miscela di sonde (50% di formammide deionizzata, 2 × SSC, 80 ng di DNA marcato con digossigenina / biotina, 10% di destrano solfato , > 1 μg C ° t-100) è stato posto in un blocco caldo a 90 ° C per 5 min, quindi immediatamente trasferito su ghiaccio fino al momento di sondare per ibridizzazione su. I vetrini essiccati alla fiamma precedentemente preparati sono stati trattati con formammide deionizzata al 70%, denaturata su blocco termico a 80 ° C per 1.5 min, immerso immediatamente in sequenza in etanolo al 70% ghiacciato, etanolo al 90% e quindi etanolo al 100% ciascuno per 5 min, quindi essiccato all’aria sul banco. Infine, sono state aggiunte sonde denaturate a ciascun vetrino e coperte con coprioggetti 24 × 32 mm. I vetrini sono stati posti in una camera umida a 37 ° C durante la notte.
Rilevamento della sonda: i vetrini coprioggetto sono stati rimossi e i vetrini sono stati lavati a temperatura ambiente in 2 × SSC per 5 min, a 42 ° C in 2 × SSC per 10 minuti ea temperatura ambiente in 1 × PBS per 5 minuti in questa sequenza. I segnali della sonda marcati con biotina sono stati rilevati con 2 mg / ml di streptavidina Alexa Fluor 488 ei segnali della sonda marcati con digossigenina sono stati rilevati con il 2% di anti-digossigenina-rodamina di pecora. Il cocktail di anticorpi (100 μl di tampone TNB, 1 μl di streptavidina Alexa Fluor 488, 1 μl di rodamina anti-dig-sheep) è stato aggiunto ai vetrini, che sono stati coperti con coprioggetti 24 × 32 mm, incubati per 1 ora a 37 ° C in una camera umida e lavata a temperatura ambiente in 1 × PBS tre volte per 5 minuti ciascuna. Il liquido in eccesso è stato rimosso ed è stato aggiunto 4 ‘, 6-diamidino-2-fenilindolo (DAPI) (Sigma, St. Louis, MO, Stati Uniti) nella soluzione antifade Vectashield (Vector, Burlingame, CA, Stati Uniti) per controcolorare il cromosomi. Le immagini sono state acquisite con un microscopio a epifluorescenza Olympus BX63. Le immagini del segnale FISH sono state analizzate utilizzando il software CellSens Dimension.
Risultati
Screening dei cloni BAC a bassa copia
Per eseguire lo screening dei cloni BAC che potenzialmente hanno un numero di copie basso sequenze nel genoma di S. spontaneum, abbiamo ricercato BLAST le librerie BAC AP85-441 sequenziate contro il genoma del sorgo. Sono state esaminate un totale di 2000 sequenze BAC corrispondenti a 10 cromosomi di sorgo con una buona collinearità e che mostravano la specificità della sequenza. Le 2000 sequenze BAC AP85-441 sono state mascherate da REPEATMASKER rispetto al database del DNA del sorgo ad alta ripetizione e al database delle graminacee TIGR per filtrare le sequenze ripetute. Infine, 114 BAC distribuiti su 10 cromosomi del sorgo sono stati selezionati per l’analisi FISH (Tabella 1 supplementare) con 7-16 BAC su ciascuno dei 10 cromosomi del sorgo (Figura 1). Per lo screening dei cloni BAC positivi, sono stati progettati 114 primer specifici sulla base delle sequenze dei cloni BAC (tabella supplementare 2) e 49 cloni BAC positivi sono stati selezionati dalle librerie BAC (tabella supplementare 3).
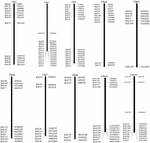
FIGURA 1. Le distribuzioni di 114 cloni BAC da AP85-441 nei cromosomi del sorgo. BAC1-114 rappresenta il numero BAC, i dati a destra sono la posizione fisica di BAC nella corrispondente regione dei cromosomi del sorgo. Unità = bp.
Forza e distribuzione del segnale BAC-FISH
campioni di DNA isolati dai 49 positivi I cloni BAC sono stati etichettati con biotina o digossigenina. Le sonde 5S rDNA e 45S rDNA sono state utilizzate come controllo e sono state ibridate ai cromosomi metafase somatici di S. spontaneum seguendo la procedura FISH. A causa della correlazione tra la potenza del segnale e la variazione del contenuto di sequenze ripetute delle sequenze BAC, abbiamo usato C ° t-100 come concorrente (Tabella 1). I cloni BAC che mostravano segnali di ibridazione forti e stabili sono stati selezionati per ulteriori analisi FISH.

TABELLA 1. La classificazione di 27 cloni positivi con DNA bloccante.
È possibile osservare un totale di 64 cromosomi distinti nella metafase del S. spontaneum. Ciascun BAC-FISH è stato eseguito in quattro esperimenti (o vetrini) indipendenti. In ogni vetrino sono stati analizzati almeno 10 spread di cromosomi somatici in metafase. I risultati hanno mostrato che le 27 sonde specifiche (Tabella supplementare 4) possono essere classificate in cinque gruppi in base al segnale di ibridazione (Tabella 1). Nel gruppo I (inclusi BAC-18, BAC-20, BAC-29 e BAC-84), le sonde hanno visualizzato otto siti distintivi senza soppressione in situ competitiva (CISS) utilizzando C ° t-100 (Figura 2A), suggerendo che pochi ripetuti sequenze esistevano in queste sequenze BAC. Nel gruppo II (inclusi BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 e BAC-74), C ° t -100 è stato utilizzato come concorrente per l’ibridazione della sonda BAC e sono stati visualizzati da sei a sette siti abbastanza distinti nei cariotipi di questo gruppo (Figura 2B). Il risultato dell’ibridazione è stato simile a 45S rDNA con sette siti di segnale, che possono essere causati dalla variazione della struttura cromosomica nel cromosoma omologo. Nel gruppo III (inclusi BAC-26, BAC-66, BAC-76 e BAC-78), il DNA C ° t-100 è stato utilizzato come concorrente e otto siti distinti potrebbero essere osservati nel cariotipo, suggerendo questo gruppo di BAC i cloni hanno sequenze di DNA ripetitive (Figura 2C). Nel gruppo IV (incluso BAC-24), più di otto siti di segnale sono stati osservati nel cariotipo e sono comparsi nelle regioni pericentromeriche e telomeriche (Figura 2D), suggerendo che queste sonde BAC avevano sequenze ripetitive in S. spontaneum.Nel gruppo V (inclusi BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 e BAC-81), le sonde hanno mostrato segnali dispersi in tutti i cromosomi con regioni limitate dei singoli cromosomi che mostrano segnali specifici (Figura 2E). Il risultato dell’ibridazione ha indicato che questo gruppo di cloni BAC conteneva grandi quantità di DNA ripetitivo in S. spontaneum, che può essere causato da una rapida divergenza evolutiva per le regioni ripetitive dopo la scissione di sorgo e S. spontaneum. Pertanto, delle 49 sonde BAC, un set di 19 sonde BAC (tabella supplementare 3) dei gruppi I, II e III ha mostrato segnali specifici sui cromosomi di S. spontaneum corrispondenti a nove dei cromosomi del sorgo accanto a Sb05. I segnali delle altre 22 sonde erano dispersi in tutti i cromosomi. Abbiamo selezionato un set di 19 sonde BAC come segnali specifici del cromosoma di S. spontaneum (Figura 3). Delle 19 sonde BAC, una si trovava su Sb02, Sb04 e Sb09; due sonde su Sb07, Sb08 e Sb10; tre sonde su Sb01 e Sb06; e quattro sonde su Sb03. Questo set di sonde ha fornito uno strumento utile per l’analisi comparativa di S. spontaneum e sorgo (Tabella 2).
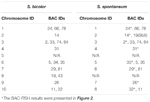
TABELLA 2. La distribuzione delle sonde BAC-FISH nei cromosomi di S. bicolor e S. spontaneum.
Costruzione di una libreria BAC specifica per cromosomi di S. spontaneum
Sb01 e Sb02 sono i due più grandi cromosomi del sorgo. Per rilevare la sintenia genomica tra sorgo e S. spontaneum, sono stati selezionati 10 cloni BAC specifici per cromosoma dai gruppi I, II e III. Di queste 10 sonde BAC, tre (ID clone BAC: 24, 66, 78) erano omologhe a Sb01 e sette sonde (ID clone BAC: 14, 18, 20, 69, 71, 73, 77) corrispondevano a Sb02. Abbiamo utilizzato il rilevamento a doppio colore di FISH per coppie BAC etichettate in modo complementare e le sonde per mappare la relazione citogenetica della catena per i cloni BAC. In questo modo, abbiamo costruito la mappa citogenetica di S. spontaneum sulla base delle 10 sonde BAC.
Per tre sonde corrispondenti a Sb01, l’analisi FISH di S. spontaneum (SES208) ha mostrato che BAC-24 e BAC -78 sonde erano principalmente localizzate sulla regione distale del cromosoma 1 di S. spontaneum e BAC-66 è stato mappato in stretta prossimità della regione centromerica (Figura 4). BAC-24 e BAC-66 localizzati sullo stesso cromosoma (cromosoma 1 di S. spontaneum); simultaneamente mentre la sonda 66 e la sonda 78 si trovano anche sullo stesso cromosoma, dimostrando che le tre sonde BAC si sono distribuite agli stessi cromosomi in S. spontaneum suggerendo così una sintenia tra S. spontaneum e sorgo per il cromosoma 1. I risultati FISH hanno rivelato che le sette sonde BAC allineato a Sb02; solo le sonde 69, 71, 73 e 77 dei sette BAC hanno generato segnali intensi su un cromosoma di S. spontaneum (SsChr2) (Figura 5); mentre le sonde 14, 18 e 20 non erano rilevabili sul cromosoma 2 di S. spontaneum ma sono state osservate su altri cromosomi di S. spontaneum. Questi risultati hanno dimostrato i riarrangiamenti cromosomici avvenuti nel corrispondente Sb02 tra S. spontaneum e sorgo.
Inoltre, la sequenza di BAC-2 condivideva un’elevata somiglianza (97%) con un frammento sul cromosoma 3 di sorgo. L’analisi FISH a doppio colore ha mostrato che sia l’rDNA di BAC-2 che quello di 45s erano mappati nella stessa posizione del cromosoma di S. spontaneum (Figura 6).
Riarrangiamento cromosomico su Sb02 tra sorgo e S. spontaneum
Per approfondire il riarrangiamento cromosomico su Sb02 tra sorgo e S. spontaneum, cromosoma -sonde specifiche corrispondenti a Sb02 (BAC-14, BAC-18 e BAC-20) e corrispondenti a Sb08 (BAC-19 e BAC-43) sono state utilizzate per l’analisi FISH. I risultati hanno mostrato che BAC-14 e BAC-19 sono stati mappati su bracci diversi degli stessi cromosomi di S. spontaneum, indicando che si sono verificati riarrangiamenti cromosomici tra i cromosomi 2 e 8 in S. spontaneum e sorgo (Figura 7).
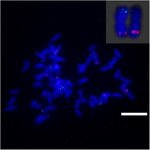
FIGURA 7. Il riarrangiamento del cromosoma nella canna da zucchero e nel sorgo come rivelato da FISH. Sonde BAC-14 e BAC-19 etichettate con biotina (verde) e digossigenina (rossa). Barre della scala = 10 μm.
Discussione
Il saccharum è un genere complesso caratterizzato da alti livelli di poliploidia, con piccoli cromosomi (Ha et al., 1999; Dhont, 2005). Distinguere i singoli cromosomi di Saccharum in base alla loro morfologia è molto impegnativo. Sfruttando le sequenze che sono ampiamente coperte dalla libreria BAC di S. spontaneum, siamo stati in grado di esaminare i potenziali cloni BAC a basso numero di copie in S. spontaneum utilizzando il genoma del sorgo come riferimento. Essendo una specie ad alto poliploide, S. spontaneum può contenere più riarrangiamenti del genoma rispetto ai diploidi.Le differenze dei genomi tra S. spontaneum e sorgo possono essere causa di inesattezza nelle previsioni delle sequenze BAC a basso numero di copie nel genoma di S. spontaneum. In questo studio, abbiamo inizialmente ottenuto 114 potenziali sequenze BAC con copia singola basata sul genoma del sorgo. Tuttavia, solo 49 potrebbero essere identificati nelle librerie BAC parziali e 27 si sono rivelate sonde BAC-FISH specifiche per i cromosomi. Ci sono almeno due ragioni per spiegare questi risultati. In primo luogo, le 114 sequenze BAC erano frammenti parziali dell’inserimento di BAC a causa delle limitazioni dell’assemblaggio di sequenze NGS e possono contenere sequenze ripetute nelle altre regioni che non hanno informazioni sulla sequenza. Queste sequenze ripetute potenzialmente scoperte potrebbero fuorviare il rilevamento di sequenze a bassa copia attraverso il confronto delle sequenze. In secondo luogo, la recente poliploidizzazione in S. spontaneum può causare la variazione di sequenze ripetute tra S. spontaneum e sorgo, e quindi provocare il segnale FISH non specifico in S. spontaneum. In uno studio precedente, il sequenziamento di 20 BAC in ibridi di canna da zucchero ha generato sequenze contig di 1,45 Mb, le sequenze che si allineavano con il genoma del sorgo coprivano 0,99 Mb, che rappresentavano circa la metà delle sequenze di BAC della canna da zucchero (Wang et al., 2010). Ovviamente, esistevano delezioni / inserzioni tra il sorgo e il Saccharum e l’utilizzo del genoma del sorgo come riferimento non può sostituire le caratteristiche uniche del genoma del Saccharum e quindi può causare risultati FISH imprevisti.
Alcuni vetrini FISH avevano forti rumore di fondo, che può essere dovuto a piccole regioni di sequenze ripetute (come sequenze SSR) nel clone BAC, che di conseguenza producono interferenze durante l’ibridazione. Il genoma del sorgo ha un contenuto ripetuto di circa il 61% (Paterson et al., 2009), mentre circa la metà del genoma è composta da sequenze ripetute negli ibridi di Saccarrum (De et al., 2014). In questi genomi ricchi di ripetizioni, è difficile sviluppare sonde FISH segnale-specifiche, che distinguono i cromosomi citogeneticamente con morfologie simili. Il CISS con DNA bloccante C ° t-100 può precludere efficacemente le sequenze ripetute. In questo studio, il blocco del DNA C ° t-100 è stato utilizzato per l’analisi FISH delle sonde BAC nei gruppi II e III (figure 2D-F), che sono state verificate come sonde BAC specifiche per il cromosoma, mentre il FISH delle sonde BAC nei gruppi IV e V ha prodotto una forte interferenza di fondo (Figure 2D – F). È ovvio che ci sono variazioni di sequenze ripetute tra le sonde BAC esaminate. Pertanto, l’ottimizzazione degli esperimenti sarebbe necessaria per l’analisi BAC-FISH in Saccharum. Sono stati visualizzati da sei a sette siti nel gruppo II, che potrebbero essere causati dalle variazioni della struttura cromosomica omologa, come la delezione dei frammenti in uno o due cromosomi omologhi. Nel grano esaploide, BAC 676D4 si è ibridato più fortemente con i cromosomi del genoma A che con i cromosomi del genoma B e D (Zhang et al., 2004). Questi segnali non rilevabili di uno o due nei cromosomi omologhi potrebbero anche essere causati dai problemi tecnici di BAC-FISH per tanti cromosomi di piccole dimensioni.
In questo studio, abbiamo identificato 27 BAC specifici per i cromosomi Sonde FISH che corrispondono a 9 dei 10 cromosomi del sorgo (tutti tranne Sb05). In uno studio della mappa genetica derivata da un incrocio tra S. officinarum e cultivar di canna da zucchero, Sb05 è stato fuso con Sb06 nella canna da zucchero (Aitken et al., 2014). Nel nostro studio, una mappa genetica basata sulla popolazione F1 di S. spontaneum ha rivelato che Sb05 era diviso in due segmenti, e che i due segmenti erano fusi rispettivamente con Sb06 e Sb07 (Zhang e Ming, dati non pubblicati). Recentemente, la mappa genetica di un membro dell’Andropogoneae, Miscanthus sinensis, ha dimostrato che Sb05 ha una scarsa collinearità con il corrispondente gruppo di collegamento in M. sinensis (Ma et al., 2012). Allo stesso modo, i due cromosomi ancestrali del mais ortologhi a Sb05 conservano il minor numero di ortologhi sintenici ai geni del sorgo (Schubert e Lysak, 2011). Pertanto, l’assenza del BAC-FISH corrispondente a Sb05 può indicare la fusione dei cromosomi in S. spontaneum.
Sorghum e S. spontaneum divergevano 12 milioni di anni fa (MYA), il numero di cromosomi di base era ridotto da x = 10 ax = 8. In Aitken et al. (2014), l’HG2 (gruppo omologo 2) della canna da zucchero allineato a Sb05 e Sb06 e HG8 (gruppo omologato 8) a Sb02 e Sb08, fornendo prove per l’evento di riduzione cromosomica di base (Aitken et al., 2014). In questo studio, abbiamo osservato che i cromosomi del sorgo Sb02 e Sb08 avevano un riarrangiamento intercromosomico in S. spontaneum, come dimostrato dall’evidenza che BAC-14 si allineava a Sb02 e BAC-19 si allineava a Sb08 (Figura 7). Il nostro studio ha fornito la prima prova fisica e citogenetica per il riarrangiamento inter-cromosomico del sorgo in S. spontaneum. I dati non pubblicati del nostro gruppo hanno anche rivelato che Sb08 è diviso in due segmenti e sono stati fusi con Sb02 e Sb09 in S. spontaneum, rispettivamente (Zhang e Ming, dati non pubblicati).Le sonde corrispondenti a Sb09, Sb08, Sb05 e Sb06 potrebbero essere ulteriormente utilizzate per indagare gli eventi inter-cromosomici tra sorgo e S. spontaneum. S. spontaneum ha una vasta gamma di livelli di ploidia (2n = 40–128) (Irvine, 1999). Queste sonde BAC potrebbero essere utilizzate per confermare il riarrangiamento inter-cromosomico di S. spontaneum con i diversi livelli di poliploidia.
A causa del diverso numero di cromosomi di base tra sorgo (x = 10) e S. spontaneum (x = 8), i cromosomi del sorgo non erano una corrispondenza uno-a-uno con S. spontaneum. Come il nostro studio di mappatura genetica menzionato in precedenza, Sb08 è stato diviso in due segmenti che si sono fusi con segmenti di Sb02 e segmenti di Sb09 in S. spontaneum; Sb05 è stato diviso in due segmenti che si sono fusi con segmenti di Sb06 e segmenti di Sb07 S. spontaneum. Pertanto, le sonde corrispondenti a Sb08 e Sb05 non erano specifiche per singoli cromosomi di S. spontaneum, mentre le altre sonde corrispondenti agli altri otto cromosomi di sorgo possono essere utilizzate come sonde BAC-FISH citogenetiche cromosomiche specifiche per S. spontaneum. Pertanto, le sonde BAC testate erano basate sugli otto cromosomi del sorgo (Tabella 2) erano sufficienti per l’identificazione cromosomica utilizzando BAC-FISH. Un cromosoma satellite è stato trovato in un cromosoma di S. spontaneum (cromosoma 3) (Ha et al., 1999). In precedenza, la FISH simultanea ha rivelato che i segnali del 45S rDNA erano localizzati sulle costrizioni secondarie dei cromosomi satellite all’interno del cromosoma 3 dal clone di S. spontaneum derivato dalla coltura dell’antera (AP85-361) (Ha et al., 1999). In questo studio, BAC-2 che corrisponde a Sb03 e 45s rDNA sono stati mappati nella stessa posizione del cromosoma di S. spontaneum, supportando così ulteriormente i risultati precedenti (Ha et al., 1999). I nostri risultati hanno anche fornito prove dirette che il cromosoma 3 di S. spontaneum denominato nello studio precedente è omologa a Sb03.
Conclusione
In questo studio, abbiamo sviluppato BAC specifici per il cromosoma di S lo spontaneum come passo verso lo sviluppo di un metodo semplice e riproducibile per l’identificazione dei cromosomi utilizzando marcatori citogenetici BAC-FISH, che confermano la fattibilità dell’isolamento di BAC specifici per cromosoma basati sul genoma del sorgo per costruire una mappa fisica della canna da zucchero. Forniamo anche la prima evidenza citogenetica di riarrangiamento inter-cromosomico tra sorgo e S. spontaneum. La creazione del sistema tecnologico BAC-FISH della canna da zucchero offre nuove opportunità e mezzi per la ricerca sulla citogenetica molecolare della canna da zucchero, tra cui l’analisi del cariotipo, la localizzazione genica e la costruzione di mappe fisiche. Questi risultati sono essenziali per l’assemblaggio del genoma di S. spontaneum richiesto per il sequenziamento dell’intero genoma.
Contributi dell’autore
GD, JS e JZ hanno concepito lo studio e progettato gli esperimenti. GD, JS, QZ, JW, QY, RM, KW e JZ hanno condotto gli esperimenti e analizzato i dati. GD e JZ hanno scritto il manoscritto. Tutti gli autori hanno letto e approvato il documento finale.
Finanziamento
Questo progetto è stato sostenuto da sovvenzioni del programma 863 (2013AA102604), NSFC (31201260), Program for New Century Excellent Talents in Fujian Province University e il finanziamento della Fujian Agriculture and Forestry University.
Conflict of Interest Statement
Gli autori dichiarano che la ricerca è stata condotta in assenza di rapporti commerciali o finanziari che potrebbe essere interpretato come un potenziale conflitto di interessi.
Ringraziamenti
Ringraziamo il Dr. Xintan Zhang per aver assemblato le sequenze BAC e la Dr. Irene Lavagi per aver modificato l’inglese.
Materiale supplementare
Dhont, A. (2005). Svelare la struttura del genoma dei poliploidi utilizzando FISH e GISH; esempi di canna da zucchero e banana. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
Estratto di PubMed | Testo completo di CrossRef | Google Scholar
Piperidis, G., D’Hont, A. e Hogarth, D. M. (2001). “Analisi della composizione cromosomica di vari ibridi interspecifici Saccharum mediante genomica in situ (ibridazione) (GISH)”, in Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD: ASSCT), 565-566.
Google Scholar
