Johdanto
Sokeriruoko (Saccharum spp., Poaceae) on yksivuotinen tai monivuotinen sato, jota kasvatetaan trooppisilla ja subtrooppisilla alueilla maailmanlaajuisesti. Sokeriruoko on tärkeä sakkaroosintuotanto, ja sen osuus maailman sokerintuotannosta on 70%. Lisäksi sokeriruoko voi C4-laitoksena muuntaa aurinkoenergian tehokkaasti kemialliseksi energiaksi ja on siksi ihanteellinen biopolttoainesato etanolin tuotantoon (Lam et al., 2009; Santchurn et al., 2014). Saccharum-suvulla on kuusi lajia: S. spontaneum, S. robustum, S. officinarum, S. barberi, S. sinense ja S. edule, joista S. spontaneumia pidetään villinä lajina, jonka kromosomin perusjoukko on x = 8. S. spontaneum on myös yksi kahdesta Saccharum-lajista, jotka myötävaikuttavat nykyaikaisten sokeriruokolajikkeiden kehitykseen (Dhont et ai., 1998; Ha et ai., 1999). S. spontaneumilla on vahva ympäristön sopeutumiskyky ja se sisältää tärkeitä geneettisiä piirteitä sairauksien ja kuivuuden vastustuskyvylle, mikä osaltaan myötävaikuttaa nykyisten lajikehybridien stressinsietokykyyn (Grivet et al., 2004). S. spontaneumilla on laajin maantieteellinen jakauma, ja sen ploidiatasot vaihtelevat välillä 2n = 5x = 40 – 2n = 16x = 128 (Panje ja Babu, 1960).
Sokeriruoko-kromosomeilla on samanlaiset morfologiat ja niillä on pieni koko 1–6 μm tiivistetyssä metafaasivaiheessa (Ha et ai., 1999; Dhont, 2005); näin ollen on erittäin haastavaa erottaa eri kromosomit perinteisten sytogeneettisten menetelmien perusteella. Lisäksi autopolyploidina sen kromosomaaliset rakenteelliset muutokset, jotka johtuvat polyploidisaatiosta, päällekkäisyydestä, deleetiosta ja rekombinaatiosta, ovat hyvin yleisiä (Piperidis et ai., 2010). S. spontaneumissa on kehitetty geneettisiä kytkentäkarttoja (Silva et al., 1995), mutta sytologisen geneettisen kartan kehitys jäi jälkeen muista ruoholajeista. Luotettavia sytologisia työkaluja yksittäisten kromosomien tunnistamiseksi voidaan käyttää tehokkaaseen genomitutkimukseen ja itusplasmin resurssien hyödyntämiseen, erityisesti nykyaikaisten sokeriruokolajikkeiden monimutkaisen genomitaustan kanssa. Kromosomispesifiset bakteerien keinotekoiset kromosomikloonit (BAC) ovat korvaamaton resurssi sokeriruokogenomitutkimuksissa, ja niillä on monia sovelluksia Saccharum spp: n fyysisessä kartoituksessa, kromosomien tunnistamisessa ja markkereiden avulla tapahtuvassa kasvatuksessa sekä sokeriruo’on genomin sekvensointi- ja kokoonpanoprojekteissa. .
Fluoresenssi in situ -hybridisaatio (FISH) on tehokas työkalu molekyylisytologiaan (Jiang ja Gill, 2006). Saccharum spp: ssä 45S rDNA: ta ja 5S rDNA: ta käytettiin koettimina S. spontaneumin, S. robustumin ja S. officinarumin peruskromosomien havaitsemiseksi erilaisten peruskromosomien tunnistamiseksi, x = 8 S. spontaneumissa ja x = 10 in S. robustum ja S. officinarum (Dhont et ai., 1998; Ha et ai., 1999). FISH: n perusteella nykyaikaisten sokeriruokolajikkeiden havaittiin sisältävän 70–80% S. officinarum -bakteerista ja 10–23% S. spontaneumin kromosomeista, kun taas 5–17% näyttää olevan yhdistelmän yhdistelmä. S. spontaneum ja S. officinarum (Dhont et ai., 1996; Piperidis et ai., 2001; Cuadrado et ai., 2004). Lisäksi kromosomien eliminointi-, rekombinaatio- ja translokaatiotapahtumat havaittiin joissakin esisoluissa, jotka olivat peräisin Saccharumin ja Erianthus arundinaceuksen välisestä hybridisaatiosta FISH: n avulla (Dhont et al., 1995; Huang et ai., 2015).
Sorghum on pieni diploidinen genomi (730 Mbp) ja matalalla kromosomien rekombinaatiotapahtumilla. Durralla on korkea synteeni sokeriruoko genomin kanssa, joten se on ihanteellinen vertailukasvi vertaileville analyyseille sokeriruo’on genomin kanssa (Ming et ai., 1998; Nazeema et ai., 2007; Wang ym., 2010; Aitken ym., 2014 ). Tässä tutkimuksessa kehitettiin luotettavia BAC-koettimia, jotka perustuivat käytettävissä oleviin sekvensseihin korkean peittoasteen BAC-resursseille FISH-kromosomien tunnistamiseksi ja sytogeneettisen kartan rakentamiseksi. Tämän tutkimuksen tavoitteena oli: (1) kehittää joukko kromosomispesifisiä BAC-FISH-koettimia S. spontaneumin kromosomissa ja (2) tutkia S. spontaneumin ja durran kromosomien uudelleenjärjestelyä.
Materiaalit ja menetelmät
Materiaalit
Saccharum spontaneum SES208 (2n = 8x = 64) käytettiin sytologisiin analyyseihin tässä tutkimuksessa. SES208-kasveja kasvatettiin pellolla Fujianin maa- ja metsätalousyliopiston (Fuzhou, Kiina) kampuksella helmikuussa 2015, ja niitä ylläpidettiin säännöllisissä sokeriruokokasvuolosuhteissa.
Haploidigenomista rakennettiin BAC-kirjasto. S. spontaneum AP85-441 (4x = 32), joka oli johdettu S. spontaneum SES208: sta (2n = 8x = 64) anteroidulla in vitro -viljelmällä. AP85-441: n genomikoko on noin 3,2 Gbp. Kirjasto koostui 38400 kloonista, joiden keskimääräinen insertikoko oli 100 kb, joka peitti 6 × koko genomista.Tätä BAC-kirjastoa on käytetty tunnistamaan sakkaroosikuljettimet ja fruktokinaasigeeniperheet S. spontaneumissa (Zhang et al., 2016; Chen et ai., 2017).
BAC-kirjaston seulonta
35 156 BAC-kloonia AP85-441-kirjastoista yhdistettiin 701 kirjastoon. Jokainen kirjasto sisältää keskimäärin 50 BAC-kloonia. DNA-kirjastot valmistettiin PhasePrepTM BAC DNA -sarjalla (Sigma, Yhdysvallat) noudattaen valmistajan ohjeita. BAC-DNA-kirjastot sekvensoitiin käyttämällä Illumina HiSeq 2500 -alustaa PE250-mallilla. Yhteensä 686 kirjastoa (18 kirjastoa epäonnistui sekvensoinnissa) sekvensoitiin, ja 267,5 Gt puhdistettua dataa tuotettiin leikkaamisen jälkeen käyttämällä Trimmomatic-versiota 0,36 (Bankevich et ai., 2012). Kunkin BAC-poolin tiedot koottiin käyttäen SPAdes-versiota 3.09 oletusparametrilla. Yhteensä 2 611 145 contigia koottiin 7,38 kbp: n jatkumalla N50 (Zhang ja Ming, julkaisematon data).
AP85-441-BAC-kirjastosekvenssejä haettiin BLAST-durran genomia vastaan E-arvolla 1e -4. Sekvenssit, joilla oli yksittäisiä BLAST-osumia durra-genomissa, valittiin ehdokas FISH-koettimiksi. Alhaisen kopiomäärän BAC-kloonien tunnistamiseksi BAC-kirjastosta, sekvenssispesifiset alukkeet suunniteltiin käyttäen Primer 5.0 -ohjelmistoa, jotta BAC-poolit voidaan seuloa PCR: llä. Alustepituudet 18-25 emäsparia, Tm-arvot 55-65 ° C ja nukleotidikoostumukset, joissa oli 40-60% sytosiinia ja guaniinia, valittiin. Kaksivaiheista PCR-menetelmää käytettiin seulomaan BAC-kirjaston 3D-ulottuvuuspoolit (Asakawa et ai., 1997; Crooijmans et ai., 2000) joillakin muunnoksilla. Kutakin 384-kuoppalevyn kloonia viljeltiin 80 ul: ssa Lysogeny-liemi + 34 mg / ml kloramfenikolia yön yli 37 ° C: ssa 384-kuoppaisella viljelylevyllä. Yhteensä kahdeksan 384-kuoppalevyä, joissa oli yhteensä 3072 BAC-kloonia, viljeltiin. Rakensimme kullekin 384-kuoppaiselle levylle ensin 16 rivipoolia (rivit A – P) ja 24 pylväspoolia (sarakkeet 1 – 24) kullekin 384-kuoppaiselle levylle sekoittamalla yhtä suuret määrät viljelmiä jokaiselle yksittäiselle kloonille ja sekoittamalla 16 rivipoolia ja 24 pylväspoolaa yhdessä saman määrän kanssa jokaisesta rivistä ja pylväspoolista yhtenä levypoolina tai superpoolina. Tässä tutkimuksessa keskityimme kahdeksan 384-kuoppaisen levyn seulontaan. Siten kahdeksalle levylle valmistettiin kahdeksan superpoolia erikseen. Kahdeksan levyn positiivinen 384-kuoppalevy tunnistettiin ja sen jälkeen seulottiin 16 seoksen positiivinen rivi. Lopuksi PCR-positiiviset kolonnit valittiin 24 kolonniseoksen joukosta. Kaksikymmentäneljä PCR-reaktiota tehtiin positiivisten kloonien tunnistamiseksi kahdeksasta 384-kuoppaisesta levystä.
PCR-monistukset suoritettiin aiemmin kuvatulla tavalla (Bouzidi et ai., 2006). Kukin reaktio sisälsi rTaq-esisekoitetta 7,5 μl, 0,6 μl kutakin aluketta (10 μM), 1,0 μl bakteerien nestemallia lopulliseen tilavuuteen 20 μl. Alkuperäisen denaturoinnin jälkeen 95 ° C: ssa 5 minuutin ajan suoritettiin 35 sykliä, joissa 95 ° C oli 30 sekuntia, 55 ° C 30 sekunnin ajan ja 72 ° C 1 minuutin ajan. PCR-tuotteet analysoitiin 1,5-prosenttisella agaroosigeelillä.
Metafaasikromosomivalmistelu
Kromosomivalmistus suoritettiin aikaisemmin kuvatulla tavalla (Lou et ai., 2010) pienin muutoksin. Lyhyesti sanottuna S. spontaneum -kasveja kasvatettiin kasvihuoneolosuhteissa juurikärkien korjuuseen. Noin 1-2 cm leikatut juurikärjet käsiteltiin 0,002 mol / l 8-hydroksikinoliinilla huoneenlämpötilassa 2–4 tuntia, huuhdottiin vedessä 15 minuutin ajan ja kiinnitettiin etanoli: etikkahappoon (3: 1) vähintään 24 tunnin ajan. h huoneenlämmössä. Juuren kärjet hajotettiin sitten entsyymiliuoksessa (4% selluloosa R-10 ja 2% pektolyaasia 0,1 M sitraattipuskurissa) 37 ° C: ssa 1 tunti, pestiin jäädionisoimattomalla vedellä 30 minuutin ajan ja inkuboitiin lopuksi etanolissa: etikkahappo (3: 1) noin 30 minuutin ajan. Objektiivilevyt valmistettiin käyttäen ”liekkikuivattua” menetelmää (Iovene et ai., 2008).
BAC-DNA: n puhdistus ja koettimien merkitseminen
BAC-DNA uutettiin PhasePrepTM BAC: lla Puhdistettu BAC-DNA leimattiin tavallisella nick-translaatioreaktiolla, joka sisälsi laimennetun DNaasi I: n, 10 x nikkelin translaatiopuskurin, DNA-polymeraasi I: n, dNTP: t, biotiini / digoksigeniinileimatut dUTP: t ja BAC-DNA: n. inkuboitiin 15 ° C: ssa 1,5 h. Leikatut tuotteet tutkittiin sitten 1,5-prosenttisella agaroosigeelillä tahran läsnäolon välillä 300 – 500 emäsparia. Saatuja koettimia säilytettiin -20 ° C: ssa, kunnes niitä käytettiin. >
In situ -hybridisaatio ja -havainto
Koettimen seos ja hybridisaatio: Ensinnäkin koetinseos (50% deionisoitua formamidia, 2 × SSC, 80 ng digoksigeniini / biotiinileimattua DNA: ta, 10% dekstraanisulfaattia , > 1 μg C ° t-100) pantiin 90 ° C: n kuumalohkoon 5 minuutiksi, sitten siirrettiin välittömästi jäihin, kunnes se oli valmis koettelemaan hybridisaatiota päällä. Aikaisemmin valmistettuja liekkikuivattuja lasilevyjä käsiteltiin 70-prosenttisella deionisoidulla formamidilla, joka oli denaturoitu lämpölohkossa 80 ° C: ssa yhden tunnin ajan.5 minuuttia, upotetaan välittömästi peräkkäin jääkylmään 70-prosenttiseen etanoliin, 90-prosenttiseen etanoliin ja sitten 100-prosenttiseen etanoliin kumpikin 5 minuutin ajan ja sitten ilmakuivataan penkillä. Lopuksi denaturoidut koettimet lisättiin kuhunkin objektilasiin ja peitettiin 24 × 32 mm: n peitelevyillä. Objektilevyt asetettiin kosteaan kammioon 37 ° C: seen yön yli.
Koettimen havaitseminen: peitelevyt poistettiin ja objektilasit pestiin huoneenlämpötilassa 2 x SSC: ssä 5 minuutin ajan, 42 ° C: ssa 2 x SSC 10 minuutin ajan ja huoneenlämpötilassa 1 x PBS: ssä 5 minuutin ajan tässä sekvenssissä. Biotiinilla leimatut koettimen signaalit havaittiin 2 mg / ml Alexa Fluor 488 -streptavidiinilla ja digoksigeniinileimatut koettimen signaalit havaittiin 2-prosenttisella lampaiden anti-digoksigeniini-rodamiinilla. Vasta-ainekokteili (100 μl TNB-puskuria, 1 μl Alexa Fluor 488 streptavidiinia, 1 μl rodamiinin anti-dig-lampaita) lisättiin dioille, jotka peitettiin 24 × 32 mm: n peittolevyillä, inkuboitiin 1 h 37 ° C: ssa. kosteaan kammioon ja pestiin huoneenlämpötilassa 1 x PBS: ssä kolme kertaa 5 minuutin ajan. Ylimääräinen neste poistettiin ja 4 ’, 6-diamidino-2-fenyyli-indoli (DAPI) (Sigma, St. Louis, MO, Yhdysvallat) haalistumista estävään liuokseen (Vectashield (Vector, Burlingame, CA, Yhdysvallat)) lisättiin. kromosomit. Kuvat otettiin Olympus BX63 -epifluoresenssimikroskoopilla. FISH-signaalikuvat analysoitiin käyttäen CellSens Dimension -ohjelmistoa.
Tulokset
Vähäkopioitujen BAC-kloonien seulonta
BAC-kloonien seulonta, joilla on mahdollisesti pieni kopioluku sekvenssejä S. spontaneum -genomissa, BLAST etsinyt sekvensoituja AP85-441 BAC-kirjastoja durra-genomia vastaan. Seulottiin yhteensä 2000 BAC-sekvenssiä, jotka vastasivat 10 durra-kromosomia, joilla oli hyvä kollineaarisuus ja joilla oli sekvenssispesifisyys. REPEATMASKER peitti vuoden 2000 AP85-441-BAC-sekvenssit korkean toistuman durra-DNA-tietokantaa ja TIGR gramineae -tietokantaa vastaan toistosekvenssien suodattamiseksi. Lopuksi 114 BAC: ta, jotka oli jaettu 10 durra-kromosomiin, valittiin FISH-analyysiä varten (täydentävä taulukko 1) ja 7-16 BAC: ta kussakin 10 durra-kromosomissa (kuvio 1). Positiivisten BAC-kloonien seulomiseksi suunniteltiin 114 spesifistä aluketta BAC-kloonisekvenssien perusteella (täydentävä taulukko 2) ja 49 positiivista BAC-kloonia seulottiin BAC-kirjastoista (täydentävä taulukko 3).
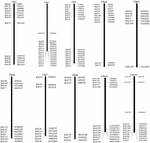
KUVA 1. 114: n AP85-441-BAC-kloonin jakaumat durra-kromosomeissa. BAC1-114 edustaa BAC-numeroa, oikealla olevat tiedot ovat BAC: n fyysinen sijainti durran kromosomien vastaavalla alueella. Yksikkö = bp.
BAC-FISH-signaalin voimakkuus ja jakauma
49 positiivisesta eristetyt DNA-näytteet BAC-kloonit leimattiin biotiinilla tai digoksigeniinilla. 5S-rDNA- ja 45S-rDNA-koettimia käytettiin kontrollina ja hybridisoitiin S. spontaneumin somaattisten metafaasikromosomien kanssa FISH-menettelyn mukaisesti. Signaalinvoimakkuuden ja BAC-sekvenssien toistuvan sekvenssipitoisuuden vaihtelun vuoksi käytimme kilpailijana C ° t-100: ta (taulukko 1). Vahvoja ja vakaita hybridisaatiosignaaleja osoittavat BAC-kloonit valittiin edelleen FISH-analyysiä varten.

TAULUKKO 1. 27 positiivisen kloonin luokittelu estävän DNA: n kanssa.
Yhteensä 64 erillistä kromosomia voidaan havaita S. spontaneumin metafaasissa. Jokainen BAC-FISH suoritettiin neljässä itsenäisessä kokeessa (tai diassa). Jokaisessa objektilasissa analysoitiin vähintään 10 somaattisten metafaasikromosomien leviämistä. Tulokset osoittivat, että 27 spesifistä koetinta (täydentävä taulukko 4) voidaan luokitella viiteen ryhmään hybridisaatiosignaalin perusteella (taulukko 1). Ryhmässä I (mukaan lukien BAC-18, BAC-20, BAC-29 ja BAC-84) koettimet osoittivat kahdeksan erottuvaa kohtaa ilman kilpailevaa in situ -vaimennusta (CISS) käyttäen ° C-t-100: ta (kuva 2A), mikä viittaa harvoihin toistuviin näissä BAC-sekvensseissä oli sekvenssejä. Ryhmässä II (mukaan lukien BAC-2, BAC-11, BAC-14, BAC-19, BAC-31, BAC-33, BAC-43, BAC-71, BAC-73 ja BAC-74), C ° t -100: ta käytettiin kilpailijana BAC-koettimen hybridisaatiossa, ja tämän ryhmän kariotyypeissä näytettiin kuusi – seitsemän varsin erillistä kohtaa (kuvio 2B). Hybridisaatiotulos oli samanlainen kuin 45S rDNA, jossa oli seitsemän signaalipaikkaa, mikä voi johtua kromosomirakenteen vaihteluista homologisessa kromosomissa. Ryhmässä III (mukaan lukien BAC-26, BAC-66, BAC-76 ja BAC-78) C ° t-100-DNA: ta käytettiin kilpailijana ja kariotyypissä voitiin havaita kahdeksan erillistä paikkaa, mikä viittaa tähän BAC-ryhmään klooneilla on toistuvia DNA-sekvenssejä (kuvio 2C). Ryhmässä IV (mukaan lukien BAC-24) havaittiin yli kahdeksan signaalipaikkaa kariotyypissä ja ne esiintyivät peritsentromeerisilla ja telomeerisillä alueilla (kuvio 2D), mikä viittaa siihen, että näillä BAC-koettimilla oli toistuvia sekvenssejä S. spontaneumissa.Ryhmässä V (mukaan lukien BAC-4, BAC-5, BAC-32, BAC-34, BAC-35, BAC-69, BAC-77 ja BAC-81) koettimet osoittivat hajaantuneita signaaleja kaikissa kromosomeissa, joilla oli rajoitettuja alueita yksittäisiä kromosomeja, jotka näyttävät spesifisiä signaaleja (kuva 2E). Hybridisaatiotulos osoitti, että tämä BAC-klooniryhmä sisälsi suuria määriä toistuvaa DNA: ta S. spontaneumissa, mikä voi johtua nopeasta evoluutioerosta toistuville alueille durran ja S. spontaneumin jakautumisen jälkeen. Siksi 49 BAC-koettimesta 19 BAC-koettimen joukko (täydentävä taulukko 3) ryhmistä I, II ja III osoitti spesifisiä signaaleja S. spontaneum -kromosomeissa, jotka vastaavat yhdeksää durra-kromosomia Sb05: n vieressä. Muiden 22 koettimen signaalit hajotettiin kaikkiin kromosomeihin. Valitsimme joukon 19 BAC-koetinta S. spontaneumin kromosomispesifisiksi signaaleiksi (kuva 3). 19 BAC-koettimesta yksi koetin sijaitsi alueilla Sb02, Sb04 ja Sb09; kaksi koetinta malleilla Sb07, Sb08 ja Sb10; kolme koetinta malleilla Sb01 ja Sb06; ja neljä koetinta Sb03: lla. Tämä koetinjoukko tarjosi hyödyllisen työkalun S. spontaneumin ja durra vertailevaan analyysiin (taulukko 2).
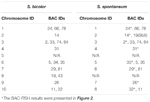
TAULUKKO 2. BAC-FISH-koettimien jakauma S. bicolorin ja S. spontaneumin kromosomeissa.
S. spontaneumin kromosomeille spesifisen BAC-kirjaston rakentaminen
Sb01 ja Sb02 ovat kaksi suurinta durra-kromosomia. Durran ja S. spontaneumin välisen genomisyntenian havaitsemiseksi valittiin 10 kromosomispesifistä BAC-kloonia ryhmistä I, II ja III. Näistä 10 BAC-koettimesta kolme koetinta (BAC-kloonin ID: 24, 66, 78) oli homologinen Sb01: n kanssa ja seitsemän koetinta (BAC-kloonin tunnus: 14, 18, 20, 69, 71, 73, 77) vastasi Sb02: ta. Käytimme FISH: n kaksiväristä havaitsemista komplementaarisesti leimattuihin BAC-pareihin ja koettimia BAC-kloonien ketjusytogeneettisen suhteen kartoittamiseen. Näin muodostimme S. spontaneumin sytogeneettisen kartan 10 BAC-koettimen perusteella.
Kolmen Sb01: tä vastaavan koettimen kohdalla S. spontaneumin (SES208) FISH-analyysi osoitti, että BAC-24 ja BAC -78 koetinta sijaitsi pääasiassa S. spontaneumin kromosomin 1 distaalisella alueella, ja BAC-66 kartoitettiin lähellä centromeerista aluetta (kuvio 4). BAC-24 ja BAC-66, jotka sijaitsevat samassa kromosomissa (S. spontaneum -kromosomi 1); samanaikaisesti, kun taas koettimet 66 ja koetin 78 sijaitsevat myös samassa kromosomissa, mikä osoittaa, että kolme BAC-koetinta jakautui samoihin kromosomeihin S. spontaneumissa, mikä viittaa kromosomin 1 S. spontaneumin ja durran väliseen synteeniaan. kohdistettu Sb02: een; vain koettimet 69, 71, 73 ja 77 seitsemästä BAC: sta tuottivat voimakkaita signaaleja yhdelle S. spontaneum -kromosomille (SsChr2) (kuvio 5); kun taas koettimia 14, 18 ja 20 ei voitu havaita S. spontaneum -kromosomissa 2, mutta havaittiin muissa S. spontaneumin kromosomeissa. Nämä tulokset osoittivat, että kromosomin uudelleenjärjestelyt tapahtuivat vastaavassa Sb02: ssa S. spontaneumin ja durra välillä.
Lisäksi BAC-2: n sekvenssillä oli suuri samankaltaisuus (97%) fragmentin kanssa kromosomissa 3 durra. Kaksivärinen FISH-analyysi osoitti, että sekä BAC-2 että 45s rDNA kartoitettiin samaan sijaintiin S. spontaneum -kromosomissa (kuva 6).
Kromosomien uudelleenjärjestely Sb02: lla Sorghumin ja S. spontaneumin välillä
Sb02: n kromosomien uudelleenjärjestäytymisen selvittämiseksi durra ja S. spontaneum, kromosomi – erityisiä koettimia, jotka vastasivat Sb02: ta (BAC-14, BAC-18 ja BAC-20) ja vastaavat Sb08: ta (BAC-19 ja BAC-43), käytettiin FISH-analyysiin. Tulokset osoittivat, että BAC-14 ja BAC-19 kartoitettiin samojen S. spontaneum -kromosomien eri käsivarsiin, mikä osoittaa, että S. spontaneumin ja durra-kromosomien 2 ja 8 välillä tapahtui kromosomien uudelleenjärjestelyjä (kuva 7).
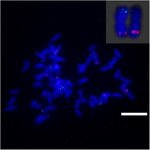
KUVA 7. Kromosomin uudelleenjärjestely sokeriruokossa ja durrassa, kuten FISH paljasti. BAC-14- ja BAC-19-koettimet, jotka on merkitty biotiinilla (vihreä) ja digoksigeniinilla (punainen). Asteikkopalkit = 10 μm.
Keskustelu
Saccharum on monimutkainen suku, jolle on ominaista korkea polyploidiataso, pienillä kromosomeilla (Ha et ai., 1999; Dhont, 2005). Yksittäisten Saccharum-kromosomien erottaminen niiden morfologian perusteella on erittäin haastavaa. Hyödyntämällä sekvenssejä, jotka ovat erittäin peitossa S. spontaneumin BAC-kirjastossa, pystyimme seulomaan S. spontaneumin mahdollisesti pienen kopiomäärän BAC-kloonit käyttämällä durra-genomia viitteenä. Korkean polyploidin lajina S. spontaneum voi sisältää enemmän genomin uudelleenjärjestelyjä kuin diploidit.S. spontaneumin ja durran väliset genomien erot voivat olla epätarkkuuden syy S. spontaneum -geenin pienen kopiomäärän BAC-sekvenssien ennusteissa. Tässä tutkimuksessa olemme alun perin saaneet 114 potentiaalista BAC-sekvenssiä, joissa on yksi kopio durra-genomiin perustuen. Kuitenkin vain 49 voitiin tunnistaa osittaisissa BAC-kirjastoissa, ja 27 havaittiin olevan kromosomispesifisiä BAC-FISH-koettimia. On ainakin kaksi syytä selittää nämä tulokset. Ensinnäkin 114 BAC-sekvenssiä olivat osittaisia fragmentteja BAC-insertiosta NGS-sekvenssikokoonpanon rajoitusten vuoksi, ja ne voivat sisältää toistosekvenssejä muilla alueilla, joilla ei ole sekvenssitietoa. Nämä mahdollisesti paljastamattomat toistosekvenssit voivat johtaa harhaanjohtavan sekvenssin havaitsemista sekvenssivertailun avulla. Toiseksi viimeaikainen polyploidisaatio S. spontaneumissa voi aiheuttaa toistuvien sekvenssien vaihtelun S. spontaneumin ja durra välillä ja johtaa siten epäspesifiseen FISH-signaaliin S. spontaneumissa. Aikaisemmassa tutkimuksessa 20 BAC: n sekvensointi sokeriruokohybrideissä tuotti 1,45 Mb: n jatkumissekvenssejä, durra-genomiin kohdistuvien sekvenssien pituus oli 0,99 Mb, mikä oli noin puolet sokeriruoko-BAC-sekvensseistä (Wang et ai., 2010). Durran ja Saccharumin välillä oli ilmeisesti deleetioita / insertioita, ja durra-genomin käyttö referenssinä ei voi korvata Saccharum-genomin ainutlaatuisia piirteitä ja voi siten aiheuttaa ennalta arvaamattomia FISH-tuloksia.
Joillakin FISH-dioilla oli vahvat taustakohina, joka voi johtua BAC-kloonin toistuvien sekvenssien (kuten SSR-sekvenssien) pienistä alueista, jotka tästä syystä tuottavat häiriöitä hybridisaation aikana. Durran genomin toistopitoisuus on noin 61% (Paterson et al., 2009), kun taas noin puolet genomista koostuu Saccarrum-hybridien toistosekvensseistä (De et ai., 2014). Näissä toistuvasti rikkaissa genomeissa on vaikea kehittää signaalispesifisiä FISH-koettimia, jotka erottavat kromosomit sytogeneettisesti samanlaisilla morfologioilla. CISS estävällä DNA: lla C ° t-100 voi tehokkaasti estää toistuvat sekvenssit. Tässä tutkimuksessa estävää DNA: ta C ° t-100 käytettiin BAC-koettimien FISH-analyysiin ryhmissä II ja III (kuvat 2D – F), joiden todettiin olevan kromosomispesifisiä BAC-koettimia, kun taas BAC-koettimien FISH ryhmissä IV ja V aiheutti voimakasta taustahäiriötä (kuvat 2D – F). On selvää, että toistettujen sekvenssien variaatioita on tutkittujen BAC-koettimien joukossa. Siksi kokeiden optimointi olisi tarpeen Saccharumin BAC-FISH-analyysiä varten. Ryhmässä II näytettiin kuusi – seitsemän kohtaa, mikä voi johtua homologisten kromosomirakenteen vaihteluista, kuten fragmentin deleetio yhdessä tai kahdessa homologisessa kromosomissa. Heksaploidisessa vehnässä BAC 676D4 hybridisoitui voimakkaammin A-genomikromosomeihin kuin B- ja D-genomikromosomeihin (Zhang et ai., 2004). Nämä yhden tai kahden signaalin havaitsematta homologisissa kromosomeissa voivat johtua myös BAC-FISH: n teknisistä ongelmista niin monien pienikokoisten kromosomien kohdalla.
Tässä tutkimuksessa tunnistimme 27 kromosomille spesifistä BAC- FISH-koettimet, jotka vastaavat 9: tä kymmenestä durra-kromosomista (kaikki paitsi Sb05). Tutkimuksessa S. officinarumin ja sokeriruoko-lajikkeen risteytyksestä johdetusta geneettisestä kartasta Sb05 sulatettiin sokeriruokoon Sb06: een (Aitken et ai., 2014). Tutkimuksessamme S. spontaneumin F1-populaatioon perustuva geneettinen kartta paljasti, että Sb05 jaettiin kahteen segmenttiin ja nämä kaksi segmenttiä yhdistettiin vastaavasti Sb06: een ja Sb07: een (Zhang ja Ming, julkaisemattomat tiedot). Äskettäin Andropogoneae-ryhmän jäsenen, Miscanthus sinensis, geneettinen kartta osoitti, että Sb05: llä on huono kollineaarisuus vastaavan sidosryhmän kanssa M. sinensisissä (Ma et ai., 2012). Samoin Sb05: lle ortologisilla kahdella esi-maissikromosomilla on pienin määrä synteettisiä ortologeja durra-geeneihin (Schubert ja Lysak, 2011). Siksi Sb05: tä vastaavan BAC-FISH: n puuttuminen voi viitata kromosomifuusioon S. spontaneumissa.
Sorra ja S. spontaneum erosivat 12 miljoonaa vuotta sitten (MYA), kromosomin perusmäärä väheni x: stä = 10 – x = 8. Julkaisussa Aitken et ai. (2014), sokeriruo’on HG2 (homologinen ryhmä 2) linjassa Sb05: n ja Sb06: n kanssa ja HG8 (homologinen ryhmä 8) Sb02: n ja Sb08: n kanssa, mikä antaa todisteita kromosomin pelkistystapahtumasta (Aitken et ai., 2014). Tässä tutkimuksessa havaitsimme, että durra-kromosomeilla Sb02 ja Sb08 oli kromosomien välinen uudelleenjärjestely S. S. spontaneumissa, kuten osoittavat todisteet siitä, että BAC-14 oli linjassa Sb02: n kanssa ja BAC-19 linjassa Sb08: n kanssa (kuva 7). Tutkimuksestamme saatiin ensimmäiset fysikaaliset ja sytogeneettiset todisteet durran kromosomien välisestä uudelleenjärjestelystä S. spontaneumissa. Ryhmämme julkaisematon data paljasti myös, että Sb08 on jaettu kahteen segmenttiin ja yhdistettiin Sb02: n ja Sb09: n kanssa S. spontaneumissa, vastaavasti (Zhang ja Ming, julkaisemattomat tiedot).Sb09: n, Sb08: n, Sb05: n ja Sb06: n vastaavia koettimia voitaisiin edelleen käyttää durran ja S. spontaneumin kromosomien välisten tapahtumien tutkimiseen. S. spontaneumilla on laaja valikoima ploidiatasoja (2n = 40–128) (Irvine, 1999). Näillä BAC-koettimilla voitiin vahvistaa S. spontaneumin kromosomien välinen uudelleenjärjestely eri polyploidiatasoilla.
Durran (x = 10) ja S. spontaneumin (x = 8), durra-kromosomit eivät olleet henkilökohtaisia vastauksia S. spontaneumin kanssa. Kuten aiemmin mainittiin geenikartoitustutkimuksessamme, Sb08 jaettiin kahteen segmenttiin, jotka sulautuivat S. spontaneumin Sb02-segmentteihin ja Sb09-segmentteihin; Sb05 jaettiin kahteen segmenttiin, jotka sulautuivat Sb06: n ja Sb07 S. spontaneumin segmentteihin. Siksi Sb08: ta ja Sb05: ää vastaavat koettimet eivät olleet spesifisiä yksittäisille S. spontaneum -kromosomeille, kun taas muita kahta muuta durra-kromosomia vastaavia koettimia voidaan käyttää S. spontaneumin kromosomispesifisinä sytogeneettisinä BAC-FISH-koettimina. Täten testatut BAC-koettimet perustuivat kahdeksaan durra-kromosomiin (taulukko 2) olivat riittävät kromosomien tunnistamiseen BAC-FISH: n avulla. Satelliittikromosomi löydettiin yhdestä S. spontaneum -kromosomista (kromosomi 3) (Ha et ai., 1999). Aikaisemmin samanaikainen FISH paljasti, että 45S rDNA: n signaalit sijaitsivat kromosomissa 3 olevien satelliittikromosomien toissijaisissa supistuksissa porkkana-viljelmästä peräisin olevasta S. spontaneum -kloonista (AP85-361) (Ha et ai., 1999). Tässä tutkimuksessa BAC-2, joka vastaa Sb03- ja 45s-rDNA: ta, kartoitettiin samaan S. spontaneum -kromosomin sijaintiin, mikä tukee edelleen aiempia havaintoja (Ha et ai., 1999). Tuloksemme antoivat myös suoraa näyttöä siitä, että edellisessä tutkimuksessa nimetty S. spontaneumin kromosomi 3 on homologinen Sb03: n kanssa.
Päätelmä
Tässä tutkimuksessa kehitimme S: n kromosomispesifisiä BAC: ita. spontaanium askeleena kohti yksinkertaisen ja toistettavan menetelmän kehittämistä kromosomien tunnistamiseksi käyttäen BAC-FISH-sytogeneettisiä markkereita, mikä vahvistaa, että durra-genomiin perustuvat kromosomispesifiset BAC: t voidaan eristää fyysisen sokeriruokokartan rakentamiseksi. Toimitamme myös ensimmäiset sytogeneettiset todisteet kromosomien välisestä järjestelystä durra ja S. spontaneum. Sokeriruoko BAC-FISH -teknologiajärjestelmän perustaminen tarjoaa uusia mahdollisuuksia ja keinoja sokeriruo’on molekyylisytogeneettiseen tutkimukseen, mukaan lukien kariotyyppianalyysi, geenien lokalisointi ja fyysisen kartan rakentaminen. Nämä tulokset ovat välttämättömiä koko genomin sekvensointiin tarvittavan S. spontaneum -genomin kokoamiseksi.
Kirjoittajan panokset
GD, JS ja JZ suunnittelivat tutkimuksen ja suunnittelivat kokeet. GD, JS, QZ, JW, QY, RM, KW ja JZ suorittivat kokeet ja analysoivat tulokset. GD ja JZ kirjoittivat käsikirjoituksen. Kaikki kirjoittajat lukivat lopullisen paperin ja hyväksyivät sen.
Rahoitus
Tätä projektia tuettiin apurahoilla 863-ohjelmasta (2013AA102604), NSFC: stä (31201260), uuden vuosisadan erinomaisten kykyjen ohjelmasta Fujianin maakunnan yliopisto ja Fujianin maa- ja metsätalousyliopiston rahoitus.
Lausunto eturistiriidoista
Kirjoittajat ilmoittavat, että tutkimus tehtiin ilman kaupallisia tai taloudellisia suhteita se voidaan tulkita mahdolliseksi eturistiriidaksi.
Kiitokset
Kiitämme tohtori Xintan Zhangia BAC-sekvenssien kokoamisesta ja tohtori Irene Lavagia englannin muokkaamisesta.
Lisämateriaali
Dhont, A. (2005). Polyploidien genomirakenteen purkaminen FISH: n ja GISH: n avulla; esimerkkejä sokeriruo’osta ja banaanista. Cytogenet. Genome Res. 109, 27–33. doi: 10.1159 / 000082378
PubMed Abstract | CrossRef-kokoteksti | Google Scholar
Piperidis, G., D’Hont, A. ja Hogarth, D. M. (2001). ”Erilaisten Saccharum-interspecifisten hybridien kromosomikoostumusanalyysi genomisen in situ (hybridisaation) avulla (GISH)”, julkaisussa Proceedings of the International Society of Sugar Cane Technologists, XXIV Congress (Brisbane, QLD: ASSCT), 565–566.
Google Scholar
